Modellierung und Simulation bieten neue Erkenntnisse für SARS-CoV-2
In unserem vorherigen Blog haben wir die Verwendung prädiktiver Modellierungswerkzeuge diskutiert, um erste Strukturen potenzieller Wirkstoffziele (z. B. Proteine) auf atomarer Ebene aufzubauen und Regionen zu verfeinern, die experimentell nicht bestimmbar waren (Siehe Video ). Zu diesen Werkzeugen gehören das Hinzufügen von Wasserstoffatomen und flexiblen Schleifen, die manchmal experimentell nicht aufgelöst werden können. Wir haben dies im Rahmen der Kryo-Elektronenmikroskopie (Kryo-EM) des SARS-CoV-2 Spike (S)-Proteins untersucht, die kürzlich in der Zeitschrift Science veröffentlicht wurde (DOI:10.1126/science.abb2507).
In diesem Blog werden wir detailliert beschreiben, wie molekulare Modellierung und Simulation von verfeinerten Strukturmodellen wie dem SARS-CoV-2 S-Protein bei der Erstellung neuer Hypothesen für die Entdeckung und das Design mutmaßlicher Therapeutika zur Behandlung von COVID-19 helfen können.
Arzneimittelbindung hängt von strukturellen Veränderungen ab
In lebenden Systemen existieren Proteine von Natur aus als dynamische Einheiten. Ihre Dynamik bestimmt oft ihre Funktion. Der Physiker Richard Feynman sagte einmal:
„Wenn wir die mächtigste aller Annahmen nennen sollten, die einen immer weiter dazu führt, das Leben zu verstehen, dann ist es, dass alle Dinge aus Atomen bestehen und dass alles, was Lebewesen tun, in Begriffen verstanden werden kann des Wackelns und Wackelns der Atome.“ 1
Das SARS-CoV-2 S-Protein ist keine Ausnahme von Feynmans Prinzip. Bevor es in menschliche Zellen eindringt, bindet das S-Protein einen Rezeptor, der als Angiotensin-Converting-Enzym 2 (ACE2) bezeichnet wird. 2 Die Rezeptorbindungsdomäne (RBD) ist Teil des S-Proteins, das ACE2 bindet (Abbildung 1A ). Die RBD kann in mindestens zwei primären Konformationszuständen existieren, die als Aufwärts- (Rezeptor-zugänglich) und Abwärts- (Rezeptor-unzugänglich) bezeichnet werden (Abbildung 1B .). ). Wenn sich die RBD im Up-Zustand befindet, ist das S-Protein „offener“, um die Bindung von ACE2 zu erleichtern. 2 Studien haben gezeigt, dass der Daunenzustand, der für den Rezeptor unzugänglich ist, stabiler ist. 2 Dies impliziert, dass mutmaßliche Therapeutika wie kleine organische Moleküle, die in der Lage sind, RBD zu binden, die RBD im Down-Zustand stabilisieren und verhindern könnten, dass das Virus mit ACE2 interagiert; Dadurch wird verhindert, dass COVID-19 Menschen infiziert.
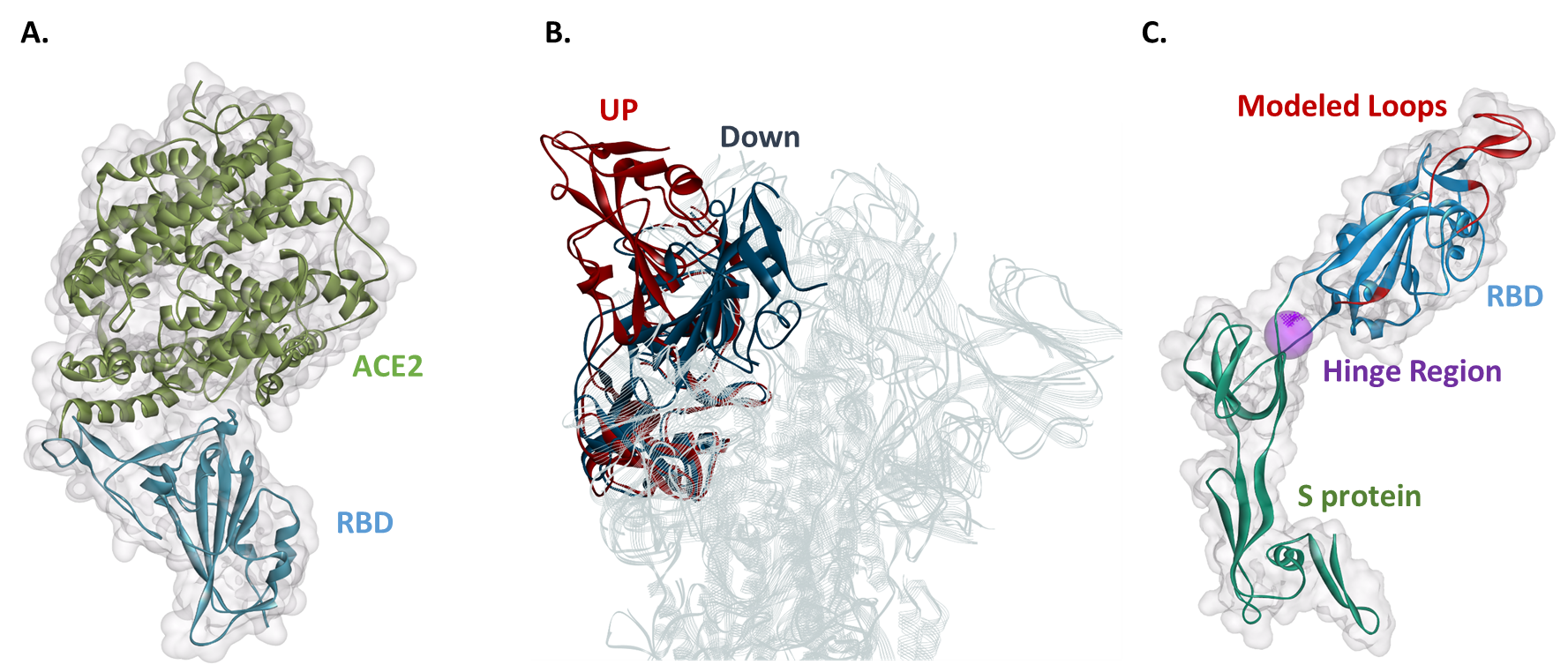
Die RBD des S-Proteins ist wie ein Scharnier an einer Tür
Ein flexibler Linker verbindet die RBD mit dem restlichen S-Protein. Die Flexibilität des Linkers ermöglicht es der RBD, durch eine Scharnier-Biege-Bewegung vom unteren in den oberen Zustand überzugehen (Abbildung 1B ). Aus dem SARS-CoV-2 S-Protein haben wir die RBD mit dem zugehörigen flexiblen Linker und der angrenzenden Domäne extrahiert (Abbildung 1C ). Wir haben die RBD-Struktur des S-Proteins im Up-Zustand (PDB 6VYB) verwendet, aber dieser Struktur fehlen drei Schleifen, die möglicherweise für die Bindung von ACE2 wichtig sind (Abbildung 1C .). ). Als Ergebnis mussten wir ein Homologiemodell der RBD mit dem flexiblen Linker aufbauen, wobei die Kryo-EM-Struktur verwendet wurde, aus der die Verkürzung ursprünglich hergestellt wurde (PDB 6VYB) mit einem zusätzlichen Templat. Das zusätzliche Templat war die Kristallstruktur des RBD allein im Komplex mit ACE2 (PDB 6M17) (Abbildung 1A ).
Die RBD-Struktur enthält Schleifen, die einen mit mehr als 20 Aminosäureresten umfassen, die in der Struktur des offenen Zustands (PDB 6VYB) nicht vorhanden sind. Zwei dieser Schleifen bilden die Interaktion mit ACE2; daher ist eine Homologiemodellierung notwendig, um die Wechselwirkungen des Spike-Proteins zu verstehen. Wir können dann Wasserstoffatome in geeigneten Protonierungszuständen zuordnen, um physiologische Bedingungen wie den pH-Wert nachzuahmen.
Wir könnten dann eine Molekulardynamik-Simulation (MD) durchführen, um den Konformationsübergang zu simulieren und/oder mögliche Bindungsstellen vorherzusagen, an denen mutmaßliche kleine Moleküle binden könnten, um die Interaktion des S-Proteins mit ACE2 zu stören. Als wir mit BIOVIA Discovery Studio mögliche Bindungsstellen für unser Homologiemodell der RBD mit dem flexiblen Linker vorhersagten, identifizierten wir eine Bindungsstelle am Gelenk des flexiblen Linkers (Abbildung 1C .). ). Wir haben diese Region als Scharnier bezeichnet und stellen fest, dass sie einer weiteren Untersuchung für die Entdeckung mutmaßlicher Therapeutika würdig sein könnte. Wenn ein kleines Molekül an der Hinge-Region binden würde, könnte es die RBD möglicherweise in einem Down-Zustand verriegeln und so die ACE2-Bindung verhindern.
Weitere Untersuchungen
Umfangreiche Untersuchungen einschließlich computergestützter Vorhersagen und biologischer Experimente könnten den Nutzen der Scharnierregion weiter aufklären. Beispiele für rechnerische Vorhersagen können die Normalmodusanalyse (NMA) und/oder die MD-Simulation sein. 3 Beispielsweise könnte eine lange Zeitskala von etwa Hunderten von Nanosekunden der MD-Simulation es Wissenschaftlern ermöglichen, mehrere Konformationen der Scharnierregion zu untersuchen.
Andererseits könnte eine NMA eine grobe und schnelle Schätzung der Konformationsübergänge liefern. 3 Spezifische Konformationen aus der MD-Simulation und/oder NMA sind Ausgangspunkte für das virtuelle Hochdurchsatz-Screening potenzieller niedermolekularer Datenbanken. Wissenschaftler könnten jedes kleine Molekül an alle Konformationen andocken und bewerten. Sie konnten dann alle resultierenden Posen einordnen und die besten Treffer zur experimentellen Validierung einreichen. Studien haben gezeigt, dass diese Methode der computergestützten Wirkstoffforschung, die oft als ensemblebasiertes virtuelles Screening bezeichnet wird, die Chancen verbessert, Wirkstoffkandidaten zu identifizieren. 4 Die Methode spiegelt auch die Realität wider, dass die Wirkstoffbindung von strukturellen Veränderungen im Protein abhängt, wie oben erwähnt. Wir glauben, dass die hier identifizierten vorläufigen Ergebnisse interessant sind und weitere Untersuchungen wert sind.
Zweitens möchten wir darauf hinweisen, dass das verfeinerte S-Protein-Strukturmodell als Ziel für Immuntherapeutika verwendet werden könnte. 5 Wissenschaftler könnten basierend auf Vorkenntnissen über die ACE2-Bindungsstelle monoklonale Antikörper entwickeln, die an die SARS-CoV-2 S-Proteine binden. Sie könnten dann in silico auftreten Affinitätsreifungsstudien zur Verbesserung der Bindungsspezifität. 6
Als aktiver Unterstützer der wissenschaftlichen Gemeinschaft, die heute an COVID-19-Lösungen mitarbeitet, entwickelt BIOVIA Dassault Systèmes BIOVIA Discovery Studio. Diese bewährte Modellierungs- und Simulationsumgebung für Biowissenschaften vereint über 30 Jahre begutachtete Forschung und erstklassige in silico Techniken. Die Software bietet Wissenschaftlern ein komplettes Toolset für den Einsatz von der Targetidentifizierung bis zur Leitstrukturoptimierung, einschließlich Tools für das Design und die Analyse von Biologika, klassische Simulationen, struktur- und fragmentbasiertes Design, virtuelles Ligandenscreening sowie ADME- und Toxizitätsvorhersage.
Als Teil der sozialen Verantwortung von Dassault Systèmes freut sich BIOVIA, qualifizierenden akademischen Forschungsgruppen, die an Studien im Zusammenhang mit SARS-CoV-2 beteiligt sind, eine kostenlose sechsmonatige Lizenz für das BIOVIA Discovery Studio anzubieten um sie bei der Suche nach schnellen, sicheren und wirksamen therapeutischen Medikamentenkandidaten gegen das SARS-CoV-2-Virus zu unterstützen. Wenn Sie wissenschaftlicher Mitarbeiter auf diesem Gebiet sind, fordern Sie bitte eine Softwarelizenz an und laden Sie sie herunter. Dieses Angebot gilt bis zum 30. Juni 2020.
Biologie
- Pixus:neue dicke und robuste Frontplatten für eingebettete Boards
- GE stellt neues Produkt für Steuerungs- und Überwachungsanwendungen vor
- DSM und Nedcam entwickeln neue Anwendungen für den großformatigen 3D-Druck
- Teradyne plant neuen Cobot-Hub für die Portfoliounternehmen UR und MiR
- PLASTICS veröffentlicht neuen Sicherheitsstandard für Robotik und Spritzguss
- Ein neuer Fahrplan für Öl- und Gaslieferketten
- Coaching für Nachhaltigkeit:Neue Prozesse und Veränderungen implementieren und aufrechterhalten
- B&R stellt neues Simulationstool für die Entwicklung digitaler Zwillinge vor
- ABB liefert Automatisierungs- und Elektrifizierungsplanung für neue Mine in Schweden
- 5G und Edge stellen neue Herausforderungen für die Cybersicherheit im Jahr 2021 dar



