Kontrolle der DNA-Translokation durch Festkörper-Nanoporen
Zusammenfassung
Verglichen mit dem Status von Bio-Nanoporen sind noch einige Herausforderungen zu bewältigen, bevor Festkörper-Nanoporen in der kommerziellen DNA-Sequenzierung eingesetzt werden können. Geringe räumliche und geringe zeitliche Auflösung sind die beiden größten Herausforderungen. Aufgrund von Beschränkungen der Nanoporenlänge und der Oberflächeneigenschaften der Festkörper-Nanoporen besteht noch Raum für eine Verbesserung der räumlichen Auflösung. Unterdessen ist die DNA-Translokation unter einer elektrischen Kraft zu schnell, was dazu führt, dass nur wenige gültige Datenpunkte erfasst werden. Die zeitliche Auflösung von Festkörper-Nanoporen könnte daher verbessert werden, wenn die DNA-Translokationsgeschwindigkeit gut kontrolliert wird. In diesem Mini-Review fassen wir kurz die Methoden zur Verbesserung der räumlichen Auflösung zusammen und konzentrieren uns auf kontrollierbare Methoden, um die Auflösung der Nanoporen-Detektion zu fördern. Darüber hinaus bieten wir einen Ausblick auf die Entwicklung der DNA-Sequenzierung durch Nanoporen.
Einführung
In den letzten Jahrzehnten wurden große Fortschritte bei der Anwendung der DNA-Sequenzierung zum Ablesen der Basensequenzen im Genom gemacht [1, 2]. Um personalisierte Medikamente zu entwickeln, haben Forscher nach einer schnelleren und kostengünstigeren DNA-Sequenzierungsmethode gesucht, bei der Zielmedikamente und medizinische Behandlungen spezifisch auf eine Person angewendet werden können [3, 4]. Da die Nanoporentechnologie beim DNA-Nachweis verwendet wurde [5], wurde sie als effektive Methode für die Sequenzierung der nächsten Generation angesehen [6, 7]. Die Nanoporen-Technologie ist eine vielversprechende Plattform, um hochempfindliche Einzelmolekül-Detektoren für DNA [5] oder RNA [8] zu identifizieren. Beim grundlegenden Detektionsschema wird eine elektrochemische Kammer durch eine dünne Membran mit einer Nanopore [9], die die leitfähige Lösung und den Analyten in der elektrochemischen Kammer verbindet, in zwei Reservoirs (cis- und trans-Kompartimente) getrennt. Durch Anlegen einer Spannung an die Membran fließen Elektrolytionen durch die Festkörper-Nanopore und bilden einen Porenstrom, der mit einem Patch-Clamp-Aufbau mit zugehöriger hochempfindlicher Elektronik gemessen wird. Wenn ein Molekül oder ein Molekülkomplex die Nanopore passiert, kann der Analyt einige Ionen aus dem durch die Nanopore definierten Volumen ausschließen, was durch Überwachung kurzer Stromänderungen nachgewiesen werden kann. Sowohl aus der Verweilzeit (Verweilzeit) innerhalb der Nanopore als auch aus der Stromamplitudensignatur können Informationen über die Moleküle gewonnen werden. Die räumliche Auflösung der Nanoporensequenzierung wird durch die Abmessungen der Nanopore bestimmt, was darauf hindeutet, dass sie als einzelner Sensor für kleine molekulare Objekte verwendet werden kann, was zu einer nachweisbaren Stromsignatur führt. Darüber hinaus lassen sich Nanoporen-Sensoren leicht in hochportable Lab-on-a-Chip-Geräte integrieren und miniaturisieren [10].
Signifikante Fortschritte wurden bei der DNA-Sequenzierung durch Nanoporen, wie Festkörper-Nanoporen [2, 11] und Protein-Nanoporen [2, 7], erzielt. DNA-Sequenzierung durch Protein-Nanoporen wurde erreicht [7]. Protein-Nanoporensysteme haben jedoch Grenzen für die Untersuchung biologischer Moleküle. Es gibt weniger Einschränkungen bei Festkörper-Nanoporen im Vergleich zu biologischen/Protein-Nanoporen. Diese Schwierigkeiten können durch Festkörper-Nanoporen überwunden werden. Im Vergleich zu Protein-Nanoporen sind sie über einen breiteren Temperatur-, Spannungs- und Lösungsmittelbereich funktionsfähig und können im Durchmesser mit Sub-Nanometer-Präzision eingestellt werden. Sie sind vielversprechend für die Anwendung in der Technologie der nächsten Generation zur DNA-Sequenzierung.
Für DNA-Sensoren wurden viele Festkörper-Nanoporen unterschiedlicher Materialien und Strukturen hergestellt. Die DNA-Sequenzierung wird jedoch nicht durch Festkörper-Nanoporen erreicht. Für Festkörper-Nanoporensensoren müssen vor ihrer kommerziellen Anwendung bei der DNA-Sequenzierung zwei Haupthindernisse hinsichtlich ihrer räumlichen und zeitlichen Auflösung überwunden werden. Die Schwierigkeit bezüglich der räumlichen Auflösung besteht darin, dass die Festkörper-Nanopore den winzigen Abstand zwischen zwei benachbarten Nukleotiden unterscheiden kann, um eine Einzelbasenauflösung zu erreichen. Das Hindernis der zeitlichen Auflösung besteht darin, dass die DNA-Translokation unter einer elektrischen Kraft zu schnell ist, was dazu führt, dass nur sehr wenige gültige Datenpunkte durch vorhandene Patch-Clamps oder andere Signalerfassungssysteme erfasst werden. Dieser Mini-Review bietet einen Überblick über die verschiedenen Methoden zur Verbesserung der räumlichen und zeitlichen Auflösung der Festkörper-Nanoporen-DNA-Detektion. Dieser Mini-Review konzentriert sich auch mehr auf Methoden zur Verlangsamung der DNA-Translokation durch Festkörper-Nanoporen.
Räumliche Auflösung
2001 berichteten Li et al. erstmals über Festkörper-Nanoporen aus Siliziumnitrid. [12]. Für die molekulare DNA-Detektion wurden verschiedene Festkörper-Nanoporen nachgewiesen, wie z. B. Siliziumoxid [13], Silizium [14], Al2 O3 [15] und HfO2 [16]. Obwohl Festkörper-Nanoporen letztendlich robust gegenüber chemischen und mechanischen Bedingungen sein können, weisen sie einige Einschränkungen auf, wie z. B. eine geringe räumliche Auflösung. Aufgrund der Dicke der Materialien können Dutzende von Basen gleichzeitig Festkörper-Nanoporen passieren. Derzeit ist die dünnste Siliziumnitrid-Nanopore 3 nm groß, was immer noch nicht die vier Arten von Basen unterscheidet [17].
Interessanterweise beträgt die Dicke einer einzelnen Schicht aus zweidimensionalem (2D) Material etwa 3.0–11.0 Å, was mit dem Abstand zwischen zwei benachbarten Nukleotiden entlang der ssDNA (3.2–5.2 Å) vergleichbar ist [18]. Zweidimensionale Membranen wie Graphen (3,4 Å [19]), MoS2 (6.5 Å [18]), WS2 (7 Å [20]) und h-BN (11 Å [21]) konnten aufgrund ihres hohen Signal-Rausch-Verhältnisses und ihrer räumlichen Auflösung DNA-Translokationen nachweisen [21,22,23]. Aus ihrer räumlichen Auflösung ist klar, dass diese Materialien für den DNA-Nachweis verwendet werden können. Außerdem ermöglichen reproduzierbare Wachstumstechniken und großtechnische Transferverfahren die Herstellung von Sub-Nanometer-Poren in 2D-Membranen im großen Maßstab.
Graphen ist eine atomar dünne Schicht von Kohlenstoffatomen, die zu einem zweidimensionalen Wabengitter angeordnet ist [24]. Forscher haben gezeigt, dass einzelne DNA-Moleküle in Lösung nachgewiesen und mit Graphen-Nanoporen charakterisiert werden können [22, 25]. Es gibt jedoch starke hydrophobe Wechselwirkungen zwischen DNA-Nukleotiden und Graphen, und die DNA wird stark verstopfen und an Graphen-Nanoporen kleben, was die Translokationsgeschwindigkeit deutlich beeinträchtigen würde [26]. Mit den hydrophilen Gruppen modifiziert [26] oder beschichtet, könnte ein hydrophiles Material [25] auf Graphen die Hydrophilie von Graphen verbessern und ein Anhaften von DNA an seiner Oberfläche vermeiden. Leider erhöht entweder die Modifikation mit hydrophilen Gruppen oder die Beschichtung mit hydrophilen Materialien die Dicke des suspendierten Films, was zu einer Zunahme der Dicke der Nanoporen führt und somit die räumliche Auflösung von Graphen verringert.
Das geschichtete Übergangsmetalldichalkogenid ist ein weiteres 2D-Material, das MoS2 . enthält [18, 27] und WS2 [20]. Ein hohes Signal-Rausch-Verhältnis (SNR> 10) und eine fünffache Verstärkung des Ionenstromsignals wurden bei der DNA-Translokation durch das MoS2 . festgestellt Nanoporenmembran [18]. Inzwischen MoS2 ist hydrophil, sodass keine spezielle Oberflächenbehandlung erforderlich ist, um eine hydrophobe Wechselwirkung zwischen DNA und ihrer Oberfläche zu vermeiden. Das andere Material, WS2 hat eine direkte Bandlücke von 2,1 eV [28] und seine Photolumineszenz(PL)-Emission ist stärker als die des gut untersuchten MoS2 [29]. Dandaet al. [20] fabrizierte eine WS2 nanopore und demonstrierten die Erzielung einer atomar kontrollierten Nanoporengröße mit kurzen Lichtpulsen, die positive Auswirkungen auf Festkörper-Nanoporen für den DNA-Nachweis haben könnte.
Außerdem haben Liu et al. [21] berichteten über das erste Experiment zur DNA-Translokation durch h-BN-Nanoporen. Ähnlich wie Graphen hat h-BN eine schlechte Hydrophilie, was dazu führt, dass die DNA Nanoporen blockiert. Anschließend haben Zhou et al. [23] verbesserten erfolgreich die Hydrophilie von h-BN-Nanoporen, indem sie die Antioxidation und Integrität von h-BN-Material nach UV-Ozon-Behandlung nutzten. Die isolierende Natur von h-BN kann in einer Lösung mit hoher Ionenstärke bemerkenswertere Haltbarkeit und isolierende Eigenschaften aufweisen als in Graphen. Es ist ein wettbewerbsfähiger Kandidat, um eine Einzelbasenauflösung auf superdünnen Nanoporenstrukturen zu erreichen.
Die Verwendung von zweidimensionalen Materialien kann möglicherweise die räumliche Auflösung der Vorrichtung erhöhen, um eine Einzelnukleotidauflösung zu erreichen. Obwohl über DNA-Nachweisexperimente für mehrere zweidimensionale Materialien berichtet wurde, hat niemand über die Erzielung der Festkörper-Nanoporen-DNA-Sequenzierung berichtet. Auch die zeitliche Auflösung der Nanoporen-Sequenzierung ist eine Herausforderung.
Zeitliche Auflösung
Die DNA-Translokationsgeschwindigkeit durch eine Festkörper-Nanopore ist mit bis zu 0,01–1 µs/Base sehr schnell [30], was zu sehr wenigen effektiven Daten führt, die von kommerziellen Patch-Clamps gesammelt werden. Daher ist es nicht möglich, jede Basis anhand des Blockadestromsignals zu unterscheiden. Die DNA-Translokationsgeschwindigkeiten von Festkörper-Nanoporen von 2D-Materialien wie Graphen, MoS2 , WS2 , und h-BN, sind in Abb. 1 dargestellt. Idealerweise sollte die DNA-Translokationsgeschwindigkeit in einer Nanopore 1–100 bp/ms betragen, um eine zufriedenstellende Aufzeichnung des Signals von jedem Nukleotid zu ermöglichen [32].
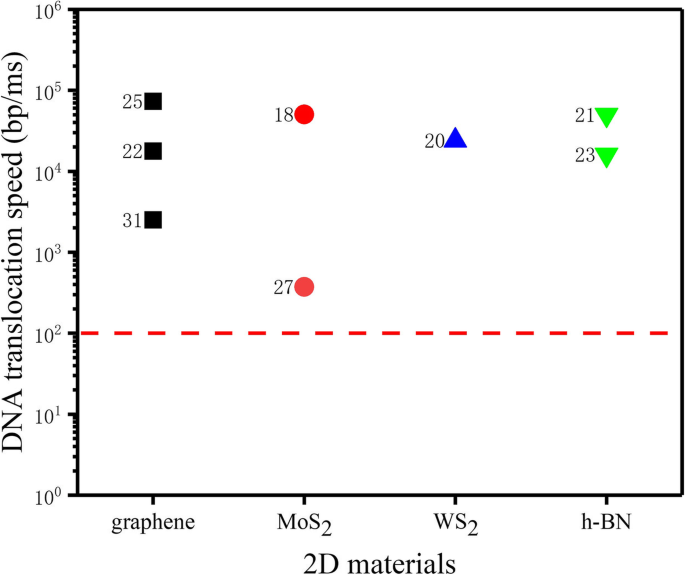
DNA-Translokationsgeschwindigkeit von 2D-Festkörper-Nanoporen [18, 20, 21, 22, 23, 25, 27, 31]. Die gepunktete rote Linie zeigt eine DNA-Translokationsrate von 100 bp/ms an
Vor diesem Hintergrund ist die Verlangsamung der Translokationsgeschwindigkeit der DNA ein wichtiges Ziel vieler Forscher. Es wurden verschiedene Methoden entwickelt, um die DNA-Translokation zu verlangsamen, um die zeitliche Auflösung der Festkörper-Nanoporen-Detektion zu verbessern. Die übliche Methode besteht darin, den Einfluss experimenteller Faktoren wie Temperatur [33], Elektrolytviskosität [27], Antriebsspannung [34], Ionenkonzentration [35] und Oberflächenladungsdichte einer Nanopore [36] durch Veränderung der DNA zu verändern Translokation hindurch. Wanunuet al. [33] konzentrierte sich auf die Verlangsamung der dsDNA-Translokation durch Festkörper-Nanoporen durch Änderung von Temperatur, Spannung und DNA-Länge. Darüber hinaus haben Feng et al. [27] zeigten, dass ein Viskositätsgradientensystem basierend auf ionischen Flüssigkeiten bei Raumtemperatur verwendet werden kann, um die Dynamik der DNA-Translokation durch MoS2 . zu steuern Nanoporen und zeigte, dass die hohe Viskosität ionischer Flüssigkeiten bei Raumtemperatur optimale Einzelnukleotid-Translokationsgeschwindigkeiten von 373 bp/ms bietet.
Obwohl viele Ansätze das Potenzial haben, die DNA-Translokationsgeschwindigkeit zu verringern und den Ionenstromnachweis zu erleichtern, können sie die Anforderungen für die DNA-Sequenzierung immer noch nicht erfüllen. Daher ist es notwendig, radikalere Methoden zu entwickeln, um den Durchgang von DNA durch Nanoporen zu kontrollieren. Hier diskutieren wir die Methoden zur Kontrolle der Nanoporenstruktur und der quantitativen DNA-Bewegung, um die zeitliche Auflösung zu verbessern.
Zwei-Nanoporen-System
Forscher haben Zwei-Nanoporen-Systeme verwendet, um die Translokation von DNA-Molekülen zu manipulieren, die das gleiche Molekül viele Male kontrollierbar nachweisen können. Zwei gestapelte Nanoporen, getrennt durch ein mikrometergroßes Hohlraumkompartiment [37] (Abb. 2a), können DNA-Moleküle für eine gewisse Zeit einfangen und kontrollierbar freisetzen. Die Dynamik von DNA-Molekülen kann durch Korrelationsanalyse aus den Signalen der beiden Poren abgeleitet werden, was einen direkten elektrischen Beweis für die Translokation liefert. Darüber hinaus kann aufgrund der Entropiebarrieren des zweischichtigen Nanoporensystems die Brownsche Bewegung von DNA-Molekülen eingeschränkt werden, was die DNA-Sequenzierungsgenauigkeit mit Nanoporen verbessern kann. Im Vergleich zu Einzelmolekül-Sensortechniken können DNA-Moleküle in Zweischicht-Nanoporensystemen mehrfach gemessen werden, indem mehrere Poren seriell angeordnet werden, anstatt sie durch eine einzelne Pore hin und her zu leiten [39].
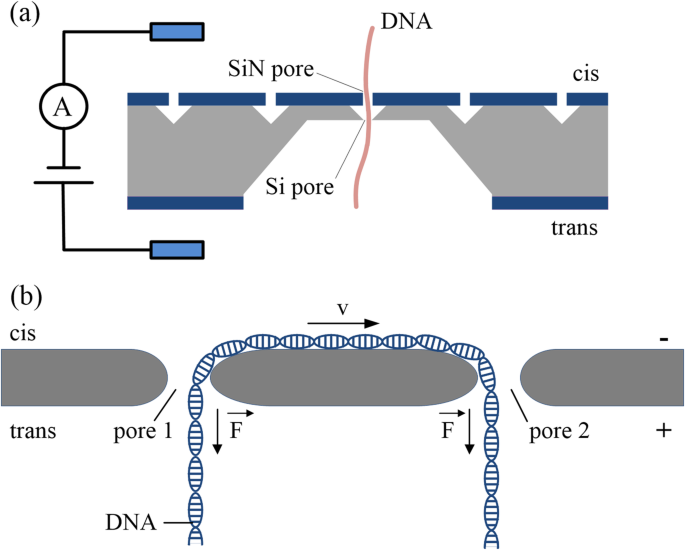
a Schema eines zweischichtigen Nanoporensystems, das für den Nanoporen-DNA-Nachweis verwendet wird [37]. b Schema eines Doppel-Nanoporen-Systems, das für den Nanoporen-DNA-Nachweis verwendet wird [38]
Doppel-Nanoporen-Systeme bieten eine weitere Methode, um den molekularen Transport zu kontrollieren und Moleküle zwischen zwei Poren effizient zu überbrücken; sie sind ein markierungsfreier mechanistischer Ansatz für die DNA-Manipulation [40]. In einem Doppelnanoporensystem gibt es zwei unabhängig adressierbare und benachbarte Nanoporen innerhalb derselben Festkörpermembran. Beim elektrophoretisch getriebenen Durchgang eines DNA-Moleküls durch eine der Nanoporen kann ein einzelnes DNA-Molekül in beide Poren eingefangen werden, was zu einem „Tauziehen“ zwischen den beiden Nanoporen führt [38] (Abb. 2b). Daher werden auf die verschiedenen Enden eines DNA-Moleküls Kräfte ausgeübt, die seine Bewegung verlangsamen und sogar vollständig zum Stillstand bringen. Das Doppel-Nanoporen-System eröffnet einen neuen Weg zum mechanischen Einfangen von DNA in Festkörper-Nanoporen und ist eine vielversprechende Methode zur Messung einer Vielzahl von Biomolekülen mit den Vorteilen der Markierungsfreiheit und der hohen Signalstärke. Rauschabstand und niedrige Kosten. Es kann die DNA-Moleküle effizient einschließen und einfangen, um die DNA-Translokation zu verlangsamen, und kann auch verwendet werden, um die Physik dieses nanoskaligen Tauziehens um DNA zu studieren [41].
Optische Falle Nanopore
Optische Fallen-Nanoporen ermöglichen es einer optischen Pinzette, Partikel mit einer Größe von weniger als zehn Nanometern einzufangen. Es ermöglicht das optische Einfangen von Proteinen [42], DNA-Fragmenten [43] und anderen Biomolekülen [44] sowie kleinen Viren [45]. Die grundlegende Theorie hinter optischen Fallen-Nanoporen ist eine selbstinduzierte optische Rückwirkungsfalle [46]. Der auf den Bereich des Nanoporen-Arrays fokussierte Laserstrahl wird ein lokales Lichtfeld hoher Leistungsdichte am Rand der Metallschicht im Loch bilden. Wenn sich ein Partikel zwischen dem lokalen Lichtfeld bewegt, kann dies eine große Änderung der lokalen optischen Transmission bewirken, was wiederum eine große optische und dielektrische Kraft auf das Partikel ausübt. Eine doppelte Nanolochstruktur wird verwendet, um den Größenengpass der eingefangenen Partikel zu durchbrechen. Mohammed et al. [47] demonstrierten die potenzielle Verwendung von optischen Fallen-Nanoporen mit 20-nm-Siliciumdioxid- und Au-Nanopartikeln. Die Hantelform des Doppelnanolochs wurde in den Au-Film gefräst und eine 25-nm-Nanopore wurde durch das suspendierte Six . gebohrt Ny Membran in der Mitte des Doppelnanolochs, wie in Abb. 3a gezeigt. Elektrophoretische Kraft, die Nanopartikel durch die Nanopore treibt, beim Passieren durch den Rand des Lochs, die selbstinduzierte plasmonische Rückwirkungskraft, die zwischen den Spitzen des Doppelnanolochs existiert, wirkt der elektrophoretischen Kraft entgegen, wodurch die Geschwindigkeit der Nanopartikel verringert wird. Die Ergebnisse zeigten, dass das optische Einfangen die elektrophoretische Translokationszeit um vier Größenordnungen verlängerte. Kimet al. [43] realisierten das optische Einfangen von Plasmid-DNA und Lambda-DNA unter Verwendung der nanoplasmonischen Strukturen einzelner Nanoporen. Die Technologie hat potenzielle Anwendungen für die DNA-Detektion und es können mehrere lokale Lichtfelder eingerichtet werden, um eine parallele Detektion zu realisieren. Hochfrequente Ionenstromoszillationen können jedoch die aktuellen Erkennungsergebnisse der DNA beeinträchtigen. Dies kann auf die Konkurrenz zwischen elektrophoretischen und selbstinduzierten Rückwirkungskräften zurückzuführen sein, die dazu führen, dass die Nanopartikel durch die Mündung der Nanopore auf und ab schweben.
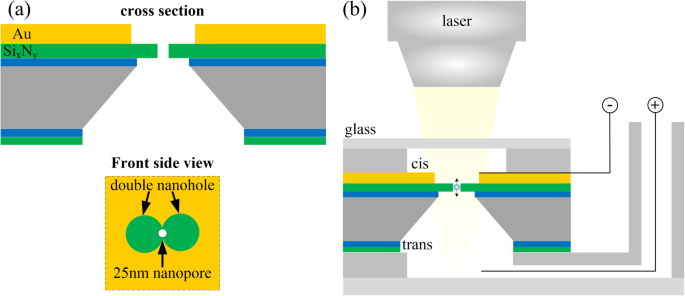
a Schema des Nanoporen-Chips der optischen Falle [47]. b Schema des selbstinduzierten Rückwirkungssystems
Optische Pinzette
Optische Pinzetten können verwendet werden, um die molekulare Translokation durch Nanoporen zu kontrollieren und wurden in den letzten Jahren häufig verwendet. 2006 haben Keyser et al. [48] demonstrierten erstmals ein molekulares Tauziehen zwischen elektrophoretischen und mechanischen Kräften durch Anwendung optischer Pinzetten zur Kontrolle der DNA-Translokation durch SiNx Nanoporen. Dieses System wirkt wie eine einfache Hooke-Feder, und die Zugkraft auf den Wulst kann basierend auf dem Hookeschen Gesetz berechnet werden:F nicht =−k Falle Z , wobei F nicht ist die optische Kraft, k Falle ist die Trap-Steifigkeit entlang der Verschiebungsrichtung und Z ist die lineare Verformung des Wulstes [48]. Die Methode der optischen Pinzette, die eine DNA-gebundene Polystyrol-Perle im Crossover eines fokussierten Laserstrahls einfängt, kann die DNA-gebundene Polystyrol-Perle in drei Dimensionen manipulieren und hat einen Piko-Newton-Bereich der Kraftempfindlichkeit, wie in Abb. 4a . gezeigt . Um die translozierende DNA innerhalb der Nanopore einzufangen, wurde die Zugkraft so abgestimmt, dass die elektrische Feldkraft (F el ) auf der DNA. Daher kann die Spannung verwendet werden, um sowohl die Geschwindigkeit der DNA-Translokation zu reduzieren als auch das DNA-Molekül aus einer Nanopore herauszuziehen [52]. Dieses System ermöglicht die simultane räumliche Abtastung und hochauflösende Kraftmessungen von Nukleinsäuren und Proteinen, was erhebliche Fortschritte in der DNA-Sequenzierung erzielt hat. Die Technik der optischen Pinzette leidet jedoch unter mehreren grundlegenden Schwierigkeiten. Erstens ist es schwierig, auf eine große Anzahl von Nanoporen zu skalieren. Zweitens wirkt sich die durch den Laser in einer optischen Pinzette verursachte Erwärmung stark auf den Ionenstrom durch eine Nanopore und den Rauschpegel aus, sodass die optisch gefangene Perle mehrere Mikrometer von der Nanopore entfernt sein muss [53].
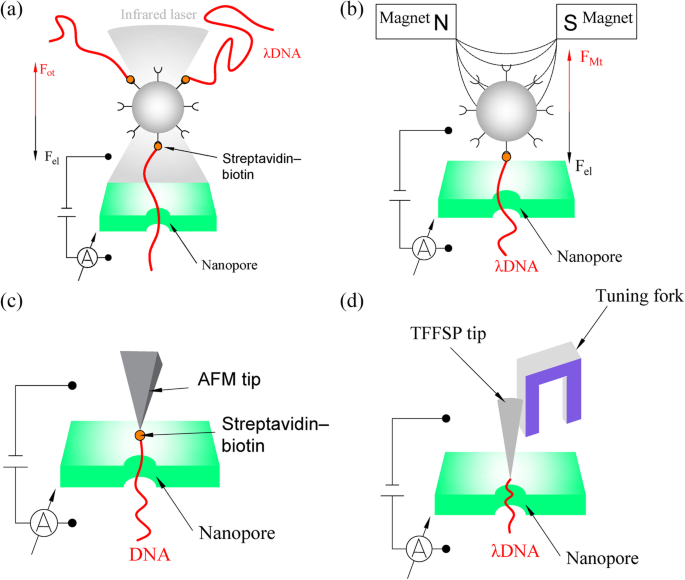
a Schema einer optischen Pinzette zum Nachweis von Nanoporen-DNA [48]. Wenn das optische Pinzettensystem im Gleichgewicht ist, wird die optische Kraft (Fot ) ist gleich der elektrischen Feldkraft (Fel ). b Schema einer magnetischen Pinzette zum Nachweis von Nanoporen-DNA [49]. Wenn das magnetische Pinzettensystem im Gleichgewicht ist, wird die magnetische Kraft (FMt ) ist gleich der elektrischen Feldkraft (Fel ). c Schema eines AFM, das zum Nachweis von Nanoporen-DNA verwendet wird [50]. d Schema des TFFS für den Nanoporen-DNA-Nachweis [51]
Magnetische Pinzette
Magnetpinzetten bieten eine weitere Möglichkeit, die DNA-Translokation durch Zugkraft zu kontrollieren, und es wurde gezeigt, dass die magnetische Pinzettentechnik die DNA-Translokation wirksam verlangsamt [49]. In diesem System können, wie in Abb. 4b gezeigt, DNA-Moleküle unter Verwendung einer starken Gold-Thiol-[54] oder Streptavidin-Biotin-[49]-Wechselwirkung an ein mikrometergroßes magnetisches Bead gebunden werden. Dann kann das freie Ende der DNA durch ein angelegtes elektrisches Feld in der Nanopore eingefangen werden. Anschließend können zwei Magnete mit kleinem Spalt verwendet werden, um einen Magnetfeldgradienten zu erzeugen. Diese Technik kann die elektrische Kraft auf die eingefangene DNA ausgleichen, um die Translokationsgeschwindigkeit zu reduzieren und sogar die Elektrophorese umzukehren. Im Vergleich zu optischen Pinzetten sind magnetische Pinzetten ein vielversprechender Kandidat für die massiv parallele Kraftspektroskopie. In diesem System können Hunderte von Kügelchen und damit DNA-Moleküle gleichzeitig innerhalb von Hunderten von Nanoporen kontrolliert werden, die leicht auf viele adressierbare Nanoporen skalierbar sind. Dies kann den Analyseprozess um Größenordnungen beschleunigen. Ein offensichtlicher Nachteil des magnetischen Pinzetten-Ansatzes im Vergleich zu optischen Pinzetten ist jedoch die fehlende dreidimensionale Kontrolle der Moleküle [55].
Force Sensing Probe
Obwohl erhebliche Fortschritte bei der Kontrolle der DNA-Translokationsgeschwindigkeit durch optische Pinzetten und magnetische Pinzetten erzielt wurden, haben die Bead-Trapping-Methoden ein Problem mit der Brownschen Bewegung, die es schwierig macht, die Bewegung der Beads mit einer Auflösung von weniger als 10 nm zu kontrollieren [51] . Um dies zu überwinden, wurde AFM verwendet, um die Geschwindigkeit der DNA-Translokation zu kontrollieren [50] und es kann auch gleichzeitig die Kraft und den Blockadestrom messen. In einer Studie unter Verwendung dieses Systems, wie in Fig. 4c gezeigt, wurde DNA an die Spitze einer AFM-Sonde angebunden und dann in den Sondenhalter geklemmt. Durch die Kontrolle der Bewegung der Sonde könnte die DNA-Translokation kontrolliert werden, um ihre Geschwindigkeit zu verringern und sogar die Elektrophorese umzukehren. Außerdem können die Messungen durch Zurückziehen der Spitze auf eine der Länge des Moleküls entsprechende Höhe über der Oberfläche wiederholt werden. Mit AFM-Unterstützung hat die DNA-Detektion in Praxis und Theorie Fortschritte gemacht, da die Detektionsauflösung durch die kombinierten Signale von Blockadestrom- und AFM-Kraftmessungen stark verbessert werden kann [56]. Allerdings gibt es immer noch ein Hindernis bei der Anwendung von Nanoporentechniken beim DNA-Nachweis, nämlich das intermittierende Auftreten regelmäßiger Schwankungen der Kraft (und des Stroms) alle 0,35–0,72 nm, wenn ein DNA-Molekül relativ reibungslos durch eine Nanoporen. Diese Fluktuationen werden auf einzelne Nukleotide zurückgeführt, die sich in einer drehkreuzartigen Bewegung durch die Nanopore bewegen [50].
Studien haben gezeigt, dass eine Stimmgabel, die als Kraftsensor verwendet werden kann, die DNA so steuern kann, dass sie eine Nanopore mit einer Geschwindigkeit im Sub-Nanometer-Bereich passiert [51, 57]. In einer Studie unter Verwendung dieser integrierten Vorrichtung, wie in Fig. 4d gezeigt, wurde ein DNA-Molekül an einer Sondenspitze befestigt, die an einen Zinken einer Stimmgabel geklebt wurde. Zum Halten der Stimmgabel wurde ein Nano-Positioniersystem verwendet, das eine Sub-Nanometer-Genauigkeit besitzt [51]. Die Position der Sondenspitze kann durch den Stimmgabel-basierten Feedback-Kraftsensor erfasst und durch Manipulation des Nanometer-Positioniersystems gesteuert werden. Diese Bewegungsgeschwindigkeit ist 10-mal langsamer als die von DNA, die mit einer optischen Pinzette manipuliert wurde, und 1000-mal langsamer als DNA, die frei durch Festkörper-Nanoporen hindurchtritt [57]. Im Vergleich zu herkömmlichem AFM kann eine Stimmgabel schnellere Scanbewegungen ermöglichen und eine hohe Kraftempfindlichkeit aufweisen, wenn sie in die Flüssigkeit eingetaucht wird. Durch Einbau von TFFS mit einer Nanopore können der Ionenstrom durch eine Nanopore, die Spitzenposition und die Schwingungsamplitude der Spitze gleichzeitig während des Durchgangs eines DNA-Moleküls durch die Nanopore gemessen werden.
Si-Sonde
Alle oben genannten Methoden, nämlich magnetische Pinzetten, optische Pinzetten, AFM und TFFS, müssen die Nanoporenposition scannen. Sie müssen die Nanopore innerhalb der effektiven Länge der DNA lokalisieren, um sicherzustellen, dass die DNA die Nanopore passieren kann. Die Nanoporen-Adressierung ist ein wichtiger Teil dieser Methoden, aber es ist schwierig, DNA an eine große Oberfläche einer Siliziumsonde (Abb. 5) zu binden, die größer ist als die Fläche des Chips, was bedeutete, dass sie die immobilisierte DNA leicht inserieren konnten eine Nanopore, ohne nach dem Ort der Nanopore in der Membran zu scannen. Die Machbarkeit der Verwendung einer DNA-immobilisierten Si-Sonde und eines Positionsreglers zur Steuerung der Bewegung von DNA in und aus Nanoporen wurde demonstriert. Die Schwierigkeit dieser Methode besteht darin, dass die Si-Sonde in die Lösung eingetaucht und durch die Peptidkopplung mit der DNA verbunden wird. Die DNA-Dichte auf der Oberfläche der Sonde ist schwer zu kontrollieren, so dass es wahrscheinlich ist, dass mehrere DNA gleichzeitig eine Nanopore passiert, was den Detektionsstrom beeinflusst.
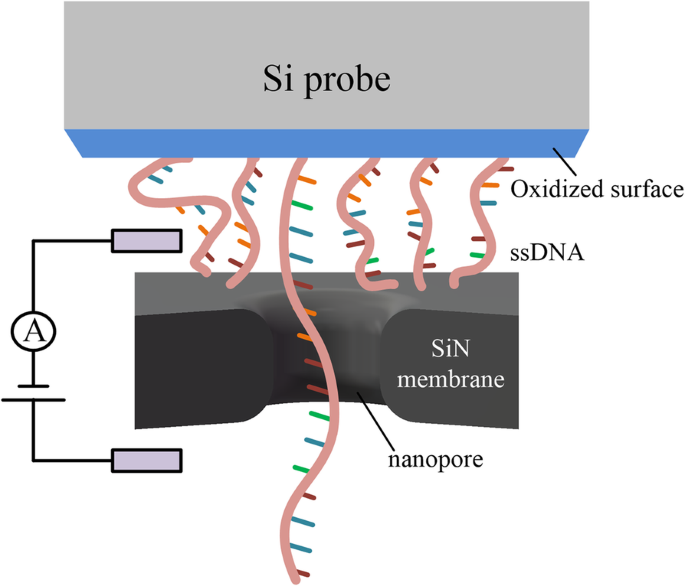
Schema des DNA-immobilisierten Si-Sondensystems zum Nachweis von Nanoporen-DNA [32]
Optische Pinzetten, magnetische Pinzetten, Rasterkraftmikroskopie (AFM) und Stimmgabel-basierte Kraftmessung (TFFS) können die tatsächlichen Kräfte und Position des Moleküls in der Nanopore erkennen, was vielversprechend ist, den DNA-Durchgang durch Nanoporen mit angemessener Geschwindigkeit zu steuern. Die Schwierigkeit, Nanoporen zu adressieren, wird durch die Verwendung einer Si-Sonde vermieden. Darüber hinaus ist die Verwendung eines Zwei-Nanoporen-Systems eine praktikable Methode, um die DNA-Passage durch Nanoporen zu kontrollieren und zu verlangsamen. Darüber hinaus hat eine optische Fallen-Nanopore Potenzial für den zukünftigen DNA-Nachweis. Hier haben wir die DNA-Translokationsgeschwindigkeit einer Festkörper-Nanopore zusammengefasst, die in einige DNA-Kontrollmethoden integriert ist, wie z. .
Schlussfolgerung
Einschichtige 2D-Materialien wie Graphen, MoS2 , WS2 , und h-BN, sind möglicherweise die dünnsten erreichbaren Materialien, da sie so dick sind wie der Abstand zwischen den Nukleotiden. Im Vergleich zu herkömmlichen Festkörper-Nanoporen-Membranen sind einschichtige 2D-Membranen ideal für Nanoporen-Bauelemente, da sie ein hohes Ionenstrom-Signal-Rausch-Verhältnis und relativ große Erfassungsbereiche aufweisen. Sie sind potenziell geeignet, DNA-Sequenzierung durch Kombination mit optischen Pinzetten, magnetischen Pinzetten, AFM, TFFS, Si-Sonden, Zwei-Nanoporen-Systemen oder optischen Fallen-Nanoporen zu realisieren. Bei diesen Techniken sind jedoch mehrere Herausforderungen aufgetreten, die vor der Kommerzialisierung der Nanoporen-DNA-Sequenzierung gelöst werden müssen. Die erste davon tritt auf, wenn sich die Kügelchen oder Sondenspitzen in der Nähe einer Nanopore befinden, wenn es schwieriger ist, DNA-Nukleotide mit Ionenstromsignalen zu unterscheiden. Molekulare Griffe oder andere längere Moleküle sollten verwendet werden, um die Länge eines DNA-Strangs hinzuzufügen, der die durch Perlen oder Spitzen hervorgerufene Wirkung auf das Stromsignal ausgleichen kann. Zweitens sollten Nanoporen-Arrays verwendet werden, um einen Hochdurchsatz und eine parallele Detektion zu realisieren, aber die Technologie der parallelen Detektion ist derzeit noch nicht ausgereift. Drittens ist es gemäß den gegenwärtigen Herstellungsverfahren schwierig, ein Zwei-Nanoporen-System und ein optisches Fallen-Nanoporensystem mit hoher Genauigkeit und Reproduzierbarkeit herzustellen, was für den Nanoporen-DNA-Nachweis sehr wichtig ist. Ein Helium-Ionenstrahl könnte die Schlüsseltechnologie zur Lösung dieses Problems sein [11, 58]. Daher erwarten wir, dass die DNA-Nanoporensequenzierung weiterhin ein Forschungsschwerpunkt sein wird und in weitere neue Ideen und innovative Ansätze integriert werden kann, um niedrige Fehlerraten, schnelle und hohe parallele Aufzeichnungen und lange Leselängen von bis zu 100 Kilobasen zu realisieren.
Verfügbarkeit von Daten und Materialien
Alle während dieser Studie generierten oder analysierten Daten sind in diesem veröffentlichten Artikel enthalten.
Abkürzungen
- 2D:
-
Zweidimensional
- 3D:
-
Dreidimensionale Materialien
- TMDs:
-
Schicht-Übergangsmetall-Dichalkogenid
- SNR:
-
Signal-Rausch-Verhältnis
- PL:
-
Photolumineszenz
- SIBA:
-
Selbstinduzierte Rückwirkung
- AFM:
-
Rasterkraftmikroskopie
- TFFS:
-
Stimmgabelbasierte Kraftmessung
Nanomaterialien
- Festkörperrelais
- DNA-Synthese
- Amazon Alexa Steuern eines Chromecast
- Verbesserung durch drahtlose Überwachung
- Führen Sie durch Zuhören
- Blog:Nachweis von Genen durch Microarray
- IBM-Wissenschaftler messen Wärmeübertragung durch einzelne Atome
- Sicherung des IoT durch Täuschung
- Lochplattierung
- Festkörper-Kohlendioxidsensor



