Analyse der Aktin- und fokalen Adhäsionsorganisation in U2OS-Zellen auf Polymer-Nanostrukturen
Zusammenfassung
Hintergrund
In dieser Arbeit untersuchen wir, wie U2OS-Zellen durch Anordnungen von Polymer-Nanosäulen beeinflusst werden, die auf flachen Glasoberflächen hergestellt werden. Wir konzentrieren uns auf die Beschreibung von Veränderungen in der Organisation des Aktinzytoskeletts und in der Lage, Anzahl und Form von fokalen Adhäsionen. Aus unseren Ergebnissen identifizieren wir, dass die Zellen aufgrund ihres Ausbreitungs- und Adhäsionsverhaltens auf Nanosäulen in verschiedene Regime eingeteilt werden können. Eine quantitative Analyse legt nahe, dass Zellen, die auf dichten Nanosäulen-Arrays ausgesät wurden, oben auf den Säulen aufgehängt sind, wobei sich fokale Adhäsionen näher an der Zellperipherie bilden als flache Oberflächen oder spärliche Säulen-Arrays. Diese Änderung ist analog zu ähnlichen Reaktionen für Zellen, die auf weichem Substrat ausgesät wurden.
Ergebnisse
In dieser Arbeit untersuchen wir, wie U2OS-Zellen durch Anordnungen von Polymer-Nanosäulen beeinflusst werden, die auf flachen Glasoberflächen hergestellt werden. Wir konzentrieren uns auf die Beschreibung von Veränderungen in der Organisation des Aktinzytoskeletts und in der Lage, Anzahl und Form von fokalen Adhäsionen. Aus unseren Ergebnissen identifizieren wir, dass die Zellen aufgrund ihres Ausbreitungs- und Adhäsionsverhaltens auf Nanosäulen in verschiedene Regime eingeteilt werden können. Eine quantitative Analyse legt nahe, dass Zellen, die auf dichten Nanosäulen-Arrays ausgesät wurden, oben auf den Säulen aufgehängt sind, wobei sich fokale Adhäsionen näher an der Zellperipherie bilden als flache Oberflächen oder spärliche Säulen-Arrays. Diese Änderung ist analog zu ähnlichen Reaktionen für Zellen, die auf weichem Substrat ausgesät wurden.
Schlussfolgerung
Insgesamt zeigen wir, dass die Kombination aus Hochdurchsatz-Nanofabrikation, fortschrittlicher optischer Mikroskopie, molekularbiologischen Werkzeugen zur Visualisierung zellulärer Prozesse und Datenanalyse verwendet werden kann, um zu untersuchen, wie Zellen mit nanostrukturierten Oberflächen interagieren, und wird in Zukunft dazu beitragen, Kultursubstrate zu schaffen, die besondere Zellfunktion.
Grafische Zusammenfassung
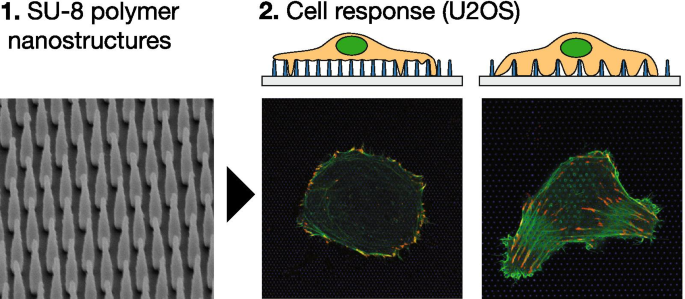
Hintergrund
In vivo befinden sich Zellen typischerweise in einer komplexen 3D-Umgebung, die als extrazelluläre Matrix (ECM) bezeichnet wird. Die ECM dient nicht nur als strukturelles Gerüst für die Zellen, sie ist auch ein Übermittler biomechanischer und biochemischer Signale und reguliert so eine Reihe von Prozessen wie Gewebemorphogenese, Homöostase und Differenzierung. Es besteht aus Wasser, Polysacchariden und Proteinen [1,2,3,4] und die Zusammensetzung variiert je nach Gewebetyp.
Motiviert durch die Notwendigkeit, Zellkulturmodelle zu erstellen, die In-vivo-Bedingungen besser darstellen, haben Forscher zunehmend damit begonnen, das Zellverhalten auch in 3D-Matrizen und in „Semi-3D“-Systemen zu untersuchen. Es wurden eine Reihe von Unterschieden im Zellphänotyp zwischen flachen Substraten und Systemen mit höherer Dimensionalität identifiziert [5, 6]. Es ist beispielsweise bekannt, dass sich Merkmale wie Lebensfähigkeit, Proliferation, Differenzierung und Morphologie zwischen Zellen auf flachen Oberflächen und Zellen, die in 3D-Matrizen eingebettet sind, unterscheiden [3, 7].
In-vivo-ähnliche Substrate reichen von „semi-3D“/2,5D-Substraten, wie z. B. ebenen Oberflächen, die mit verschiedenen Nanostrukturen verziert sind, bis hin zu „echten 3D“-Systemen wie Kollagengelen oder Matrigelmatrizen [8,9,10,11]. Darüber hinaus kann die kontrollierte Positionierung von Liganden auf Oberflächen neue Erkenntnisse darüber liefern, wie Zellen mit verschiedenen chemischen Mustern interagieren [12,13,14]. Auch mechanische Faktoren wie Struktursteifigkeit oder sogar Oberflächenchemie beeinflussen nachweislich die Zellfunktion [15,16,17]. Zu diesem Zweck wurde eine Vielzahl unterschiedlicher Substrate für zelluläre Studien entwickelt [3, 18, 19, 20, 21, 22].
Es wurde auch vorgeschlagen, dass 3D-Kultivierungssysteme die In-vivo-Wirkung eines Arzneimittels genauer vorhersagen könnten und diese Systeme daher Anwendungen in der Arzneimittelforschung finden könnten [16, 23, 24]. Die präzise Kontrolle nanoskaliger topographischer Muster kann auch verwendet werden, um die Zellmorphologie zu regulieren. Falten und Rillen können beispielsweise verwendet werden, um die quergestreifte Ausrichtung von Kardiomyozyten nachzubilden und so physiologisch relevantere Bedingungen besser darzustellen, um verschiedene Krankheiten zu modellieren [25, 26].
Die Verbindung des Zytoskeletts der Zelle mit der ECM wird durch fokale Adhäsionen (FAs) erleichtert, einen Multiproteinkomplex, der Zelloberflächen-Integrine und Gerüstproteine umfasst. Abhängig von einem komplexen Satz regulatorischer Mechanismen bilden und zerlegen sich die FAs mit einer für die Vorwärtsbewegung erforderlichen Umsatzrate, beispielsweise bei der Zellmigration. Von den FAs ist bekannt, dass sie eine mechanische Kraft auf die ECM ausüben, und umgekehrt ist bekannt, dass die ECM, die eine Kraft auf die Zellen ausübt, die Integrinaffinität und -avidität in der Membran beeinflusst [27].
Eines der Proteine, von denen bekannt ist, dass es ein wesentlicher Bestandteil der FA ist, ist Vinculin. Es ist eines der Linker-Proteine, die an der Verankerung von F-Actin am Integrin-Komplex beteiligt sind. Ein Mangel an Vinculin verändert die Zellmorphologie, Adhäsion und Motilität [28] und beeinträchtigt die Fähigkeit der Zellen, Kraft auf das Substrat zu übertragen [29,30,31]. Vinculin ist nicht nur an der mechanischen Verbindung des Aktinzytoskeletts mit den Integrinkomplexen beteiligt, es besitzt auch die Fähigkeit Aktinfilamente zu vernetzen und zu bündeln [32,33,34], bestehende Aktinbündel zu modifizieren [35], Aktinfilamente zu verkappen, nukleieren neue Aktin-Polymerisationsstellen [36] und rekrutieren Aktin-Modifikatoren [37].
Zellen reagieren auf die 3D-Matrizen, indem sie die Anzahl und Art der Zell-Substrat-Adhäsion ändern und Veränderungen in der räumlichen Organisation des Zytoskeletts induzieren. Diese Veränderungen wiederum beeinflussen Verteilung, Größe und Dynamik der gebildeten Adhäsionen [4, 38,39,40,41]. Diese Neuordnung kann zu Veränderungen der Zellproliferation, Morphologie und Motilität führen [42].
Um den Einfluss komplexer 3D-Umgebungen auf Zellen zu verstehen, müssen neue Modellsysteme entwickelt werden, in denen zelluläre Prozesse untersucht und mit flachen Kontrollen verglichen werden können. Da bekannt ist, dass die zelluläre Reaktion von den physikalischen, mechanischen und chemischen Eigenschaften des Kultursubstrats abhängt, ist es wünschenswert, zelluläre Substrate mit genau kontrollierten Eigenschaften herzustellen [43,44,45]. Darüber hinaus ist es von großem Vorteil, wenn die Zellen und das Substrat mit bereits etablierten Analysetechniken wie der optischen Mikroskopie leicht untersucht werden können.
Ein Substrattyp, der in letzter Zeit Aufmerksamkeit erregt hat, sind flache Oberflächen, die mit Nanosäulen oder Nanodrähten verziert sind [18, 21, 46,47,48,49,50,51,52,53]. Im Vergleich zu beispielsweise Hydrogelen imitieren diese strukturierten Oberflächen nicht die echte 3D-Umgebung, sondern weisen eine gut definierte Oberflächentopographie auf. Diese Substrate werden typischerweise als 2,5D bezeichnet. Solche Systeme wurden bereits verwendet, um den Transport biologisch relevanter Moleküle in Zellen zu erleichtern [54, 55], um die enzymatische Aktivität zu überwachen [56], um die Kernmechanik zu testen [57] und um zu untersuchen, wie die Abstimmung der Membrankrümmung verschiedene Zellmembran-bezogene . beeinflusst Prozesse [58,59,60]. Durch die Herstellung von Nanostrukturen auf transparenten Substraten ist es möglich, diesen Ansatz in die optische Mikroskopie zu integrieren.
Die Zahl möglicher Kombinationen verschiedener Zelllinien, Nanostrukturtyp und -geometrie ist groß, und Beispiele aus der Literatur sind reichlich vorhanden. Liet al. beschrieben das Zellverhalten auf Oberflächen, die mit zufällig positionierten Galliumphosphid-Nanostrukturen dekoriert sind, und quantifizierten den Anteil von Zellen mit großen FAs [61]. Die Zell- und FA-Morphologie wurde auf Oberflächen mit verschiedenen Flächendichten von Nanodrähten untersucht. Die Ergebnisse zeigten, dass Zellen, die auf Oberflächen geringer Dichte ausgesät wurden, mit dem Substrat in Kontakt standen und große FAs um die Zellränder herum bildeten. Große FAs wurden in einer hohen Zellfraktion auf diesen Arrays nachgewiesen. Für hohe Nanodraht-Flächendichten wurden die Zellen oben auf den Nanodraht-Arrays aufgehängt und punktförmige FAs unter den Zellen beobachtet. Ein geringerer Anteil der Zellen auf diesen Arrays zeigte im Vergleich zu Zellen auf der Oberfläche mit geringer Nanodraht-Flächendichte große FAs.
Buch-Månson et al. untersuchten Wechselwirkungen zwischen Zellen und nanostrukturierten Oberflächen für Silizium-Nanosäulen-Arrays, die zufällig auf einem Si-Substrat positioniert sind [62]. Bei dem verwendeten Herstellungsverfahren wurde die Flächendichte, aber nicht der Säule-Säulen-Abstand kontrolliert. Die Untersuchung von FAs zeigte, dass Zellen auf den Arrays mit der mittleren Flächendichte die größte Anzahl von FAs aufwiesen, die auch die asymmetrischste Form aufwiesen. Es wurde vermutet, dass sich einige dieser FAs an den Seitenwänden der Nanosäulen bildeten. Dies wurde bei Oberflächen mit niedriger und hoher flächiger Nanosäulendichte nicht beobachtet.
In früheren Arbeiten haben wir detaillierte Protokolle zur Herstellung von SU-8-Polymer-Nanostrukturen auf flachen Glasoberflächen beschrieben [63] und das Zellverhalten für zwei verschiedene Zelllinien auf diesen Oberflächen untersucht [45, 48]. In dieser Arbeit verwenden wir Elektronenstrahllithographie (EBL), um Oberflächen herzustellen, die mit vertikal ausgerichteten SU-8-Polymerstrukturen dekoriert sind, um Veränderungen der Aktin-Zytoskelett- und FA-Organisation in der Osteosarkom-Epithelzelllinie U2OS zu untersuchen. Wir führen sowohl qualitative als auch quantitative Analysen der durch die Oberfläche induzierten Veränderungen mit verschiedenen topologischen Hinweisen durch.
Ergebnisse
Unter Verwendung zuvor etablierter Protokolle haben wir Deckgläser aus Glas hergestellt, die mit genau definierten Arrays von vertikal ausgerichteten SU-8-Nanosäulen (NP) mit variabler Trennung und definierter Geometrie dekoriert sind [63]. Oberflächen mit NP-Flächendichten von 456, 205, 115 und 29 NPs/100 μm 2 (entsprechend den Steigungen von 500 nm, 750 nm, 1000 nm und 2000 nm) verwendet wurden. Zunächst untersuchen wir allgemeine Trends in der Zellmorphologie, der Struktur des Aktinzytoskeletts und der Zell-Substrat-Interaktionen. Wir verfolgen dies durch einen quantitativen Vergleich der Zell- und FAs-Morphologie auf verschiedenen nanostrukturierten Substraten und Flachglaskontrollen. Wir kombinieren hochauflösende Mikroskopie mit Hochdurchsatz-Fertigung, um eine qualitative Analyse von mindestens \({\approx 100}\) Zellen für jeden Oberflächentyp durchzuführen, mit Bildgebung nach 24 h und 48 h. Insgesamt analysieren wir> 400 hochauflösende Bilder und 20 3D-Datensätze.
Abbildung 7 zeigt eine schematische Darstellung der NP-Arrays (A, B) und elektronenmikroskopische Bilder von hergestellten Substraten (C, D). Glasobjektträger mit nanofabrizierten Strukturen wurden unter Verwendung von Paraffin unter einer 35-mm-Hohlbodenkultur befestigt, wobei die Strukturen nach oben zeigen. Von oben nach unten und mit dem Titel versehene Seitenansichten der Elektronenmikroskopie, die in den Fig. 1C, D gezeigt sind, zeigen Nanosäulen-Arrays mit einem Abstand und einer Höhe von 1000 nm. Tabelle 1 zeigt geometrische Parameter der in dieser Arbeit verwendeten Arrays, ihre Klassifizierung sowie die entsprechende NP-Flächenanzahldichte. Wir klassifizieren NP-Arrays in dicht und spärlich, abhängig vom beobachteten Zelladhäsionsverhalten (siehe unten).
Abbildung 1 zeigt repräsentative U2OS-Zellen, die 24 Stunden lang auf Glas (A) und nanostrukturierter Oberfläche (B–F) kultiviert wurden. Die Zellen wurden mit pCMV-LifeAct-GFP und pTAG-RFP-Vinculin cotransfiziert, was die Visualisierung von F-Actin und Vinculin durch die Produktion von fluoreszierenden LifeAct-TagGFP2 (im Folgenden:LifeActGFP) bzw. TagRFP-Vinculin-Fusionsproteinen ermöglicht. F-Aktin-Netzwerk und vinculinreiche Bereiche in FAs werden deutlich nachgewiesen. An der Zellperipherie befindet sich das LifeActGFP-Signal in unmittelbarer Nähe der Membran und daher verwenden wir dieses Signal, um die Zellmorphologie zu visualisieren. Das Signal von SU-8-NPs wird in Blau angezeigt (siehe Abschnitt „Experiment“).
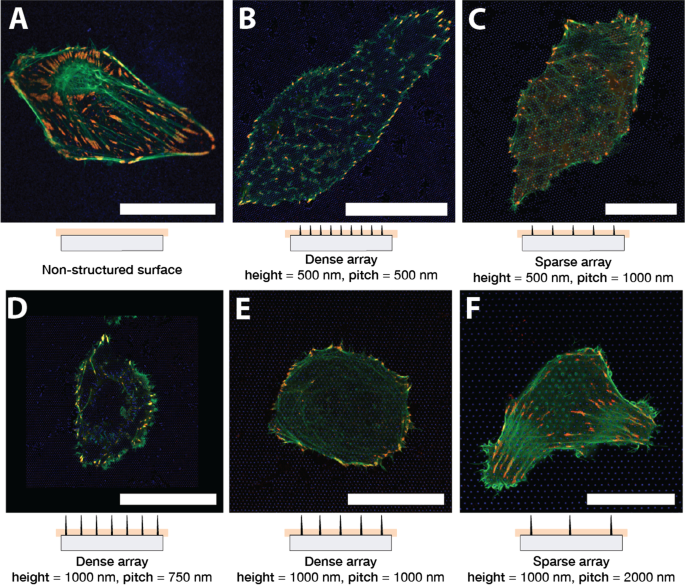
U2OS, das fluoreszierendes LifeActGFP (grün) und TagRFP-Vinculin (rot) auf verschiedenen Oberflächentypen exprimiert. Gelbe Färbung weist auf überlappende Signale von LifeActGFP- und TagRFP-Vinculin-Kanälen hin. Unterhalb jeder mikroskopischen Aufnahme ist eine schematische Seitenansicht des entsprechenden NP-Arrays zusammen mit der ungefähren Position der Aufnahmeebene gezeigt. Zellen auf A . abgebildet unstrukturierte Flachglasoberfläche und auf Pillar-Arrays mit unterschiedlichen Array-Pitch und Strukturhöhen:B 500 nm Abstand und 500 nm Höhe, C 1000 nm Abstand und 500 nm Höhe, D 750 nm Abstand und 1000 nm Höhe, E 1000 nm Abstand und 1000 nm Höhe und F 2000 nm Steigung und 1000 nm Höhe. Alle dargestellten Bilder sind von repräsentativen Zellen. Maßstabsleiste 25 μm. Beachten Sie, dass C hat einen anderen Maßstab als die anderen Bilder
Vorläufige Tests mit transfizierten Zellen zeigten, dass Zellen, die sowohl auf Glas- als auch auf strukturierten Oberflächen ausgesät wurden, nach etwa 6 h vollständig ausgebreitet zu sein schienen. Es wurden keine klaren Unterschiede in der Zellausbreitung zwischen den verschiedenen Oberflächen beobachtet, und eine visuelle Untersuchung ergab keine Anzeichen einer verringerten Zelllebensfähigkeit aufgrund der Säulenanordnungen zu diesem Zeitpunkt oder in späteren Experimenten. In den folgenden Experimenten wurden Zellen 6 h nach der Aussaat transfiziert und dann 24 h, 48 h nach der Transfektion abgebildet, entsprechend der Transfektion 30 h, 54 h nach der Aussaat. Im Folgenden werden diese beiden Zeitpunkte mit der Beobachtungszeit nach der Aussaat bezeichnet, also 24 h, 48 h.
Zellen wurden auf NP-Arrays mit einer Höhe von 500 nm, 1000 nm und einer Steigung von 750 nm, 1000 nm, 2000 nm ausgesät. Nach der anfänglichen Ausbreitung wurde beobachtet, dass die Zellen entweder rund oder länglich waren, ähnlich der Situation auf flachen Oberflächen. Es wurde gefunden, dass diese allgemeine Morphologie über mehrere Experimente hinweg konsistent war. Zellen, die auf spärlichen NP-Arrays ausgesät wurden, hatten im Allgemeinen eine ähnliche Form wie die Zellen auf der Glasoberfläche, siehe Abb. 1F, die eine repräsentative Zelle auf einem 2000 nm großen Array darstellt. F-Aktin-Fasern waren auch an der Basis der NPs und in der Nähe der Glasoberfläche vorhanden, was darauf hindeutet, dass die Zellen in der Lage waren, den substratnahen Bereich zu erreichen. Wie zuvor beobachtet [45, 62, 64], schienen Zellen auf dichten Arrays typischerweise oben auf den NPs suspendiert zu sein (Abb. 1B, D, E). Zellen auf dichten Anordnungen scheinen nahe der Glasoberfläche weniger prominentes F-Aktin zu haben, was darauf hindeutet, dass die Aktinfasern nicht zwischen Säulen in der Nähe des Substrats gebildet wurden. Die Beziehung zwischen NP-Höhe und Abstand bestimmt, ob die Zellen am Substrat hafteten oder oben auf dem NP-Array suspendiert waren. Dies ist beispielsweise in Fig. 1C dargestellt, wo kürzere NPs dazu führen, dass die Zelle das Substrat kontaktiert, während längere NPs den Kontakt behinderten, Fig. 1E. Diese Beobachtungen von Aktinfasern wurden durch Z-Stapeln, die für einige der Oberflächen durchgeführt wurden, weiter bestätigt, wie in Abb. 2 dargestellt.
Um ein detaillierteres Verständnis darüber zu erhalten, wie Zellen an der strukturierten und nicht strukturierten Oberfläche haften, haben wir die Verteilung von FAs untersucht, wie sie durch die Anwesenheit des TagRFP-Vinculin-Fusionsproteins sichtbar gemacht wird. Zellen auf flachen Oberflächen bildeten typischerweise längliche FAs, die unter dem gesamten Zellkörper verteilt waren, wie in Fig. 1A gezeigt.
Auf spärlichen Arrays konnten U2OS die Glasoberfläche zwischen den NPs kontaktieren und adhärierten ähnlich an Zellen auf Glas, gezeigt in Abb. 1C, F. Bei diesen NP-Arrays bildeten sich FAs auf Glas zwischen den NPs und das F-Aktin-Signal wurde auch in dem nahe der Basis der NPs aufgenommenen Bild nachgewiesen. Dies deutete darauf hin, dass die Zellen in der Lage waren, die Membran um die Nanostrukturen herum zu biegen. Zellen auf dichteren Arrays, wie 750 nm, 1000 nm Abstand und 1000 nm Höhe, wurden jedoch deutlich daran gehindert, zwischen den Nanostrukturen am Substrat zu haften, wie in den Bildern 1D, E gezeigt, die nahe der Basis der aufgenommen wurden NPs. Um die Peripherie herum waren die Zellen jedoch typischerweise in der Lage, sich zwischen den Nanostrukturen unter Bildung von FAs an das Substrat anzuheften, was oft durch die Symmetrie des darunter liegenden Säulenarrays gesteuert wurde.
Zellen, die sich auf NP-Arrays mit kürzerer Länge und mit einem Säulenabstand von 1.000 nm ausbreiteten, bildeten Adhäsionen sowohl in Richtung der Peripherie als auch unter dem Zellkörper. Die F-Aktin-Faserorientierung wurde durch die Symmetrie des darunterliegenden Arrays gelenkt, wie in 1C gezeigt. Die Lage und Orientierung der Vinculin-haltigen FAs zeigte jedoch kein klares Muster, wobei FAs zwischen den NPs gebildet wurden.
U2OS-Zellen auf 500 nm-Säulen mit einem Abstand zwischen den Säulen von 500 nm bildeten im Allgemeinen weniger und kleinere Adhäsionen im Vergleich zur ebenen Oberfläche, wie in Fig. 1B gezeigt. Bei Zellen, die auf diesem Array ausgesät wurden, wurden Aktinfasern hauptsächlich in der Nähe der Glasoberfläche an Positionen beobachtet, an denen sie in FAs endeten. Auch dies ist ein Zeichen dafür, dass das Aktinnetzwerk daran gehindert wurde, die Oberfläche zu kontaktieren, und es wurde daher angenommen, dass die Zellen oben auf dem Array suspendiert waren. Die Zellen schienen jedoch ein intakteres F-Aktin-Netzwerk zu haben, das sich über den Säulen bildete. Teile des Aktinnetzwerks, das zwischen den Säulen beobachtet wurde, schienen sich mit Strukturen in der darunter liegenden NP-Anordnung auszurichten. Dies ist in Abb. 1B zu sehen, da sich F-Aktinfasern und FAs überwiegend entlang einer der Gitterrichtungen, d. h. parallel zu offenen „Linien“, des Säulenarrays bilden.
Sowohl auf dichten als auch auf spärlichen Anordnungen beobachteten wir „ringartige“ F-Aktin-Strukturen, die sich um NP bildeten, das nach oben in den Zellkörper hineinragt. Die F-Aktin-Ringstruktur schien auf spärlichen Anordnungen stärker ausgeprägt zu sein, wie in Abb. 1E, F gezeigt.
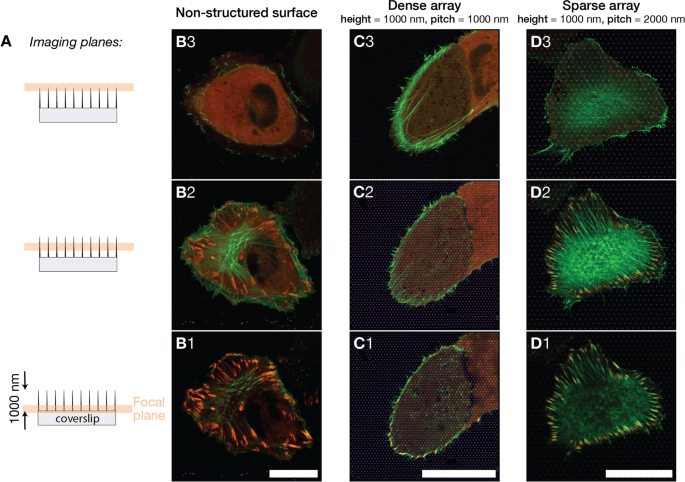
A Bilder von U2OS-Zellen auf verschiedenen Oberflächen wurden in drei angezeigten Fokusebenen aufgenommen. Die Bilder wurden in der Nähe des Säulenfußes, etwa auf halber Säulenhöhe, und nahe der Säulenspitze aufgenommen. Die Abbildungsebenen befanden sich 0,0 μm, 0,4 μm, 0,8 μm von der flachen Glasoberfläche entfernt. B –D ) Zusammengeführte Fluoreszenzbilder, die U2OS zeigen, das fluoreszierendes LifeActGFP (grün) und TagRFP-Vinculin (rot) auf verschiedenen Oberflächentypen exprimiert, gelbe Farbe weist auf überlappende LifeActGFP und TagRFP-Vinculin hin. Die abgebildeten Oberflächen waren B unstrukturierte Glasoberfläche, C 1000 nm gepitchtes Säulenarray, D 2000 nm gepitchtes Säulenarray. Die präsentierten Bilder sind repräsentativ für Zellen auf jedem Oberflächentyp. Der vertikale Abstand zwischen den Fokusebenen für jeden Oberflächentyp beträgt ungefähr 400 nm. Maßstabsleisten 25 μm
Basierend auf den oben präsentierten Ergebnissen haben wir drei Oberflächen für eine detailliertere und quantitative Beschreibung der Zellmorphologie und der FAs ausgewählt. Wir untersuchen Zellen auf dichten Arrays (Pitch 1000 nm, Länge 1000 nm), spärlichen Arrays (Pitch 2000 nm, Länge 1000 nm) und vergleichen die Ergebnisse mit Zellen auf flachen Glasoberflächen, die als Kontrolle verwendet wurden.
Durch den Einsatz des Airyscan-Detektors zusammen mit der dedizierten Bildnachbearbeitung konnten wir eine Bildgebung mit einer xy-Auflösung von ca. 140 nm und einer z-Auflösung von ca. 400 nm durchführen [65]. Abbildung 2 zeigt Bilder von Zellen auf den drei Oberflächen, wobei die Bildebenen etwa 400 nm voneinander entfernt sind. Die Untersuchung von F-Aktin-Bündeln bei verschiedenen z . Zellen auf einer ebenen Oberfläche hatten ein deutlich sichtbares F-Aktin-Netzwerk in derselben Fokusebene oder direkt über den FAs (siehe Abb. 2-B2). Bei Zellen auf dichten Arrays wurde das F-Aktin-Netzwerk in einer höheren Brennebene innerhalb der Zelle gefunden als die FA-Ebene, die mit dem Glasträger in Kontakt stand (Abb. 2-C1 und C2/C3). Für Zellen auf spärlichen Arrays war die Situation ähnlich wie bei den Zellen auf Glaskontrollen und Aktinnetzwerk und FAs wurden in derselben Höhe nachgewiesen (Abb. 2-D2). Diese Daten unterstützen die anfängliche Beobachtung, dass Zellen auf spärlichen Arrays die Oberfläche zwischen den Strukturen anhefteten, während Zellen auf dichten Arrays hauptsächlich in der Lage waren, an der Oberfläche um die Zellperipherie herum zu haften.
Um die Unterschiede in FAs und Zellmorphologie für die drei ausgewählten Oberflächen zu analysieren und zu quantifizieren, wurde ein Python-basiertes Bildanalyseskript verwendet (siehe „Experimental“). Für die quantitative Analyse wurden mehr als 300 hochauflösende Bilder analysiert. In diesen Bildern wurden> 400 Zellen und> 7700 FAs identifiziert, Tabelle 2 listet die Anzahl der nachgewiesenen Zellen und FAs für die drei in die Analyse einbezogenen Oberflächentypen auf. Für alle Oberflächen wurden die Zellen sowohl 24 h als auch 48 h nach der Transfektion abgebildet. In der folgenden Analyse werden geometrische Parameter wie Oberfläche, Rundheit und Aspektverhältnis für Zellen auf 3 Oberflächentypen und nach 24 h, 48 h verglichen. Weitere Analysen finden Sie in den Ergänzenden Informationen. Oberfläche, Zirkularität und Aspektverhältnis für Zellen sind in Abb. 3 und für die Anzahl der FAs, kombinierte FA-Fläche pro Zelle und Anteil von FA-Fläche zu Zelle in Abb. 4 dargestellt. Die geometrischen Parameter wurden wie im „Experimental“ beschrieben definiert. Sektion.
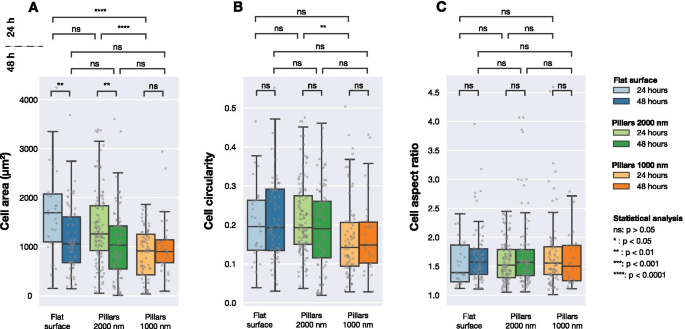
Berechnete Zellfläche, Zellzirkularität und Zellaspektverhältnis für U2OS-Zellen, die fluoreszierende LifeActGFP- und TagRFP-Vinculin-Fusionsproteine auf verschiedenen Oberflächen exprimieren, 24 h und 48 h nach der Transfektion abgebildet. Die Bilderkennung erfolgte anhand der Intensität des LifeActGFP-Signals. Jeder graue Punkt entspricht einer Zelle und die Boxplots zeigen Medianwerte (Q 2) sowie die erste (Q 1) und drittes Quartil (Q 3). Statistische Unterschiede zwischen den Verteilungen wurden mit dem nicht-parametrischen Mann-Whitney-Test ermittelt
Abbildung 3 fasst die gesammelten Daten für ebene und strukturierte Oberflächen zusammen. Wie in Abb. 3A gezeigt, wurden nach 24 h Zellkultur signifikante Unterschiede in der Zellfläche beobachtet. Nach 48 h gab es jedoch keinen signifikanten Unterschied zwischen der durchschnittlichen Zellfläche auf den untersuchten Oberflächen. Bei der Betrachtung der Zellzirkularität (Abb. 3B) wurden keine signifikanten Unterschiede zwischen verschiedenen Oberflächen festgestellt, außer zwischen Zellen, die auf dichten und spärlichen Säulen ausgesät wurden, die nach 24 Stunden abgebildet wurden. Zellen auf allen drei Oberflächen hatten die gleichen durchschnittlichen Seitenverhältnisse, wie in Fig. 3C dargestellt.
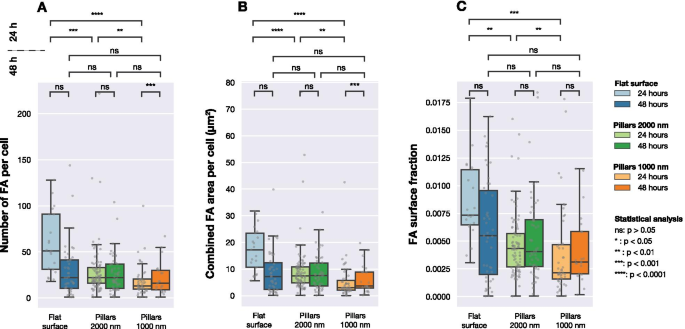
Anzahl der FAs, kombinierte FA-Fläche pro Zelle und Anteil der FA-Fläche zu Zellfläche für Zellen, die auf drei verschiedenen Oberflächentypen abgebildet wurden (Flach- und Säulenarrays mit Abständen von 1000 nm und 2000 nm). Jeder graue Punkt entspricht einer Beobachtung aus einer Zelle. Die statistische Signifikanz zwischen den Verteilungen wurde mit dem nicht-parametrischen Mann-Whitney-Test bestimmt
4 zeigt die Verteilung der Anzahl der detektierten FAs pro Zelle, die Gesamtoberfläche der FAs in jeder Zelle und das Verhältnis von FA-Fläche zu Zellfläche. Nach 24 h war die Anzahl der FAs, die von Zellen auf den drei verschiedenen Oberflächen gebildet wurden, signifikant unterschiedlich. Wie in 4B gezeigt, war die gesamte FA-Oberfläche pro Zelle für Zellen, die auf flachen und strukturierten Oberflächen ausgesät wurden, unterschiedlich. Das gleiche kann man sehen, wenn man die relative Menge an FAs (die Gesamtfläche der detektierten FAs geteilt durch die Gesamtzellfläche) für die verschiedenen Oberflächen vergleicht, wie in Fig. 4C gezeigt.
Nach 48 h Kultur wurden jedoch keine signifikanten Unterschiede zwischen den Zellpopulationen mehr beobachtet. Bei der Betrachtung der Anzahl der FAs pro Zelle, der kombinierten FA-Fläche pro Zelle oder des FA-Oberflächenanteils wurden keine Unterschiede zwischen den drei Oberflächen gefunden.
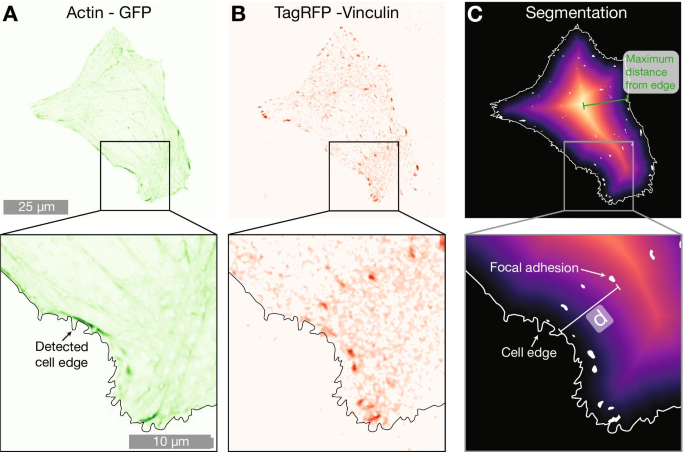
Beispielzelle in einem 2000-nm-Säulen-Array, das A . ausdrückt LifeActGFP (grün) und B TagRFP-Vinculin (rot). Beiden Abbildungen überlagert ist die detektierte Zellkante, die unter Verwendung des Signals der Expression von LifeActGFP bestimmt wurde (wie in A . gezeigt ). C Zeigt eine Entfernungskarte vom erkannten Zellrand sowie erkannte FAs an. Die kürzeste Entfernung (gekennzeichnet mit d in der Abbildung) von jedem segmentierten Vinculinfleck (weiße Bereiche) bis zur Zellperipherie (durch eine durchgezogene weiße Linie gekennzeichnet) wurde für alle FAs in den Bildern berechnet
Um zu verstehen, ob das Vorhandensein von NPs die Lokalisation von FAs in der Zelle beeinflusst, haben wir weitere Analysen unter Verwendung der Position der FAs durchgeführt. Mikroskopiedaten zeigten, dass sich FAs in Zellen auf dichten NP-Arrays näher an der Zellperipherie befanden, wie in den Fign. 1 und 2. Um diesen Trend zu quantifizieren, haben wir den kürzesten Abstand von jedem FA zum Zellrand berechnet. Dies wurde wie in Fig. 5 dargestellt durchgeführt. F-Aktin wurde verwendet, um die Lage der Peripherie zu bestimmen, und durch Erstellen von Distanzkarten wurde die Distanz zwischen jedem Zentrum einer detektierten FA zur Zellperipherie berechnet. Um Unterschiede in der Zellgröße zu berücksichtigen, haben wir die Abstände zwischen dem erkannten Zellrand und FA durch den maximalen Abstand vom Rand zum geometrischen Zentrum für jede Zelle normalisiert (ein Abstand, der dem Radius für Zellen mit kreisförmiger Form entspricht). Die durch den maximalen Abstand normalisierten Daten sind in Abb. 6 und Tabelle 3 dargestellt. Bei Zellen auf ebenen Oberflächen sind die FAs mehr zur Mitte der Zelle hin verteilt, während die FAs sowohl bei dünnbesetzten 2000 nm-Arrays als auch bei dichten 1000 nm-Arrays die FAs befinden sich näher an der Zellperipherie. Dieser Effekt ist am deutlichsten bei dichten Arrays mit 1000 nm Pitch. Ergebnisse eines alternativen Normalisierungsansatzes, bei dem die FA-Positionen nach Zelloberfläche normalisiert wurden, sind in den Zusatzinformationen enthalten. Diese Daten zeigen dieselben qualitativen Trends wie die Daten, normiert durch den maximalen Abstand zum Rand in jeder Zelle, die in Fig. 6 dargestellt ist.
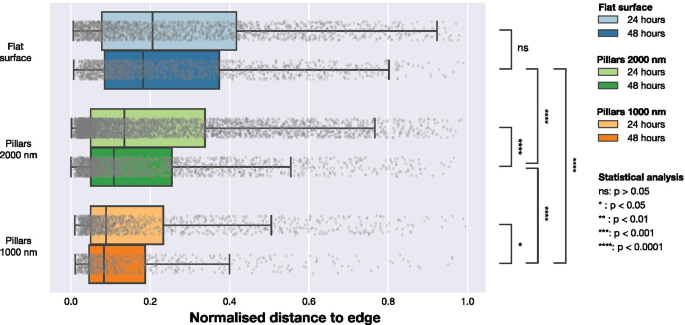
Verteilung der FAs-Positionen in Bezug auf den nächsten Zellrand, normiert auf den maximalen Abstand vom geometrischen Zentrum der Zelle zum Rand. Die Abstände wurden durch Berechnung des Abstands von jedem beobachteten FA zum Zellrand ermittelt, der durch das LifeActGFP-Signal definiert und für die drei Oberflächentypen nach 24 h und 48 h aufgetragen wurde. Graue Punkte stellen einzelne Beobachtungen von FAs dar und ihre Verteilungen sind in den Boxplots zusammengefasst. Um die Wahrscheinlichkeit zu testen, dass die FAs von den verschiedenen Oberflächen und Zeitpunkten aus derselben Verteilung stammten, wurden Mann-Whitney-Tests an allen Verteilungen mit den in der Abbildung angegebenen Signifikanzniveaus durchgeführt
Diskussion
Die Organisation des Aktinzytoskeletts und die Bildung von Adhäsionen sind Prozesse, die auf ebenen Oberflächen ausführlich untersucht wurden. Die vorliegende Studie wurde entwickelt, um Veränderungen in der Organisation des Aktin-Zytoskeletts und fokale Adhäsionen auf Nanosäulen-Arrays zu untersuchen und zu quantifizieren. In dieser Studie haben wir nicht untersucht, wie Nanosäulen-Arrays die Zellmigration beeinflussen. Wir erwarten jedoch, dass Zellen auf den dichten Arrays eine höhere Motilität aufweisen könnten, wie zuvor bei denselben Säulenarrays für embryonale Mausfibroblasten beobachtet [45].
Indem man den Zellen erlaubt, sich über einen längeren Zeitraum auszubreiten und an der Oberfläche anzuhaften, ist es möglich, die Aktin-Zytoskelett-Organisation und das Vorhandensein vollständig ausgereifter FS zu beobachten. 24 h nach der Aussaat beobachten wir signifikante Unterschiede in der Zellfläche, der Rundheit und dem Seitenverhältnis der auf den Oberflächen ausgesäten Zellen. Nach 48 h wurden jedoch keine signifikanten Unterschiede festgestellt, was darauf hindeutet, dass die Zellen nach 24 h noch nicht vollständig an den Oberflächen adhäriert sind und die Nanostrukturen hauptsächlich die FA-Organisation beeinflussen, bevor sie vollständig ausgereift sind. Weder nach 24 h noch nach 48 h ist eine FA-Bildung oben oder an den Seiten der NPs zu beobachten.
Veränderungen in der Aktin-Zytoskelett-Organisation hängen auch damit zusammen, wie eine Zelle mit der Umgebung interagiert. So wachsen beispielsweise sowohl Stressfasern als auch FAs unter Dehnung und scheinen funktionell voneinander abhängig zu sein [66]. Andere haben berichtet, dass bei Zellen, die auf weiche oder nachgiebige Oberflächen ausgesät wurden, häufig rundere Zellen und FA-Lokalisierung um Kanten herum beobachtet werden [67]. In unseren Ergebnissen beobachten wir einen ähnlichen Trend. Zellen auf dichten Anordnungen neigen dazu, Fasern um den Zellrand herum zu zeigen, wie in den Fig. 1 und 2 gezeigt. 1E oder 2B. Bei diesen Zellen scheinen sich FAs nahe am Zellrand zu bilden. Wir spekulieren, dass, wenn Zellen keine flache Oberfläche haben, beispielsweise wenn die Zellen auf den Säulen aufgehängt sind, die FAs-Verteilung der FAs auf weichen Substraten ähnelt, wie sie von Prager-Khoutorsky und Mitarbeitern verwendet werden [67].
Aus unseren Ergebnissen beobachten wir, dass Zellen, die auf den Säulen aufgehängt sind, ein entwickeltes Aktinnetzwerk über den Säulen zu haben scheinen, jedoch ohne FAs, die sich auf den Säulen selbst bilden. Bei Zellen auf spärlichen Arrays scheinen die Zellen jedoch weniger von den NPs beeinflusst zu werden und sowohl das Aktinnetzwerk als auch die FAs erscheinen eher „flache Oberflächen“.
Die Interaktion zwischen FAs und Aktin-Zytoskelett ist komplex und noch nicht vollständig charakterisiert. Es ist bekannt, dass FAs, die das Aktin-Zytoskelett mit der ECM verbinden, als Zugpunkte wirken und die Bildung von Stressfasern in den Zellen fördern. Umgekehrt beeinflussen Aktinfasern wiederum die Organisation und Reifung von FS. Zahlreiche Studien beschreiben, wie Zellen dazu neigen, auf dichten NP-Anordnungen suspendiert zu werden [61, 62, 68] und wie Zellmembranen mit einzelnen NPs interagieren [69, 70, 71]. Diese Beobachtungen werden durch theoretische Studien [64] bestätigt und das zelluläre Verhalten auf Säulen ist ziemlich gut verstanden.
Der Mechanismus hinter der Bildung von FAs und der Anheftung an das Substrat um den Zellrand auf dichten Anordnungen bleibt unklar. Insofern ist der Vergleich mit Zellen auf weichem Substrat besonders interessant. For soft substrates, actin fibres are organised in a ring like fashion close to the cell edge and FAs form around the cell periphery [67]. On the nanopillar arrays similar type of architecture is observed, but the actin fibres are typically shorter. Similar qualitative trends in terms of actin organisation and FA formation were observed by Li et al. for cells seeded on random nanowire arrays made from gallium phosphide [61].
In our studies we also observed formation of F-actin rings around NPs. The formation of F-actin rings around NP has previously been described for fibroblasts on similar surfaces [45] and for U2OS cells on nanostructures with a range of structure sizes [58].
Contrasting our results to other studies highlight an important aspect of studies on cellular response to NP arrays:cellular response may vary considerably depending on cell type, NP material, NP geometry and as well as other parameters. For example, Buch-Månson et al. studied fibroblasts and investigations of FAs showed that cells suspended on arrays with intermediate NP density had the highest number of FAs. In our results we do not see a similar trend. However, these studies cannot be directly compared as Buch-Månson et al. studied another cell line using a system with different array geometry, surface porosity and NPs length [62].
There are also studies describing the effect FAs placement has on cells [41]. By modelling cells on planar substrates Stolarska et al. suggest that the cells can control intra-cellular stresses by three mechanisms:FA position, FA size and attachment strength. FAs around the periphery allows the cells to be more sensitive to changes in the micro-environment. This could also be an underlying mechanisms for cells on NPs. Yet, it is not obvious that the results for the planar substrate are directly transferable to NP decorated surfaces.
Cell-interactions with the surrounding environment, for flat substrate, NPs arrays or in vivo ECM, are regulated by a complex set of relations between actin organisation, membrane mechanics, cell dynamics and contact with FAs. To further explore these relations, applying flat surfaces structured with NPs could be one promising approach. Such surfaces may also aid in exploring discrepancies in the cellular response to environmental cues between different cell lines.
Conclusions
In order to create more physiologically relevant systems for cellular studies, a plethora of 3D and 2.5D approaches have been proposed. One approach is to use flat-surfaces decorated with vertically aligned nanostructures as a simple model system. High resolution live cell imaging of co-transfected U2OS cells expressing pCMV-LifeAct-GFP and pTAGRFP-Vinculin have been used to study the influence of nanopillar arrays on actin cytoskeleton focal adhesion organisation. Our present results indicate that the U2OS cells spreading on surfaces decorated with nanopillars can be categorised into three different regimes by how they respond to the nano-structures. These observed changes are quantified by analysing more than 400 high-resolution images, and indicate that tuning geometrical properties of the nanostructured surface can be used to direct cell behaviour.
More specifically, the U2OS cells were found to either contact the substrate, attach preferably around the cell edge, or be fully suspended on top of the vertical NP arrays. In the latter case, we hypothesise that the resulting reorganisation of FA and cytoskeleton is an effect analogous to what is seen for softer substrates.
Increased understanding of how cells behave on nano-structured surfaces, such as pillar arrays, could help us discover more details about complex cellular processes. For example, it is still poorly understood how changes in the actin cytoskeleton and its architecture influence cell signalling. By studying the cell response on nanostructured surfaces in a systematic way, the potential connection between actin cytoskeleton, cell adhesions and a plethora of biochemical signalling pathways could be further explored. We therefore envision that further development of the presented platform and analysis could have implications for advanced in vitro applications or for development of smarter in vivo biointerfaces.
Methods
Fabrication of Nanostructures and Sample Mounting
SU-8 nanostructures were fabricated as previously explained [63]. Briefly, 24 mm by 24 mm glass cover slips (#1.5, Menzel-Gläser, thickness 170 μm) were cleaned by immersion in acetone, isopropyl alcohol, rinsed in de-ionised water and dried. The cover slips were then oxygen plasma treated for 2 min (Diener Femto plasma cleaner, power 100 W, base pressure 0.3 torr), followed by dehydration for 10 min on a 150 °C hot plate. Samples were then placed in a desiccator containing an open vial of Hexamethyldisilazane (HMDS, Sigma Aldrich product no:440191). HMDS was applied by vapour deposition, the desiccator was pumped to low vacuum using a diaphragm pump for 5 min and the samples were kept in HMDS atmosphere for 60 min.
Substrates for EBL were prepared directly after HMDS treatment by spin coating SU-8 2001 (Microchem Corp.) to a desired thickness of 500 nm and 1000 nm. SU-8 was made fluorescent by adding either Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102 (all Sigma Aldrich) to a final concentration of 100 μg mL −1 widerstehen. After spin coating samples were dehydrated on a hot plate at 95 °C. To mitigate charging during EBL exposure samples were then covered by a layer of conductive polymer AR-PC 5091 Electra 92 (AllResist GmbH) by spin coating at 2000 rpm for 60 s to thickness of 50 nm.
An Elionix ELS-G100 100 kV EBL-system was used to fabricate SU-8 nanopillars (NPs) with processing parameters as described in our previous work [63]. Table 1 summarise the arrays fabricated for this work. Pillar arrays were exposed using the Elionix dot-pattern generator where each pillar is exposed in a single exposure. Arrays were exposed over an area of 2000 μm by 4000 μm, with a current of 500 pA in write fields of 500 μm by 500 μm. NPs had a tip diameter of about 100 nm as a base diameter of 150 nm and 200 nm for structures of length 500 nm and 1000 nm respectively.
After EBL exposure, the samples were rinsed in DI-water to remove the conductive polymer, then post exposure baked for 2.3 min at 95 °C and developed twice in mr-Dev 600 (Micro Resist Technology GmbH) developer for 20 s, rinsed in isopropyl alcohol and dried. Samples were then treated with oxygen plasma (Diener Femto plasma cleaner, power 50 W, base pressure 0.3 torr) for 30 s to render SU-8 hydrophilic and to give it similar surface chemistry as glass by oxidising surface epoxy-groups to hydroxyl.
Fabricated structures were imaged using Scanning electron microscopy (SEM) and samples sputter coated with 5 nm Platinum/Palladium alloy deposited with a 208 HR B sputter coater (Cressington Scientific Instruments UK). SEM was performed with a FEI Apreo SEM, at 5 kV and 0.2 nA with sample 45° pre-titled stage and with additional tilting of 30°.

A Side view schematic representation of nano-structured surface mounted in petri dish. Glass slides are mounted using paraffin such that structures are pointing upwards. B Tilted schematic representation of nano-pillar array on flat surface, and two important parameters for the nano-pillar arrays (height and pitch). These figures are not drawn to scale. C , D Overview of the nanopillar arrays employed in this work. Top-down and tilted side-view scanning electron micrographs of fabricated nano-pillar array with pillars of height 1000 nm and pitch 1000 nm. Scalebars 2000 nm
When exposing the pillars, an indexing system was also exposed to make navigation during live-cell imaging more reliable. Arrays were optically inspected after fabrication to ensure free and standing pillars. The short Oxygen plasma treatment to render the SU-8 structures did not lead to any optically visible change to the structures. Lastly, the samples were mounted underneath 35 mm diameter dishes (Cellvis, Mountain View, CA, USA) with 14 mm holes and nano-structures pointing upwards, as indicated schematically in Fig. 7. As flat surfaces, areas outside the structured part of the same samples were used. Before usage, all dishes were disinfected with 70% ethanol twice and dried.
Cell Culture and Transfection
U2OS-cells (ATCC) were cultivated in Dulbecco’s modified Eagle’s Medium (DMEM Prod. 41965039, Fischer Scientific) with 10% fetal bovine serum (FBS) and kept at 5% CO2 and 37 °C. Before detachment, cells were washed with PBS and detached with Trypsin-ethylenediaminetetraacetic acid (trypsin-EDTA) and seeded on nanostrucutred or flat surfaces. For the diameter 14 mm glass wells 15,000 cells were seeded.
For the standard transfection experiments, cells were allowed 6 h for adhering to surfaces before transfection. U2OS cells were transiently transfected using Lipofectamine 2000 (Invitrogen, Fischer Scientific) by adapting the manufacturer protocol to our system. Briefly, 2 μL Lipofectamine 2000 was added to 50 μL Opti-MEM I Reduced Serum Media (Prod. 11058021, Gibco , Fischer Scientific) and incubated for 5 min at room temperature. Plasmid DNA coding for fluorescent LifeAct-TagGFP2 and TagRFP-vinculin fusion proteins were co-transfected by using 0.5 μg plasmid DNA (vinculin-pTagRFP and pCMVLifeAct plasmids) was diluted in 50 μL Opti-MEM I and incubated at room temperature for 5 min. For co-transfection of TagRFP-vinculin and pCMVLifeAct 0.5 μg of each plasmid was used.
The diluted DNA was added to the diluted Lipofectamine 2000 in a 1:1 ratio, and left to incubate for 20 min at room temperature. 40 μL of the combined transfection complex was then added to each well. After 18 h, 1.5 mL DMEM (Prod. 41965039) supplemented with 10% FBS and 1% 10000U/mL Penicillin-Streptomycin was added to each dish.
For reverse transfection experiments, the same amounts of reactants were used, but the transfection complex was added to a suspension of U2OS cells, and the suspension was then added to the wells.
Microscopy
Live cell imaging was performed usin g a Zeiss LSM 800 Airyscan with an inverted Axio Observer Z1 stand connect to a PeCon compact incubator. Imaging was performed in an humidified environment at 37 °C, with 5% CO2 flow. High resolution imaging was performed using a Zeiss Plan-Apochromat 63x/1.4NA DIC M27 oil objective with Cargille Immersion Oil Type 37 (n =1.51) suited for use at 37 °C. All images were taken using the system optimised pixel size both in-plane (typically 34 nm) and for stacks in the vertical axis (typically 180 nm).
To minimise imaging bias, imaging was performed in a standardised manner where each pillar array was raster scanned and cells expressing both LifeActGFP and Vinculin RFP were imaged. The high resolution images were then processed using a Zeiss algorithm for reconstruction of AiryScan images and exported as CZI-files for further manual and automatised image processing.
Image Analysis
For all cells, cell shape was based on the expression LifeActGFP fusion protein and expression of TagRFP-vinculin was used to identify FAs. Segmentation of images was performed using a script written in Python 3 [72] using CZIfile [73] (version 2017.09.12) for reading the microscopy images in Zeiss-format. The python packages Scipy [74] and Scikit-image [75] were used for multi-dimensional image processing and image segmentation respectively.
To reduce the influence from fluorescence cross-talk from pillars (due to Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102), the pillar/surface channel was used as a background and subtracted from the TagRFP-vinculin imaging channel. A median filter (size:10 pixels) was applied to remove noise from the TagRFP-vinculin channel, followed by classification of the image into regions based on their intensity value using a Multi-Otsu approach. Multi-Otsu thresholding with three classes was applied. The first class was typically the background, the second class constituted the cytosolic vinculin, whereas vinculin rich areas in FAs appeared brighter and could be classified into a third class. The quality of the image segmentation was briefly assessed by comparison to manual segmentation.
Area of cells and vinculin rich regions were described by counting pixel numbers and from this the actual area was found by correcting for the pixel size. Shape geometries were described by fitting each region with an ellipse with the same second-moment as the segmented region. In order to describe the cell area geometry, three measures were used:(1) Aspect ratio defined as the ratio of the ellipse major axis to the minor axis. (2) Circularity given as,
$$\begin{aligned} C =\frac{4\pi *\text{Area}}{\text{Perimeter}^2}, \end{aligned}$$ (1)and roundness given as,
$$\begin{aligned} R =\frac{4*\text{Area}}{\pi *\text{MajorAxis}^2}. \end{aligned}$$ (2)Segmented vinculin areas with a fitted ellipse that were too round (aspect ratio \(\le 1.5\)) or too elongated (aspect ratio \(\ge 8.5\)) were rejected. In addition, vinculin areas smaller than 0.05 μm 2 were filtered out. In order to find the distance between each vinculin area and the cell edge, the shortest euclidean distance between each centroid (the centre of the fitted ellipse for each vinculin area) and the cell edge was calculated.
Statistical Analysis
Statistical comparisons of distributions were performed by using the non-parametric two-tailed Mann-Whitney test neither assuming normal distribution nor equal standard deviation. P -values \(\ge {0.05}\) were considered to represent a non-significant (ns) difference between the two populations. Significant values were denoted with * for p in 0.01 to 0.05, ** for p in 0.001 to 0.01, *** for p in 0.0001 to 0.001 and lastly **** for p \(\le {0.0001}\).
Abkürzungen
- DMEM:
-
Dulbecco’s Modified Eagle’s Medium
- EBL:
-
Electron Beam Lithography
- ECM:
-
Extracellular Matrix
- EDTA:
-
Ethylenediaminetetraacetic Acid
- FA:
-
Focal Adhesion
- FBS:
-
Fetal Bovine Serum
- GFP:
-
Green Fluorescent Protein
- HMDS:
-
Hexamethyldisilazane
- NP:
-
Nanopillar
- RFP:
-
Red Fluorescent Protein
- SEM:
-
Scanning Electron Microscopy
Nanomaterialien
- Beispielschaltungen und Netzlisten
- Spezialbatterien
- Solarzelle
- Graphen- und Polymerverbundstoffe für Superkondensatoranwendungen:ein Rückblick
- In-vitro-Studie zum Einfluss von Au-Nanopartikeln auf HT29- und SPEV-Zelllinien
- Synthese von ZnO-Nanokristallen und Anwendung in invertierten Polymersolarzellen
- Untersuchung der Adhäsionskraft und des Glasübergangs dünner Polystyrolfolien durch Rasterkraftmikroskopie
- Förderung des SH-SY5Y-Zellwachstums durch Goldnanopartikel, die mit 6-Mercaptopurin und einem Neuronen-penetrierenden Peptid modifiziert sind
- Einfluss der elastischen Steifigkeit und Oberflächenhaftung auf das Prellen von Nanopartikeln
- Neueste Fortschritte bei Synthesemethoden und Anwendungen von Silbernanostrukturen



