Identifizierung charakteristischer Makromoleküle von Escherichia coli-Genotypen durch Rasterkraftmikroskop-Nanoskalen-Mechanische Kartierung
Zusammenfassung
Die Kategorisierung von Mikrobenstämmen erfolgt konventionell nach der molekularen Methode, selten werden die morphologischen Eigenschaften der Bakterienstämme untersucht. In dieser Forschung haben wir die makromolekularen Strukturen der Bakterienoberfläche durch mechanisches AFM-Mapping aufgedeckt, deren Auflösung nicht nur durch die nanoskalige Spitzengröße, sondern auch durch die mechanischen Eigenschaften der Probe bestimmt wurde. Diese Technik ermöglichte die nanoskalige Untersuchung membranartiger Strukturen von Mikrobenstämmen mit einfacher Probenvorbereitung und flexiblen Arbeitsumgebungen, wodurch die zahlreichen Einschränkungen in der Elektronenmikroskopie überwunden und biochemische Analysemethoden mit Markierungen ermöglicht wurden. Die charakteristischen Makromoleküle, die sich auf der Zelloberfläche befinden, wurden als Oberflächenproteine angesehen und erwiesen sich als spezifisch für Escherichia coli Genotypen, von denen die durchschnittlichen Molekülgrößen mit Durchmessern von 38 bis 66 nm charakterisiert wurden und die Molekülformen nierenartig oder rund waren. Zusammenfassend lässt sich sagen, dass die makromolekularen Oberflächenstrukturen einzigartige Eigenschaften aufweisen, die mit dem E verknüpft sind. coli Genotyp, was darauf hindeutet, dass die genomischen Auswirkungen auf zelluläre Morphologien mithilfe der mechanischen AFM-Kartierung schnell identifiziert werden können.
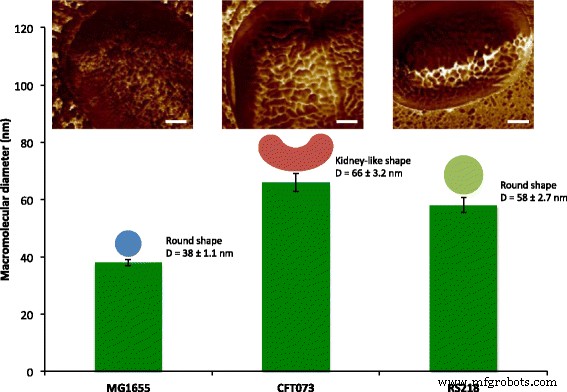
Quantifizierung von Oberflächenmakromolekülen von E. coli Zellen mit mechanischer AFM-Abbildung. Oberflächenmakromoleküle der Zelloberfläche von drei E. coli Genotypen, MG1655, CFT073 und RS218, wurden mit Größen von 38 bis 66 nm und mit runden oder nierenähnlichen Formen charakterisiert. Die Topografiebilder wurden mit Adhäsionskartierung mit den Skalenbalken = 200 nm eingefärbt.
Hintergrund
Aufgrund der minderwertigen Auflösung der optischen Mikroskopie und der eingeschränkten Arbeitsumgebung der Elektronenmikroskopie ziehen Forscher im mikrobiellen Bereich selten das Aussehen von Bakterienzellen in Betracht, sondern wenden stattdessen molekulare oder chemische Analysemethoden zur Identifizierung genomischer Fragmente, Expression von Proteinen usw Es überrascht nicht, dass diese Methoden eine Reihe von Nachteilen haben, einschließlich ihrer arbeitsintensiven und zeitaufwendigen, und daher werden einfachere, effizientere und flexiblere Ansätze benötigt. Das 1982 von Binnig et al. erfundene Rasterkraftmikroskop (AFM) wurde entwickelt, um eine nanoskalige Sonde zu verwenden, die von einem Laserstrahl überwacht wird, um die Oberfläche einer Probe mit nanoskopischer oder sogar atomarer Auflösung zu beobachten [1]. Durch die Abbildung der Morphologie über eine physikalische Sondenspitze überwindet diese Technik die Auflösungsgrenzen und Umgebungsbeschränkungen sowohl der optischen als auch der Elektronenmikroskopie und bietet eine Reihe von Vorteilen, wie z. 3]. Im Hinblick auf mikrobielle Studien umfassen die aktuellen Anwendungen von AFM jedoch hauptsächlich die qualitative Abbildung der statischen oder dynamischen Morphologien von Bakterienzellen oder der Expression von Flagellen und Pili [4,5,6], während sich nur wenige Studien auf die Oberflächenultrastruktur von mikrobielle Zellen und die quantitative Analyse zellulärer Eigenschaften.
In dieser Forschung wurde AFM für die Oberflächenstudie von Escherichia coli . ausgewählt (E. coli ) Zellen, und die Formen und Abmessungen der einzelnen Bakterien wurden durch die AFM-Topographie und Phasenbilder beobachtet. Darüber hinaus ergab eine simultane mechanische Kartierung zusätzliche biomechanische Informationen über die Oberflächenkomponenten, wobei kleine Unterschiede in den Hafteigenschaften zwischen den Makromolekülen und der umgebenden Matrix bei jedem physischen Kontakt zwischen der Spitze und der Probe nachgewiesen werden konnten. Unter Anwendung solch fortschrittlicher Techniken auf die mikrobiellen Felder haben wir drei E. coli Genotypen, die einen Laborstamm und zwei humanpathogene Stämme zur Identifizierung von Oberflächenmakromolekülen enthielten. Die Ergebnisse zeigten, dass die Technik eine Bildauflösung bieten könnte, die die Skala der AFM-Spitze übersteigt, indem die mechanische Verteilung der Probe erfasst wird. Zusammenfassend schlagen wir vor, dass eine solche Entwicklung in der Oberflächenwissenschaft nicht nur Forschern, die auf mikrobiellen Gebieten arbeiten, Details zum zellulären Erscheinungsbild liefern, sondern auch zu unserem Wissen über die Oberflächeneigenschaften anderer Bio- oder Nanomaterialsysteme beitragen würde.
Methoden
Mikrobielle Proben
Die drei E. coli Die in dieser Studie getesteten Stämme wurden klinisch isoliert und vom Labor von Prof. Ching-Hao Teng am Institut für Molekulare Medizin der National Cheng Kung University zur Verfügung gestellt. MG1655 ist der Darm- und Wildtyp-Laborstamm von E. coli K-12 und die beiden anderen Stämme sind menschliche Krankheitserreger – CFT073, die Hauptursache für Harnwegsinfektionen, und RS218, das mit neonataler Meningitis bei Säuglingen in Verbindung gebracht wird [7].
Funktionalisiertes Substrat
Für die kovalente Bindung zwischen der festen Oberfläche und mikrobiellen Zellen wurden zwei Schritte der Oberflächenmodifikation angewendet. Zuerst wurde eine Lösung von 3-Aminopropyltriethoxysilan (APTES, Sigma-Aldrich Co. LLC, USA) verwendet, um ein anfängliches NH2 . zu bilden -funktionalisierte Schicht auf der Oberfläche, wo die stabile APTES-Beschichtung zu den stabilen Si-O-Bindungen beigetragen hat – Si, das von APTES und O von der oxidierten Oberfläche bereitgestellt wird. Mit zwei COOH-Funktionen verbindet sich Glutaraldehyd zu NH2 Funktion von APTES mit einem COOH und arbeitet mit dem NH2 auf der Bakterienoberfläche mit dem anderen COOH.
Die sauberen Substrate wurden eine Stunde lang in eine APTES-Lösung mit einer Mischung aus 5 % APTES in Ethanol getaucht und mit Ethanol und ddH2 . gespült O. Die Objektträger wurden unter Verwendung eines Stickstoffstroms getrocknet und dann über Nacht in eine Glutaraldehydlösung, 2% in PBS, gegeben und mit PBS gewaschen.
Probenvorbereitung
Einzelne Kolonien der E. coli Stämme wurden aus den Lysogenie-Brühe (LB)-Agarplatten ausgewählt und in LB-Brühe inkubiert. Nach den Kultivierungszeiten von 12 h wurde die Bakterienlösung dann 1:100 in frischer voräquilibrierter LB-Brühe verdünnt. Nach weiteren 12 h für die mikrobielle Kultivierung wurde die Bakterienlösung bei 1500 × g . zentrifugiert (4000 U/min) für 3 min und resuspendiert in LB-Brühe, wobei dieser Vorgang zweimal wiederholt wird. 200 Mikroliter der Bakterienlösung wurden auf das funktionalisierte Substrat getropft und 30 Minuten ruhen gelassen. Die Probe wurde dann zweimal in destilliertes Wasser getaucht, um die nicht anhaftenden Zellen zu entfernen, und sofort unter AFM in Umgebungsluft abgebildet.
AFM-Charakterisierung
Für die Oberflächenuntersuchung mikrobieller Proben wurden ein AFM-Instrument (Bruker Nano, Santa Barbara, CA, USA) und eine Siliziumnitrid-Sonde mit der kalibrierten Federkonstante von 0,7 N/m und einem Spitzenradius von 10 nm ausgewählt. Die AFM-Scanrate und die Zeilenpixel betrugen 0,5 Hz bzw. 256 Zeilen für die Scangröße von 10 μm für die erste Erkennung der Topographie, und die Parameter wurden dann auf 0,3 Hz und 512 Zeilen für die Scangröße von 2 μm für eingestellt die Detailbeobachtung. Der quantitative nanomechanische (QNM) Modus von PeakForce wurde für das nanomechanische Mapping verwendet, bei dem die Hafteigenschaften der Oberfläche aus der maximalen Anziehungskraft unter den Rückzugskraft-Abstands-Kurven berechnet wurden.
Unsere vorherige Studie zu Streptococcus mutans zeigten die mechanische Evolution auf der Bakterienoberfläche für 2 h, überwacht durch kontinuierliche mechanische AFM-Kartierung, und es wurde bestätigt, dass die mikrobiellen Proben innerhalb dieser Dauer am Leben blieben [8,9,10]. Um die Lebensfähigkeit des E. coli Proben, die in dieser Arbeit verwendet wurden, wurden die Voruntersuchungen an den Bakterienproben durchgeführt, und die kontinuierliche Änderung der Oberflächenadhäsion für 4 Stunden implizierte, dass die Zellen nach der Probenvorbereitung mindestens 4 Stunden lang am Leben bleiben (siehe zusätzliche Datei 1). Folglich wurden die in dieser Arbeit getesteten mikrobiellen Proben innerhalb von 2 h nach der Probenvorbereitung mit AFM gemessen. Alle E. coli Genotypen wurden einzeln und zu unterschiedlichen Zeiten kultiviert, und die AFM-Messungen wurden unmittelbar nach der Probenpräparation durchgeführt. Mit anderen Worten, die Bakterienproben wurden nicht bis zur Untersuchung aufgereiht, so dass die Auswirkungen der Haltezeit auf die Unterschiede zwischen E. coli Belastungen wurden minimiert. Die quantitativen Daten für jedes E. coli Belastungen wurden aus den innerhalb von 2 h in dieser Studie durchgeführten Messungen gesammelt.
Statistische Analyse
Prism (GraphPad Software, USA) wurde für die statistische Analyse in dieser Arbeit verwendet. Zelllängen und makromolekulare Größen wurden als Mittelwerte zusammen mit dem Standardfehler des Mittelwerts (SEM) dargestellt. Die mehrfachen Vergleiche zwischen E. coli Genotypen wurden unter Verwendung einer gewöhnlichen Einweg-Varianzanalyse (ANOVA) verarbeitet. Das Konfidenzniveau von 95 % (p < 0,05) wurde ausgewählt, und die Sternchen zeigen den Grad des gefundenen signifikanten Unterschieds an. Probennummer n jedes Stammes war> 40.
Ergebnisse und Diskussion
Oberflächen-Ultrastruktur durch multiples Mapping
Beim Scannen mit AFM bei einem Beobachtungsmaßstab von 10 μm an einer Bakterienprobe werden mehrere einzelne E. coli MG1655-Zellen waren zu sehen und die Zellform in drei Dimensionen konnte aus dem topografischen Bild beobachtet werden (Abb. 1a); die klaren Umrisse der Zellen in zwei Dimensionen wurden durch das Ablenkungsfehlerbild erhalten (Abb. 1b), und neben den Bakterienzellen konnten mehrere Tubuli gefunden werden. Die wellenförmigen Filamente (Abb. 1c) stimmten mit dem in einer anderen Arbeit berichteten Auftreten von mikrobiellen Geißeln überein, die einen solchen Befund als Geißeln bestätigten, und die kürzeren und haarähnlichen Pili waren ebenfalls zu sehen [11]. Beim Verkleinern des Beobachtungsbereichs der Sonde für die detaillierte Untersuchung einzelner mikrobieller Zellen zeigte die Topographie einen leichten Unterschied in der vertikalen Richtung zwischen den Zelloberflächen, wie in Abb. 1d gezeigt, und das Ablenkungsfehlerbild schien mehr morphologische Informationen zu liefern, während der erfasste Umgebungslärm war zu groß, um die Ultrastruktur der Zelloberfläche zu untersuchen (Abb. 1e). Als die simultanen biomechanischen Eigenschaften der Probe während des Kontakts zwischen Spitze und Testobjekt gemessen wurden, stellte sich heraus, dass die Bakterienoberfläche tatsächlich aus einer riesigen Menge von Makromolekülen mit spezifischer Form und Größe besteht, wie die Adhäsionskraftkartierung ( Abb. 1f); Daher wurde die morphologische Auflösung in topografischen und Ablenkungsfehlerbildern mit biomechanischen Informationen weiter verbessert.
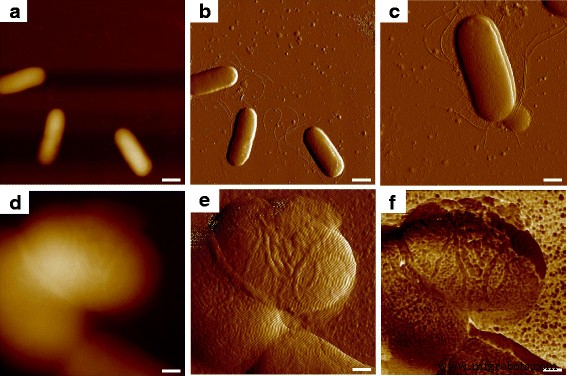
Oberflächenultrastruktur von E. coli MG1655 mit AFM-Mehrfachzuordnung. a und b waren die topografischen und Ablenkungsfehlerbilder und c war das detaillierte Ablenkungsfehlerbild der Bakterienzelle mit der Expression von Flagellen und Pili. Eine einzelne Zelle wurde dann fokussiert, wobei d und e waren die topographischen und Ablenkungsfehlerbilder und f war die entsprechende Adhäsionskartierung. Die Maßstabsbalken = 1 μm in a und b , 500 nm in c , und 200 nm in d –f
In Abb. 2a ist die Expression von Flagellen durch E. coli MG1655 war offensichtlich zu sehen, wo die Größe der Filamente ähnlich der in Abb. 1b war, wobei die Hafteigenschaften relativ geringer waren als die des Substrats. Darüber hinaus wurde festgestellt, dass die Bakterienoberfläche aus kreisförmigen Komponenten besteht, die im Vergleich zur umgebenden Matrix als weniger adhäsiv charakterisiert wurden. Diese Beobachtung ähnelt unseren früheren Ergebnissen an den Gewebeschichten der Haut von Mäusen, und die wiederkehrenden und vergleichbaren Körner wurden als Makromoleküle angesehen, deren Struktur dichter und konsistenter ist als die im intermolekularen Bereich, so dass die Unterschiede in der Adhäsionsleistung leicht zu spüren [12]. Die äußerste Schicht der Zellhülle bei Gram-negativen Bakterien ist eine Schicht aus selbstorganisierenden Proteinen, wie in Abb. 2b dargestellt, die als Oberflächenschicht-(S-Schicht-)Protein bekannt ist [13]. S-Schicht-Strukturen wurden traditionell durch Elektronenmikroskopie gemessen, während die Anforderungen von Vakuumumgebungen und leitfähiger Beschichtung die nativen und Echtzeitinformationen über die Proteine verloren. Obwohl einige Forschungen die S-Schicht-Proteine extrahierten und sie für das AFM-Scannen auf einem Glimmersubstrat wieder zusammensetzten, fehlten die Ergebnisse für die In-situ- und Echtzeit-Performance der S-Schicht-Struktur [14, 15]. Basierend auf der zellulären Architektur und einigen früheren Bildern der mikrobiellen Oberfläche durch Elektronenmikroskopie betrachteten wir die beobachteten Makromoleküle als S-Schicht-Proteine [16].
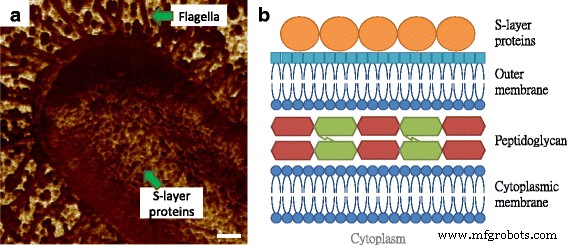
Illustration der vertikalen und Oberflächenstrukturen von E. coli Zellen. a Die Oberflächenmakromoleküle auf E. coli MG1655-Zelle, abgebildet durch AFM-Adhäsionskartierung. b Die molekulare Architektur der Zellhülle in Gram-negativen Mikroben, die aus zytoplasmatischer Membran, Peptidoglycan, äußerer Membran und S-Schicht besteht. Der Maßstabsbalken = 200 nm
Vergleicht man die AFM- und Transmissionselektronenmikroskopie (TEM)-Messungen, hat erstere gegenüber letzteren mehrere Vorteile, wie eine einfachere Probenvorbereitung, weniger eingeschränkte experimentelle Anforderungen und biologisch verträglichere Bildgebungsanwendungen. Die Auswahl von TEM im biologischen Bereich wird im Allgemeinen aufgrund seiner Fähigkeit, die interzellulären Organellen zu sehen, und der ultra-auflösenden (typischerweise Nanometer- oder Sub-Nanometer-) Bildgebung, die erhalten werden kann, ausgewählt. Die AFM-Adhäsionskartierung in der aktuellen Arbeit verbesserte die Merkmalsauflösung und präsentierte die Anordnungen von Oberflächenmakromolekülen in einer Weise, die nicht von der Spitzengröße, sondern von der intrinsischen Struktur der Probe selbst abhing. Darüber hinaus ermöglicht dieser Ansatz eine Auflösung der mikrobiellen Oberfläche im Nanomaßstab über einen Bereich von mehreren zehn Mikrometern im Quadrat.
Genom-manipulierende Unterschiede in morphologischen Eigenschaften
Nach Beobachtung der Oberflächenultrastruktur von E. coli MG1655-Zellen, ist ein interessantes Thema, wie die Makromoleküle in anderen Stämmen angeordnet sind. Die humanpathogenen E. coli CFT073 und RS218 wurden daher mit AFM mit den gleichen experimentellen Parametern untersucht, und es gab keine signifikanten Unterschiede in den Zellformen und -abmessungen zwischen diesen drei Genotypen bei der Merkmalsgröße von 10 μm, wie in Abb. 3a–c zu sehen. Adhäsionskraftkartierung wurde verwendet, um die S-Schicht-Proteine zu identifizieren, und unterschiedliche Strukturen mit verschiedenen Formen und Größen wurden unter den verschiedenen E. coli Dehnungen, wie in Abb. 3d–f gezeigt. Um den Vergleich zu erleichtern, sind die detaillierten Adhäsionskartierungsbilder des E. coli Stämme wurden in Abb. 3g–i dargestellt. Die Oberflächenmakromoleküle wurden in MG1655- und RS218-Zellen als runde Form charakterisiert, obwohl sie unterschiedliche Moleküldurchmesser von 38 ± 1,1 nm (n = 80) für MG1655 und 58 ± 2,7 nm (n = 46) für RS218. Auf der anderen Seite besaßen die CFT073-Zellen eine einzigartige Form von S-Schicht-Proteinen, die nierenartig waren, mit einem Längenunterschied zwischen zwei Endpunkten von 66 ± 3,2 nm (n = 44). Nach der Analyse der Größen der S-Schicht-Proteine dieser drei Genotypen für mehrere Vergleiche zeigten die Ergebnisse die signifikanten Unterschiede zwischen diesen Stämmen (Abb. 4).
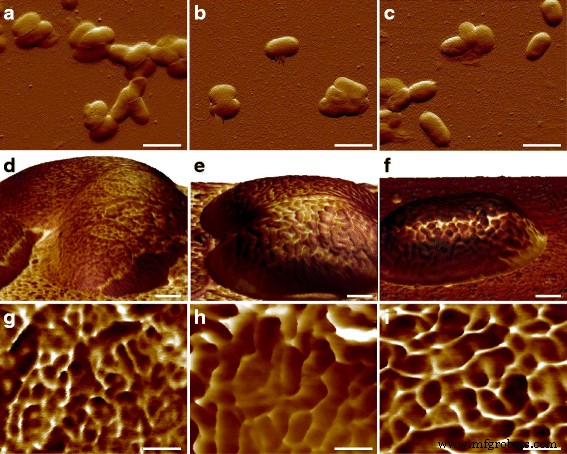
Morphologische Merkmale des E. coli Genotypen. Die obere Reihe zeigte die Ablenkungsfehlerbilder von a MG1655, b CFT073 und c RS218. Die mittlere Reihe zeigte die 3D-Topographien eingefärbt mit Adhäsionskartierung auf d MG1655, e CFT073 und f RS218-Zellen. Die untere Reihe war die detaillierte Adhäsionskartierung auf g MG1655, h CFT073 und i RS218. Bei der Adhäsionskartierung bedeutet die dunklere Farbe weniger Adhäsionsleistungen und umgekehrt. Die Maßstabsbalken betrugen 2 μm für a –c , 200 nm für d –f , und 100 nm für g –ich
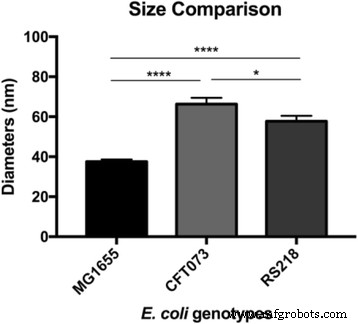
Molekülgrößen von E. coli Genotypen. Die Durchmesser der Oberflächenproteine wurden durch AFM erfasst und über eine Einweg-ANOVA für die Mehrfachvergleiche verarbeitet. ****p < 0,001 und *p < 0.05
Es wird berichtet, dass mikrobielle S-Schichten eine wichtige Rolle bei vielen Funktionen spielen, die den Schutz von Zellen vor schweren Umgebungen, Phagozytose-Angriffen und räuberischen Bakterien beinhalten. Darüber hinaus dienen S-Schichten auch als Adhäsin, das die effektive Besiedelung ermöglicht [17]. Die S-Schicht-Strukturen wurden durch TEM gut untersucht und in verschiedene Gittertypen mit einem Abstand zwischen Mitte und Mitte im Bereich von 4–35 nm kategorisiert [16]. Die Abweichung zwischen unseren AFM-Ergebnissen und TEM-Berichten aus der Literatur wurde als unterschiedliche Bildgebungsmethoden angesehen, wobei TEM die 2D-Morphologie der S-Schicht-Struktur liefert und AFM die 3D-Topographie erfasst, die die zahlreichen Einflüsse umfasst, die durch zelluläres Radiant und Rauheit und die Geometrie der AFM-Sonde.
Ursprünglich wurden verschiedene Arten von S-Schicht-Strukturen für möglich gehalten, um ihre verschiedenen taxonomischen Eigenschaften zur Unterscheidung zwischen Bakterienarten zu nutzen, obwohl dann festgestellt wurde, dass die mikrobiellen Färbungen sogar für einzelne Arten unterschiedliche Proteingitter aufweisen können [13, 16, 18, 19]. Während einige Studien die Rolle der S-Schicht bei der Filamentbildung untersuchten, wurden die Proteintypen auf Zellmembranen und die genomischen Größenunterschiede zwischen E. coli Genotypen wurden die Unterschiede in den S-Layer-Proteinen selten festgestellt [20,21,22]. Die Ergebnisse der aktuellen Studie zeigten die Unterschiede in den morphologischen Merkmalen zwischen E. coli MG1655, CFT073 und RS218 und legen nahe, dass das Aussehen der Oberflächenmakromoleküle wahrscheinlich spezifisch für das Individuum E war. coli Genotyp.
Schlussfolgerungen
In dieser Arbeit wurden genomspezifische nanostrukturelle Informationen über die Bakterienoberfläche durch mechanisches AFM-Mapping erfasst, das die adhäsiven Unterschiede zwischen den Makromolekülen und der umgebenden Matrix unterschied. Die Oberflächenmakromoleküle der mikrobiellen Zellen wurden entsprechend der molekularen Architektur gramnegativer Mikroben als Oberflächenschichtproteine betrachtet. Es wurde festgestellt, dass die Anordnungen und Größen dieser Makromoleküle für das getestete E spezifisch sind. coli Genotypen mit unterschiedlichen Formen und Größen, wobei sich diese Unterschiede durch statistische Analysen als signifikant erwiesen haben. Zusammenfassend betrachten wir die bakterielle S-Schicht-Struktur als genomabhängig und kann die potenzielle Methode für die schnelle Diagnose von mikrobenassoziierten Krankheiten oder mikrobiellen Stämmen sein. Um die praktische Anwendung der S-Schicht-Charakteristik umzusetzen, ist für den Komplementkatalog die Untersuchung auf weitere bakterielle Genotypen erforderlich. Wir bauen derzeit die Datenbank auf, die die morphologischen Eigenschaften von Bakterien und die physiologischen/pathologischen Leistungen verbindet, und glauben, dass dies ein vielversprechender Fortschritt für die praktische Anwendung der AFM-Untersuchung sein wird.
Abkürzungen
- AFM:
-
Rasterkraftmikroskop
- ANOVA:
-
Einseitige Varianzanalyse
- APTES:
-
3-Aminopropyltriethoxysilan
- LB:
-
Lysogenie-Brühe
- QNM:
-
Quantitative Nanomechanik
- S-Schicht:
-
Oberflächenschicht
- TEM:
-
Transmissionselektronenmikroskopie
Nanomaterialien
- IBM-Wissenschaftler erfinden ein Thermometer für die Nanoskala
- AFM-Pioniere mit Kavli-Preis ausgezeichnet
- Der nächste Schwall des großen Öls ist im Nanobereich
- Nichtleiter leiten Strom im Nanobereich
- Biogene Synthese, Charakterisierung und Bewertung des antibakteriellen Potenzials von Kupferoxid-Nanopartikeln gegen Escherichia coli
- Oberflächeneffekt auf den Öltransport in Nanokanälen:eine molekulardynamische Studie
- Kleinwinkelstreuung von nanoskaligen Fettfraktalen
- Lokalisierte Oberflächenplasmonenresonanz-Abhängigkeit von falsch ausgerichtetem abgeschnittenem Ag-Nanoprismen-Dimer
- Hochdurchsatz-Herstellung hochwertiger Nanofasern unter Verwendung eines modifizierten Elektrospinnens mit freier Oberfläche
- Herstellung von nanoskaligen Gruben mit hohem Durchsatz auf Polymerdünnschicht unter Verwendung der AFM-Spitzen-basierten dynamischen Pfluglithographie



