Magnetischer Graphen-Feldeffekttransistor-Biosensor für den Einzelstrang-DNA-Nachweis
Zusammenfassung
Hier wurde ein magnetischer Graphen-Feldeffekttransistor-Biosensor durch die Übertragung eines Graphenfilms aus der chemischen Gasphasenabscheidung auf ein Glassubstrat hergestellt, um einen Sensorfilm und einen leitenden Kanal herzustellen. Durch Fixieren von 1-Pyrenbutansäuresuccinimidylester auf einem Graphenfilm als Anker wurde ein Sondenaptamer auf dem Graphenfilm immobilisiert, um magnetisch markierte komplementäre einzelsträngige DNA einzufangen. Unsere Experimente zeigten, dass die Impedanz des Biosensors in einem periodischen Magnetfeld eine periodische Schwingung aufwies, deren Amplitude mit der komplementären DNA-Konzentration korrelierte. Basierend auf diesem Prinzip wurde der magnetische Graphen-Feldeffekttransistor verwendet, um einzelsträngige DNA mit einer Nachweisgrenze von 1 pM nachzuweisen. Die Ergebnisse wurden unter Verwendung eines Modells erklärt, bei dem die magnetische Kraft bewirkt, dass sich der DNA-Strang verbiegt, was zu einer magnetischen Nanobead/DNA-Modulation der doppelten leitfähigen Schicht von Graphentransistoren führt. Da außerdem ein periodisches Magnetfeld eingeführt werden könnte, um periodische Impedanzänderungen von MGFETs zu erzeugen, könnte die Abtastintegration verwendet werden, um das Signal-Rausch-Verhältnis effizient zu verbessern, indem die Anzahl der Perioden des externen Magnetfelds erhöht wird. Daher wurde in dieser Arbeit ein neuartiger Biosensor für den DNA-Nachweis mit hoher Empfindlichkeit vorgestellt. Basierend auf dem Nachweisprinzip kann dieses System auch ein potenzielles Werkzeug zum Nachweis anderer Biomoleküle, Zellen usw. sein.
Einführung
Der DNA-Nachweis ist von großer Bedeutung für das Studium der Molekularbiologie und die Diagnose genetischer Erkrankungen [1,2,3]. Bis heute wurden verschiedene Biosensoren für den DNA-Nachweis entwickelt, darunter fluoreszierende Biosensoren [4, 5], elektrochemische Biosensoren [6,7,8,9] und Feldeffekttransistor(FET)-Biosensoren [10,11,12,13 ], wobei letztere aufgrund ihrer hohen Sensitivität und Spezifität große Aufmerksamkeit auf sich gezogen haben. Kaistiet al. [12] entwickelten einen FET-Biosensor zum Nachweis von unmarkierter einzelsträngiger DNA unter Verwendung von Peptidnukleinsäuresonden. Kimet al. [13] stellten einen DNA-Ladungssensor vom FET-Typ her, der auf der standardmäßigen komplementären Metalloxid-Halbleitertechnologie basiert.
Aufgrund seiner hohen spezifischen Oberfläche, seiner hohen elektrischen Leitfähigkeit und seiner hervorragenden Elektronenmobilität wurde Graphen als ideales Material für die Herstellung von FET-Biosensoren angekündigt [14,15,16]. Cai et al. [15] entwickelten einen Graphen-FET (GFET)-Biosensor für den ultrasensitiven Nachweis von DNA über Peptid-Nukleinsäure-DNA-Hybridisierung. Unsere Gruppe hat auch einen Mehrkanal-GFET-Biosensor vorgeschlagen, um die Bindungskinetik und Affinität von DNA-Hybridisierung und Einzelbasen-Fehlpaarung zu bestimmen [16].
Bei einem herkömmlichen GFET erzeugt ein elektrisches Feld der externen Gate-Elektrode eine doppelte leitfähige Schicht an der Grenzfläche zwischen dem Graphenfilm und dem Lösungselektrolyten [17,18,19]. Basierend auf einem Captive-Modell von GFETs [16] lädt und entlädt die Gate-Elektrode die doppelt leitende Schicht durch den Elektrolyten, wodurch die GFET-Leitfähigkeit moduliert wird. Daher hängt die Leitfähigkeit eines GFET von der Intensität des externen elektrischen Felds und der Ionenkonzentration im Elektrolyten ab.
Während der Forschung wurde festgestellt, dass die Forschung zur Empfindlichkeit von GFETs das fM-Niveau erreicht hat. Ping et al. [20] und Zheng et al. [21] haben über konventionelle GFET-Biosensoren mit einer Nachweisgrenze im fM-Bereich berichtet. Die obige Literatur erreicht jedoch eine extrem hohe Empfindlichkeit durch Halbleiteranalysator-Detektion, die für praktische Anwendungen teuer und unbequem ist. Darüber hinaus werden Ag/AgCl-Elektroden häufig als externe Gate-Elektroden verwendet, die aufgrund ihrer Größe und Wiederverwendbarkeit für den Bau integrierter Biosensoren ungeeignet sind.
Hier wurde ein magnetischer GFET-(MGFET)-Biosensor entwickelt, bei dem ein magnetisches Feld anstelle eines elektrischen Felds verwendet wird, um die GFET-Leitfähigkeit zu modulieren. Der leitfähige Kanal wurde unter Verwendung eines Graphenfilms durch chemische Gasphasenabscheidung (CVD) erreicht, der mit zwei Indium-Zinn-Oxid-(ITO)-Elektroden auf ein Glassubstrat übertragen wurde. Der Graphenfilm wurde mit 1-Pyrenbutansäuresuccinimidylester (PBASE) funktionalisiert, um die Verknüpfung eines Sondenaptamers zum Einfangen und Hybridisieren mit komplementärer magnetisch markierter einzelsträngiger DNA (cDNA) zu ermöglichen. Durch Anlegen eines periodischen Magnetfelds an die Rückseite der MGFETs wurde eine periodische elektrische Impedanz des MGFET erreicht. Außerdem stand die Fluktuation der elektrischen Impedanz der MGFETs in einem periodischen Magnetfeld mit der Konzentration der cDNA in Beziehung. Ein entsprechendes im Labor hergestelltes Detektionsgerät wurde konstruiert, um die MGFET-Impedanz in Echtzeit zu detektieren. Da das Magnetfeld nicht direkt mit den MGFETs in Kontakt steht, sind die hierin hergestellten MGFETs einfacher zu integrieren und anzuwenden als herkömmliche GFET-Biosensoren. Die Herstellung von MGFETs, der Aufbau des im Labor hergestellten Detektionssystems und das Detektionsprinzip wurden in diesem Artikel ausführlich beschrieben.
Methoden
Materialien und Instrumente
Ein Glassubstrat mit ITO-Elektroden wurde von Hua Nan Xiang Cheng Ltd. (China) bezogen. Das Sonden-Aptamer, die cDNA und die fehlgepaarte DNA wurden von Sangon Biotech Inc. (Shanghai, China) bezogen. Die Sequenz des Sondenaptamers war (5′-NH2 -TGG ACC CCC TCA TAA CGC CTC CTT TTC-FAM-3′), Sequenz der komplementären DNA war (5′-NH2 -GAA AAG GAG GCG TTA TGA GGG GGT CCA-3′), die Sequenz der vollständig fehlgepaarten DNA war (5′-NH2 -TCC CCT TCT TAT GGC CTG TTT TTC AAC-3′) und die Sequenz der fehlgepaarten Einzelbasen-DNA war (5′-NH2 .). -GAA AAG GAG TCG TTA TGA GGG GGT CCA-3′). PBASE und Dimethylsulfoxid (DMSO) wurden von Sigma-Aldrich (Shanghai, China) bezogen. Mit Carboxylgruppen modifizierte magnetische Nanoperlen (MBs) (10 mg/ml) wurden von Xianfeng Nano Material Technology Co., Ltd. (Nanjing, China) bezogen. 1-Ethyl-3-(3-dimethylaminopropyl)carbodiimid-Hydrochlorid, N-Hydroxysuccinimid, Natriumdodecylbenzolsulfonat (SDS) und Natriumdodecylsulfat-phosphatgepufferte Kochsalzlösung (PBS, P5368-10PAK; pH 7,4) wurden von Sigma-Aldrich (Shanghai) bezogen , China).
Ein Raman-Mikroskopsystem (SPEX-1403, SPEX) wurde verwendet, um die Qualität von Graphen zu charakterisieren sowie die Funktionalisierung von MGFETs zu verifizieren. Ein Fluoreszenzphotometer (LS55, PerkinElmer) wurde verwendet, um die Kopplung magnetischer Nanopartikel an cDNA zu charakterisieren. Ein im Labor hergestelltes Datenerfassungssystem wurde verwendet, um die Impedanz von MGFETs in Echtzeit aufzuzeichnen.
Kopplung von cDNA an MBs
Nach 20 Minuten gleichmäßiger Dispergierung durch Ultraschall wurde eine 20 µl Suspension von mit Carboxylgruppen modifizierten MBs mit 200 µl 1-Ethyl-3-(3-dimethylaminopropyl)carbodiimidhydrochlorid (2 µg/ml) und 200 µl N- Hydroxysuccinimid (2 µg/ml) für 15 Minuten, um aktivierte MBs zu erhalten [22, 23]. Dann wurden 20 µl cDNA-Lösung zu der MBs-Lösung gegeben und 2 Stunden lang bei Raumtemperatur unter kontinuierlichem sanftem Schütteln inkubiert. Dann wurde ein Magnetfeld angelegt, um die cDNA-Proben durch MBs anzureichern. Die magnetischen Nanobeads/DNA (MB/cDNA)-Konjugate wurden dreimal mit PBS gewaschen und zur zukünftigen Verwendung in PBS dispergiert.
Herstellung von MGFETs
Die Herstellung von MGFETs wird im Folgenden ausführlich beschrieben. Zunächst wurde ein CVD-Graphenfilm auf eine Glasplatte als leitenden Kanal zwischen den beiden ITO-Elektroden übertragen (Abb. 1a), wie zuvor beschrieben [18, 19]. Zweitens wurde in DMSO gelöstes PBASE (10 mM) für 12 h bei Raumtemperatur in die MGFETs injiziert und durch π-π-Stapelung vollständig mit Graphen reagieren gelassen (Abb. 1b). Die MGFETs wurden dann nacheinander mit DMSO und PBS gewaschen, um jegliche nicht umgesetzte PBASE zu entfernen. Drittens wurden 2 µM des Sonden-Aptamers in die MGFETs eingebracht und mit PBASE 4 Stunden lang bei Raumtemperatur inkubiert, damit das Sonden-Aptamer ausreichend mit PBASE reagieren konnte (Abb. 1c). Die MGFETs wurden dann jeweils dreimal mit 0,2% SDS gewaschen, um ungebundenes Sonden-Aptamer zu entfernen.
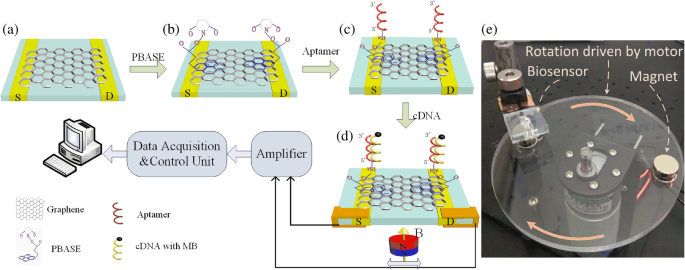
Funktionalisierung und Detektionsprinzip der MGFETs. a Durch chemische Gasphasenabscheidung gewachsener Graphenfilm. b Funktionalisierung von Graphen durch PBASE. c Immobilisierung von Sondenaptamer über PBASE. d Hybridisierung des Sonden-Aptamers mit cDNA. e Foto des Erkennungsgeräts
Ergebnisse und Diskussion
Charakterisierung von MGFETs
Ein durch das CVD-Verfahren hergestellter Graphenfilm wurde als leitender Kanal zwischen zwei ITO-Elektroden auf ein Glassubstrat übertragen (Abb. 1a). Der übertragene Graphenfilm wurde mit dem Raman-Spektrum charakterisiert (Abb. 2). Das Auftreten der drei charakteristischen Peaks des Graphens demonstrierte die erfolgreiche Übertragung des Graphenfilms auf das Glassubstrat [24, 25]. Das Intensitätsverhältnis zwischen dem 2D-Band und dem G-Band (I2D /IG ) zeigte, dass das übertragene Graphen ein mehrschichtiger Film war [26]. Außerdem ist das Intensitätsverhältnis zwischen dem D-Band und dem G-Band (ID /IG ) war klein, was auf eine sehr geringe Defektdichte hinweist.
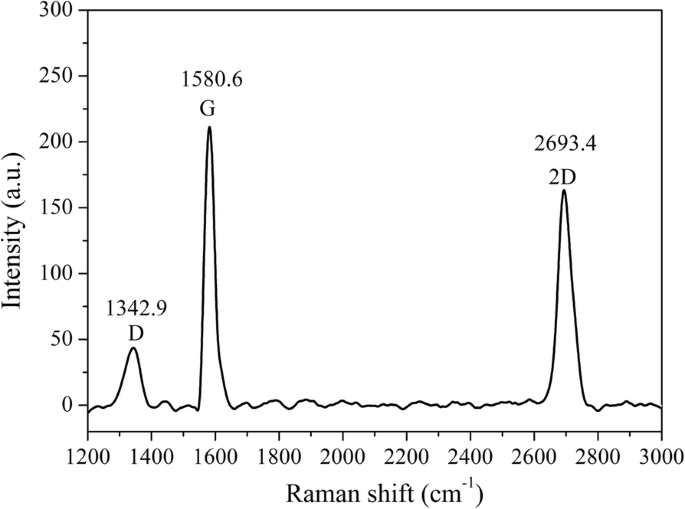
Raman-Spektrum
Aufgrund des Fehlens funktioneller Gruppen waren die Aptamerketten auf dem CVD-Graphenfilm schwer zu modifizieren. Daher wurde PBASE basierend auf seiner aromatischen Pyrenylgruppe auf den Graphenfilmen über π-π-Stapelung als Linker modifiziert. Am anderen Ende von PBASE könnte der Succinimid-Anteil von PBASE an das 5′-NH2 . gekoppelt werden -markiertes Sonden-Aptamer basierend auf der N-Hydroxysuccinimid (NHS)-Vernetzungsreaktion (Abb. 1c). Um die Bindung des Sonden-Aptamers auf dem Graphenfilm zu beurteilen, wurde das 3′-Ende des Sonden-Aptamers mit dem FAM-Fluorophor markiert (Sequenz:5′-NH2 -TGG ACC CCC TCA TAA CGC CTC CTT TTC-FAM-3′). Unmittelbar nach der Einführung des Aptamers wurde die Fluoreszenzintensität offensichtlich erhöht, was auf ihre erfolgreiche Modifikation auf der Graphenoberfläche hinweist (Abb. 3). Eine Erhöhung der Aptamerkonzentration der Sonde führte zu einer Zunahme der Fluoreszenzintensität, die einen konstanten Wert erreichte und daher eine Sättigung der Sondenaptamer auf den MGFETs bei ungefähr 2 µM anzeigt. Daher wurden nachfolgende Experimente bei einer Sondenaptamerkonzentration von 2 μM durchgeführt.
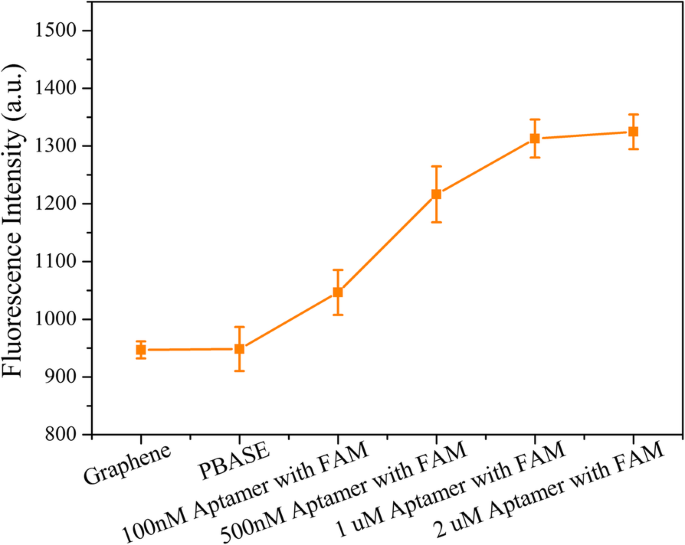
Charakterisierung der Modifikation von MGFETs durch Sondenaptamer. Fehlerbalken repräsentiert die Standardabweichung von 5 unabhängigen Analysen
Charakterisierung von MB/cDNA
Die Morphologie der MBs und MB/cDNA-Konjugate wurde durch Transmissionselektronenmikroskopie (TEM) charakterisiert (Abb. 4a, b). Die Partikelgrößenverteilung von MBs zeigte eine durchschnittliche Partikelgröße von ungefähr 7 nm (Abb. 4c). Um Sensitivität und Genauigkeit bei der Biosensorik für cDNA sicherzustellen, sollten MBs für cDNA zu groß sein, um cDNA vollständig einzufangen. MBs in einer Konzentration von 4 µg/ml wurden aktiviert, um die Bindung an die hier verwendeten cDNA-Proben sicherzustellen. Durch Markierung von cDNA durch FAM wurde die Fluoreszenzintensität ausgenutzt, um die Kopplungseffizienz zu charakterisieren und die cDNA-Konzentration zu optimieren (Abb. 4d). Tatsächlich nahm die Fluoreszenzintensität des Überstands offensichtlich nach der Einführung von MBs in die cDNA-Lösungen ab, was darauf hindeutet, dass cDNA von den MBs eingefangen und angereichert wurde. Das erfolgreiche Einfangen von cDNA durch MBs wurde durch die Beobachtung bestätigt, dass bei einer cDNA-Konzentration von 10 nM die Fluoreszenzintensität des Überstands der von PBS entsprach, was darauf hindeutet, dass die gesamte cDNA eingefangen und von MBs angereichert wurde (Abb. 4d ).
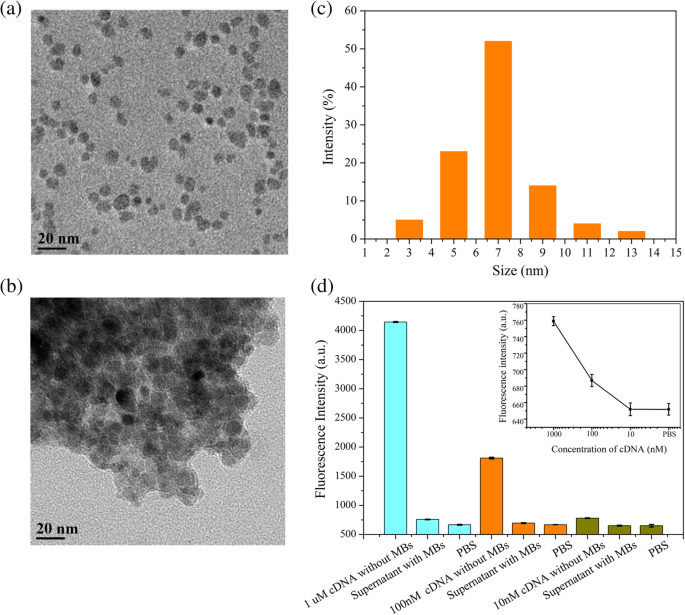
Charakterisierung der MB/cDNA-Kopplung. a TEM von MB. b TEM von MB/cDNA-Konjugaten. c Partikelgrößenverteilung von MBs. d Charakterisierung der MB/cDNA (FAM)-Kopplung. Fehlerbalken repräsentiert die Standardabweichung von 5 unabhängigen Analysen
Analyse der Magnetfeldstärke
MB/cDNA-Konjugate wurden in die MGFETs für 10&supmin;&sup7; gegeben, um eine vollständige cDNA-Hybridisierung mit dem Sonden-Aptamer zu ermöglichen. Da das Sondenaptamer ohne die modifizierten Aminogruppen nicht mit MBs koppeln konnte, konnten die überschüssigen MBs durch dreimaliges Waschen der MGFETs mit PBS entfernt werden. Daher verblieben nur die MB/cDNA-Konjugate auf den MGFETs (Abb. 1d). An einem rotierenden Motor wurde ein Permanentmagnet angebracht, um ein periodisches Magnetfeld an die MGFETs anzulegen (Abb. 1e). Ein im Labor hergestelltes Detektionsgerät wurde verwendet, um die Impedanzfluktuation der MGFETs aufzuzeichnen.
Da die Impedanz von MGFETs durch ein Magnetfeld als Backgate moduliert wurde, wurde die Korrelation zwischen der Magnetfeldstärke und der Impedanz von MGFETs untersucht, um die Parameter der Magnetfeldstärke zu optimieren (Abb. 5). Es wird allgemein angenommen, dass die zwischen dem Graphen und dem Elektrolyten gebildete doppelte leitfähige Schicht durch das externe elektrische Feld moduliert wird, wodurch die Leitfähigkeit von GFETs moduliert wird [19, 27, 28]. Bei MGFETs wurde durch die magnetische Kraft zwischen den MBs und dem Magnetfeld der Abstand zwischen MB/cDNA-Konjugaten und dem Graphenfilm mechanisch gesteuert, wodurch die doppelte leitfähige Schicht von MGFETs moduliert wurde [29, 30]. Die Impedanz von MGFET-Biosensoren variierte mit zunehmender Magnetfeldstärke in drei Stufen, was dadurch erklärt werden könnte, dass die MB/cDNA-Kette als elastischer dünner Stab betrachtet wird [31]. Die erste Stufe trat in dieser Arbeit bei einer Magnetfeldstärke von weniger als 100 mT auf. Basierend auf dem elastischen Dünnstabmodell von DNA-Ketten ist es schwierig, eine Biegung des DNA-Strangs durch die Magnetfeldkraft zu bewirken, da die Magnetfeldkraft geringer ist als die radiale Stützkraft des DNA-Strangs; daher sind die MGFETs nicht empfindlich gegenüber dem Magnetfeld. In der zweiten Stufe mit einer magnetischen Feldstärke von 100 bis 200 mT reicht die magnetische Feldstärke aus, um die radiale Stützkraft des elastischen DNA-Dünnstabs zu überwinden, was zu einer schnellen Biegung der MB/cDNA und dann zu einer empfindlichen Reaktion von . führt die MGFETs an das Magnetfeld. Schließlich erreicht in der dritten Stufe mit einer Magnetfeldstärke über 220 mT die Biegung des elastischen DNA-Stabs ihre Grenze; daher reagieren die MGFETs nicht auf die Änderung des Magnetfelds, was zu einer stabilen Impedanz der MGFETs führt, wie in Fig. 5b gezeigt.
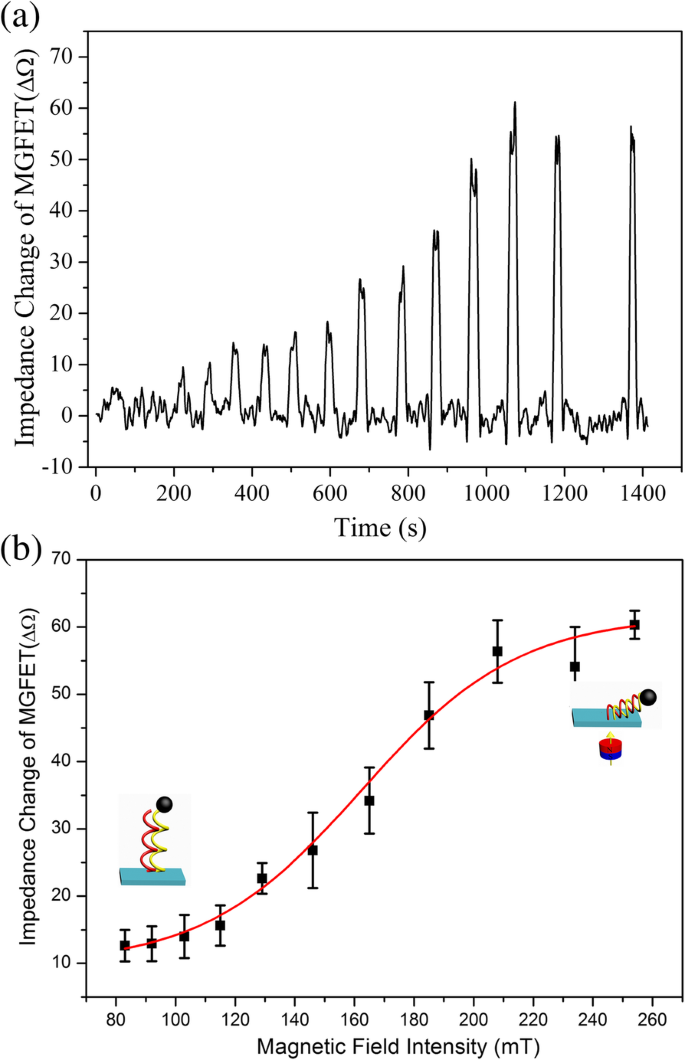
Einfluss der magnetischen Feldstärke auf die Impedanz von MGFETs. a Impedanz von MGFETs bei variierender magnetischer Feldstärke im Zeitbereich. b Zusammenhang zwischen Impedanz von MGFETs und Intensität des Magnetfelds. Fehlerbalken repräsentiert die Standardabweichung von 5 unabhängigen Analysen
Nachweis von cDNA
Die Änderungen der MGFET-Impedanz bei unterschiedlichen MB/cDNA-Konjugatkonzentrationen wurden bei einer festen Magnetfeldstärke von 240 mT gemessen, um die Durchführbarkeit und Empfindlichkeit für den cDNA-Nachweis zu bestimmen.
Die MGFET-Impedanz bei jeder cDNA-Konzentration wurde in Echtzeit aufgezeichnet (Abb. 6a). Wenn ein Permanentmagnet auf die Rückseite der MGFETs geladen wurde, stieg die Impedanz schnell an. Umgekehrt wurde, wenn ein periodisches Magnetfeld angelegt wurde, eine periodische Änderung der Impedanz beobachtet. Basierend auf dieser Impedanzperiodizität wurde ein Sample-Integration-Algorithmus (SIA) verwendet, um das Signal-Rausch-Verhältnis der MGFETs zu erhöhen. Der Zeitraum ohne Anlegen eines Magnetfelds war T0 und die Periode mit Anlegen des Magnetfelds war TM (Abb. 6a) könnte die SIA mit den folgenden Schritten beschrieben werden:(1) während T0 , alle durch Rauschen erzeugten Datenpunkte wurden auf Null normiert, (2) die Datenpunkte, die während jedes TM . erhalten wurden Zeitraum wurden der Reihe nach abgetastet und gemittelt. Nach einer SIA-Verarbeitung über vier Zyklen wurde die periodische Impedanzänderung in MGFETs erhalten, wie in Fig. 6b gezeigt. Theoretisch könnte das Signal-Rausch-Verhältnis der MGFETs durch ausreichend lange Abtastzeiten effektiv verbessert werden.
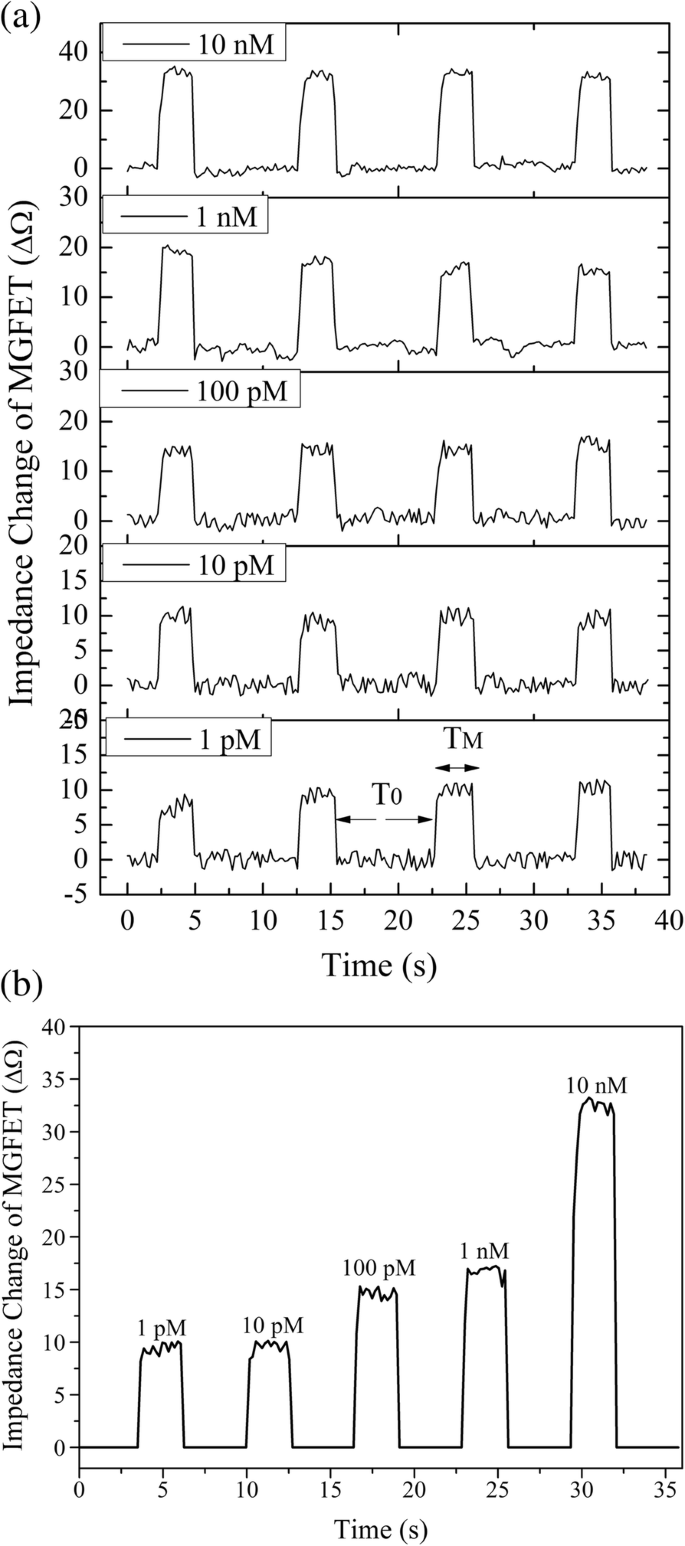
a Zeitbereich von Impedanzschwankungen bei unterschiedlichen cDNA-Konzentrationen. b Impedanzänderungen von MGFETs in Abhängigkeit von der cDNA-Konzentration
Die Impedanzänderungen in MGFETs korrelierten positiv mit der cDNA-Konzentration (Abb. 6b). Die Korrelation zwischen der Impedanzänderung von MGFETs und der Konzentration der cDNA wurde untersucht (Abb. 7). Die hohe Empfindlichkeit der MGFET-Biosensoren in dieser Arbeit basiert hauptsächlich auf den folgenden zwei Aspekten:erstens könnte die mechanische Bewegung von MB/cDNA-Konjugaten den Modulationseffekt auf die doppelt leitfähige Schicht im Vergleich zu DNA allein verstärken, und zweitens da ein periodisches Magnetfeld angelegt werden konnte, um periodische Impedanzänderungen von MGFETs zu erzeugen, basierend auf dem Abtastintegrationsprinzip, wurde nur die MGFET-Impedanz mit dem Magnetfeld abgetastet und integriert, um das Rauschen zu reduzieren. Daher könnte das Signal-Rausch-Verhältnis des Systems stark optimiert werden, indem die Anzahl der Perioden des externen Magnetfelds erhöht wird.
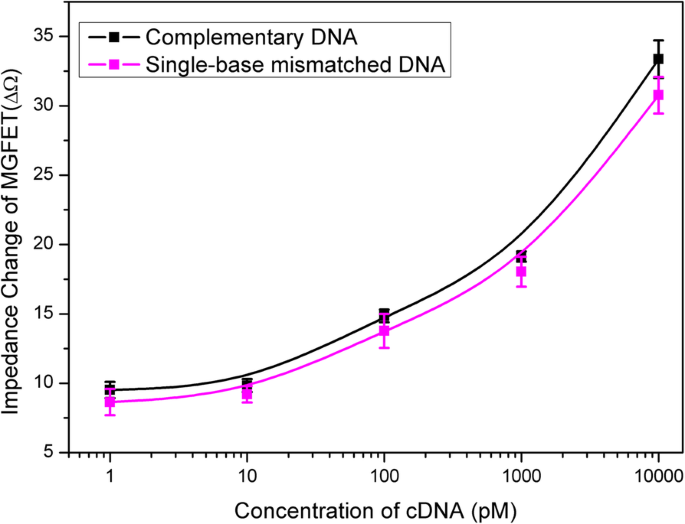
Beziehung zwischen Impedanz von MGFETs und Konzentration der Ziel-DNA. Fehlerbalken repräsentiert die Standardabweichung von 5 unabhängigen Analysen
Selektivität der MGFETs
Die Spezifität der MGFETs wurde durch den Nachweis von zwei verschiedenen Ziel-DNA-Sequenzen bewertet, einschließlich vollständig fehlgepaarter DNA-Ketten und fehlgepaarter Einzelbasen-DNA-Ketten. Ähnlich dem oben beschriebenen Verfahren wird eine vollständig fehlgepaarte DNA (Sequenz:5′-NH2 -TCC CCT TCT TAT GGC CTG TTT TTC AAC-3′) und Einzelbasen-fehlgepaarte DNA (Sequenz:5′-NH2 -GAA AAG GAG TCG TTA TGA GGG GGT CCA-3′) wurden jeweils an MBs gekoppelt. Die in PBS-Lösung gelöste fehlgepaarte MB/DNA wurde 10 min lang in die MGFET-Biosensoren gegeben, um ausreichend mit dem Aptamer zu reagieren. Die MGFETs wurden dreimal mit PBS gewaschen, um die fehlgepaarte DNA zu entfernen. Da das Konjugat von MB/DNA nicht mit Aptamer hybridisieren konnte, wurden bei vollständig fehlgepaarten DNA-Ketten fast alle MB/DNA-Konjugate entfernt. Daher hat die Zugabe von vollständig fehlgepaarter MB/DNA fast keinen Einfluss auf die Leitfähigkeit von Graphen, wie in Abb. 8 gezeigt, was auf eine hohe Selektivität des Biosensors hinweist. Darüber hinaus haben wir auch die Selektivität der Biosensoren durch fehlgepaarte Einzelbasen-DNA-Ketten untersucht, wie in Abb. 7 gezeigt der nicht komplementäre Zielstrang bei jeder bestimmten Konzentration. Daher könnte der fehlgepaarte Einzelbasenstrang in dieser Arbeit nachweisbar sein. Obwohl das Aptamer und die komplementären DNA-Ketten alle kommerzielle Produkte sind, die hauptsächlich die Selektivität der Biosensoren bestimmt haben, haben die MGFETs und ihr Detektionssystem auch zur hohen Sensitivität für die DNA-Detektion beigetragen.
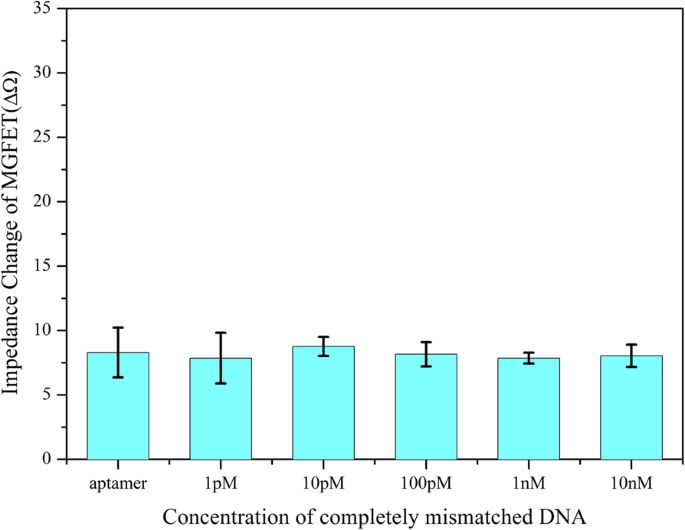
Beziehung zwischen der Impedanz von MGFETs und der Konzentration von vollständig fehlgepaarter DNA. Fehlerbalken repräsentiert die Standardabweichung von 5 unabhängigen Analysen
Schlussfolgerungen
Hier wurde ein MGFET-Biosensor basierend auf Graphen und magnetischen Nanopartikeln zum Nachweis von cDNA vorgestellt. In den MGFETs wurden magnetische Nanopartikel am Ende der cDNA-Sequenz modifiziert. Durch die magnetische Kraft zwischen den MBs und dem Magnetfeld wurde der Abstand zwischen den MB/cDNA-Konjugaten und dem Graphenfilm mechanisch gesteuert, wodurch die doppelte leitfähige Schicht der MGFETs moduliert wurde. Darüber hinaus können wir auch schlussfolgern, dass die Impedanz der MGFETs für einen bestimmten DNA-Strang die Belastung des DNA-Strangs widerspiegelt, die wiederum die Biegung des DNA-Strangs widerspiegelt (Einschub, Abb. 5b). Somit haben die gegenwärtigen MGFETs das Potenzial, bei der Untersuchung der mechanischen Parameter von DNA-Ketten verwendet zu werden. Daher können die MGFETs nicht nur als Biosensor für den cDNA-Nachweis fungieren, sondern möglicherweise auch die mechanischen Parameter von DNA-Ketten nachweisen.
Verfügbarkeit von Daten und Materialien
Alle während dieser Studie generierten oder analysierten Daten sind im Artikel enthalten.
Abkürzungen
- cDNA:
-
Komplementäre magnetisch markierte einzelsträngige DNA
- Lebenslauf:
-
Chemische Gasphasenabscheidung
- DMSO:
-
Dimethylsulfoxid
- FET:
-
Feldeffekttransistor
- GFET:
-
Graphen-Feldeffekttransistor
- MB:
-
Magnetische Nanoperlen
- MGFET:
-
Magnetischer Graphen-Feldeffekttransistor
- NHS:
-
N-Hydroxysuccinimid
- PBASE:
-
1-Pyrenbutansäuresuccinimidylester
- PBS:
-
Natriumdodecylsulfat-Phosphat-gepufferte Kochsalzlösung
- SDS:
-
Natriumdodecylbenzolsulfonat
- SIA:
-
Beispielintegrationsalgorithmus
- TEM:
-
Transmissionselektronenmikroskopie
- ITO:
-
Indium-Zinn-Oxid
Nanomaterialien
- Blog:Nachweis von Genen durch Microarray
- Nanodiamanten für magnetische Sensoren
- Demonstration eines flexiblen Graphen-basierten Biosensors für den empfindlichen und schnellen Nachweis von Eierstockkrebszellen
- Graphen- und Polymerverbundstoffe für Superkondensatoranwendungen:ein Rückblick
- Kohlenstoff-Nanodots als Dual-Mode-Nanosensoren für den selektiven Nachweis von Wasserstoffperoxid
- Ein hochempfindlicher elektrochemischer DNA-Biosensor aus Acryl-Gold-Nanokomposit zur Bestimmung des Geschlechts von Arowana-Fischen
- Temperaturabhängige Kristallisation von MoS2-Nanoflocken auf Graphen-Nanoblättern für die Elektrokatalyse
- Ultrasensitiver Biosensor zum Nachweis von Vibrio cholerae-DNA mit Polystyrol-Co-Acrylsäure-Komposit-Nanosphären
- Empfindliche Methode zur Erkennung von Transistordefekten
- Festkörpersensor zur Erkennung und Charakterisierung elektrischer Felder



