Ultrasensitiver Biosensor zum Nachweis von Vibrio cholerae-DNA mit Polystyrol-Co-Acrylsäure-Komposit-Nanosphären
Zusammenfassung
Ein ultrasensitiver elektrochemischer Biosensor zur Bestimmung von pathogenen Vibrio cholerae (V . Cholerae ) DNA wurde auf der Grundlage einer Polystyrol-Co-Acrylsäure (PSA)-Latex-Nanosphären-Gold-Nanopartikel-Komposit (PSA-AuNPs)-DNA-Trägermatrix entwickelt. Zur Messung der Biosensor-Antwort wurde die Differenzpulsvoltammetrie (DPV) unter Verwendung eines elektroaktiven Anthraquninon-Oligonukleotid-Markers verwendet. Die Beladung der DNA-Latex-Partikelelektrode mit Gold-Nanopartikeln (AuNPs) hat den Faradayschen Strom der DNA-Hybridisierung deutlich verstärkt. Zusammen mit der Verwendung einer berichteten Sonde hat der Biosensor eine hohe Empfindlichkeit gezeigt. Der DNA-Biosensor ergab einen reproduzierbaren und breiten linearen Reaktionsbereich auf die Ziel-DNA von 1,0 × 10 –21 bis 1,0 × 10 –8 M (relative Standardabweichung, RSD = 4,5 %, n = 5) mit einer Nachweisgrenze (LOD) von 1.0 × 10 –21 M (R 2 = 0.99). Der Biosensor erzielte zufriedenstellende Erholungswerte zwischen 91 und 109% (n = 3) für die Erkennung von V . Cholerae DNA in gespikten Proben und könnte für sechs aufeinanderfolgende DNA-Assays mit einem Wiederholbarkeits-RSD-Wert von 5 % (n = 5). Die Reaktion des elektrochemischen Biosensors war stabil und konnte bis zu einer Lagerzeit von 58 Tagen bei 95 % der ursprünglichen Reaktion gehalten werden.
Hintergrund
Vibrio cholerae , ein durch Lebensmittel übertragbarer Krankheitserreger, kann beim Menschen durch akute wässrige Diarrhoe Cholera-Epidemien auslösen. Der Cholera-Ausbruch ist in einigen Teilen der Welt immer noch ein ernstes Problem, z.B. Asien und Afrika und führt zu einem niedrigen sozioökonomischen Status [1,2,3,4]. Dieser enterische Erreger ist eine der Hauptursachen für Morbidität und Mortalität, insbesondere in Entwicklungsländern [5]. Die Epidemie und Pandemie der Cholera in verschiedenen Regionen werden hauptsächlich durch V. Cholera Serogruppen O1 und O139 [1, 2, 6]. V. Cholera Serogruppe O1 hat zwei Hauptserotypen, nämlich Inaba und Ogawa, die sich bei Cholera-Epidemien abwechseln können. Der dritte Serotyp, Hikojima, existiert ebenfalls, ist jedoch selten und instabil. Die für die O1-Antigenbiosynthese verantwortlichen Gene wurden als rfb bezeichnet. Die Mutation, die die Serotypen Inaba und Ogawa definiert, ist eine einzelne Deletionsmutation im rfbT-Gen [7]. Es wurde jedoch auch berichtet, dass gelegentlich lebensmittelbedingte Ausbrüche bei Menschen mit schwerem Durchfall durch Nicht-O1/Nicht-O139 V verursacht werden. Cholera durch die Aufnahme von nicht ausreichend gekochten Meeresfrüchten [8] oder die Exposition gegenüber einer kontaminierten aquatischen Umgebung [9]. Die erste Epidemie von V. Cholera O139 trat 1992 in Bangladesch und Indien auf und breitete sich dann schnell auf andere Länder Südostasiens aus [10]. Weltweit wurden der Weltgesundheitsorganisation (WHO) im Jahr 2005 insgesamt 131.943 Cholera-Fälle und 2272 Todesfälle gemeldet [11].
Die Suche nach einer wirksamen Methode zur Überwachung oder Diagnose von toxigenen V. Cholera Bakterien sind unerlässlich, um die Cholera-Epidemie zu kontrollieren. Traditionelle Identifizierung von V. Cholera wird häufig durch Isolierung und Screening der Bakterien erreicht, wobei eine Voranreicherung mit alkalischem Peptonwasser (APW) gefolgt von einer Isolierung von V. Cholera auf dem Thiosulfat-Citrat-Gallensalz-Saccharose-Agar (TCBS)-Kulturmedium und Identifizierung durch Objektträger-Agglutinationstest mit spezifischen Antiseren [12]. Diese Technik ist jedoch sehr zeit- und arbeitsintensiv, und das mehrere Tage später erhaltene Ergebnis hätte eine Verzögerung der klinischen Diagnose und der Patientenbehandlung bedeutet. Molekulares Verfahren mit PCR-Amplifikation zum Nachweis von V. Cholera [13] hat die Diagnosezeit verkürzt [14], aber die PCR-Methode erfordert qualifizierte professionelle und teure Infrastruktur, die in Ländern mit geringen Ressourcen nur schwer durchzuführen ist. Für den diskreten oder gleichzeitigen Nachweis von V wurden auf dem Prinzip der Immunchromatographie basierende Schnelltests beschrieben. Cholera Serogruppen O1 und O139. Einige andere Immunoassay-basierte Techniken zum Nachweis von V. Cholera sind wie Enzyme-Linked Immunosorbent Assay (ELISA), Koagglutination, Immunfluoreszenz und Quarzkristall-Mikrowaage (QCM). Die meisten dieser Techniken erfordern jedoch hochentwickelte Instrumente, lange Testzeiten und hochqualifiziertes Personal mit detaillierten technischen Kenntnissen [15,16,17,18,19,20].
Elektrochemische Methoden haben aufgrund ihrer hohen Sensitivität, Spezifität, Einfachheit und ihres ökonomischen Protokolls sowie ihrer schnellen Detektion und Kompatibilität mit der Mikrofabrikationstechnologie große Aufmerksamkeit beim Nukleinsäurenachweis auf sich gezogen [21, 22]. Darüber hinaus können elektrochemische Verfahren, die mit Miniaturisierungstechnologien gekoppelt sind, für die dezentrale In-situ-Analyse verwendet werden, beispielsweise der mikrofluidische Chip-basierte DNA-Biosensor, der für die praktische Anwendung sehr nützlich ist [23]. Es gibt eine breite Palette von Elektroden, die bei elektrochemischen Messungen verwendet werden, wie z. B. Glaskohlenstoffelektroden (GCE), Kohlepastenelektroden (CPE), Goldelektroden und Platinelektroden. In letzter Zeit konzentrierten sich Studien auf die Verwendung von Siebdruckelektroden (SPEs) aufgrund einiger ihrer einzigartigen Eigenschaften, wie z .
Es gibt einige elektrochemische Methoden zum Nachweis von V. Cholera bestand aus einer Reihe komplexer Schritte. Ein elektrochemisches V. Cholera Genosensor von Liew et al. [24] verwendeten eine elektrochemische Adsorptionsmethode, um die DNA-Sonde auf dem Kohlenstoff-SPE zu immobilisieren. Die lyophilisierten AuNPs-modifizierten mehrschichtigen PSA-Partikel mit Polyelektrolyten bildeten ein Biokonjugat mit Avidin, um als Reporter-Marker im Sandwich-DNA-Hybridisierungsassay zu fungieren. Die Zugabe von Sorbitolstabilisator war jedoch erforderlich, um die PSA-AuNPs-Avidin-Biokonjugate zu erhalten und die Betriebsdauer des DNA-Biosensors auf bis zu 30 Tage zu verlängern. Enzymatische elektrochemische V. Cholera Der DNA-Biosensor wurde kürzlich von Yu et al. [25], wobei die thiolierte anti-Fluorescein-konjugierte alkalische Phosphatase (anti-FCAP)-markierte DNA-Sonde durch Gold-Thiol-Chemie an die Gold-SPE gebunden wurde. Die Ziel-DNA wurde mit einem universellen Fluorescein markiert, um eine DNA-Hybridisierungserkennung zu ermöglichen, die über die enzymatische Umwandlung von &agr;-Naphthylphosphat in elektroaktives &agr;-Naphthol erreicht wird. Nichtsdestotrotz erforderte dieses Nachweisschema eine lange Assayzeit von ungefähr 95 min für die DNA-Hybridisierung, die Markierung der DNA-Hybride mit einem funktionellen Enzym, gefolgt von der Inkubation der Elektrode in einem elektroinaktiven &agr;-Naphthylphosphat-Substrat, bevor eine amperometrische Messung durchgeführt werden kann. Ein weiterer enzymatischer elektrochemischer DNA-Biosensor basierend auf Avidin-beschichtetem Kohlenstoff-SPE, konjugiert mit einer biotinylierten DNA-Sonde, wurde von Low und Teammitgliedern entwickelt [26]. Bei dieser Doppelhybridisierungsstrategie wurde auch eine Digoxigenin (DIG)-markierte Reportersonde verwendet, die die cDNA-Sequenz flankierte. Als elektrochemischer Marker wurde Meerrettichperoxidase-gebundenes Anti-DIG (anti-DIG-HRP) verwendet, das gleichzeitig die Oxidation von 3,3′,5,5′-Tetramethylbenzidin (TMB) mit der Reduktion von H . katalysieren konnte 2 O2 um einen Elektronentransfer auf die Elektrodenoberfläche für die elektrochemische Transduktion des DNA-Hybridisierungsereignisses zu ergeben. Ein einfaches DNA-Biosensor-Design basierend auf einer mit einer thiolierten DNA-Sonde immobilisierten goldbeschichteten Glaselektrode wurde von Patel et al. [22] zum schnellen Nachweis von V. Cholera , und Methylenblau wurde als DNA-Hybridisierungsindikator verwendet. Der lineare Nachweisbereich des Systems ist jedoch auf μM-Ebenen beschränkt, was seine Anwendung in klinischen Proben einschränkte.
Latex-Gold-Nanopartikel wurden zuvor als DNA-Hybridisierungsmarker über Avidin/Biotin-Bindung an die DNA-Sonde beim Nachweis von V verwendet. Cholera [24], Fischpathogen Aphanomyces invadans [27], E. coli [28] und unspezifische DNA-Hybridisierung [29], wobei die Latexkügelchen mit einer mehrschichtigen Polyelektrolytschicht beschichtet wurden, bevor die negativ geladenen Kolloide aus Goldnanopartikeln elektrostatisch daran befestigt wurden. Kawde und Wang [29] befestigten die PSA-Latexpartikel an einer DNA-Reportersonde, die als DNA-Hybridisierungsmarker verwendet werden sollte, indem Streptavidin-beschichtete Latexpartikel mit Biotin-beschichteten AuNPs beladen wurden. Kuanet al. [24, 27] und Liew et al. [24, 27] berichteten ebenfalls über dieselbe Avidin-Biotin-Bindungsmethode unter Verwendung von Gold-PSA-DNA-Sondenkonjugaten. Pinijsuwanet al. [28] berichteten über die Verwendung einer elektrostatischen Methode zur Beladung von PSA-Partikeln, die an eine DNA-Reportersonde gebunden waren, und die mit Polyelektrolyt goldbeschichteten PSA-Partikel wurden als Markierung für die Hybridisierung verwendet, die die DPV-Stromantwort verstärkte.
In dieser Studie berichten wir über einen anderen DNA-Immobilisierungsansatz unter Verwendung von Latex-Gold-Nanopartikeln als DNA-Sonden-Immobilisierungssubstrat, um ein hochempfindliches Nachweissystem für V zu entwickeln. Cholera DNA. Die Immobilisierung der DNA wurde mit einem sehr einfachen und schnellen Verfahren unter Verwendung von 1-Ethyl-3-(3-dimethylaminopropyl)carbodiimid-Hydrochlorid/N-Hydroxysuccinimid (EDC/NHS)-Chemie als Kopplungsreagenz zur Verbesserung der Immobilisierungseffizienz [30] auf dem carboxylierten Latex durchgeführt Partikeloberfläche. Der DNA-Hybridisierungsnachweis basierte auf dem Sandwich-Assay, der eine Hybridisierungsreaktion zwischen immobilisierter DNA-Sonde und Zielsequenz gefolgt von einer Signal/Reporter-Sonde beinhaltete. Anthrachinon-2-sulfonsäuremonohydrat-Natriumsalz (AQMS) wurde als elektrochemischer Marker verwendet, um das Hybridisierungsereignis zu überwachen. Die vorgeschlagenen Latexpartikel im Submikrometerbereich verbesserten die DNA-Sondenbindungskapazität, und die Empfindlichkeit des DNA-Biosensors wurde durch den Einbau der hochleitfähigen Goldnanopartikel (AuNPs) verbessert. Der DNA-Biosensor zeigte eine außergewöhnliche Empfindlichkeit beim Nachweis von V. Cholera cDNA und extrem niedrige Nachweisgrenze bei zeptomolaren Konzentrationen im Vergleich zur bisher berichteten Avidin-Biotin-Technologie [24, 26].
Methoden
Chemikalien und Reagenzien
Styrol (St) und Acrylsäure (AA) wurden von Fluka bezogen. HAuCl4 ·3H2 O, Trinatriumcitrat-Dehydrat, Natriumdodecylsulfat (SDS), 1-Ethyl-3-(3-dimethylaminopropyl)carbodiimid-Hydrochlorid (EDC) und N-Hydroxysuccinimid (NHS) wurden von Sigma-Aldrich bezogen. Ammoniumpersulfat (APS), Bromwasserstoffsäure und Brom wurden von Riedel-De Haën, Ajax Finechem bzw. Panreac geliefert. Alle chemischen Lösungen wurden mit entionisiertem Wasser hergestellt. Sowohl synthetische 30-Basen-Ziel- als auch Mismatch-Oligonukleotide wurden von der Bio Service Unit (NSTDA) bezogen. Nicht-komplementäre DNA (ncDNA) und Signalsonde stammten von Sigma und die 5'-Amino-modifizierte Einfangsonde stammte von Bioneer. Die Einfangsonde wurde in 0,05 M Kaliumphosphatpuffer (pH 7) hergestellt, während Ziel-DNA, fehlgepaartes Ziel, Reportersonde und nicht-komplementäre DNA-Lösungen in Natriumphosphatpuffer (0,05 M, pH 7) hergestellt wurden. Die in der vorliegenden Studie verwendeten Oligonukleotidsequenzen sind in Tabelle 1 gezeigt.
Vorrichtung
Die elektroanalytische Messung erfolgte mit einem Potentiostat/Galvanostat (Autolab PGSTAT12, Metrohm) ausgestattet mit GPES (4.0.007) Software. Die voltammetrischen Experimente wurden mit einem konventionellen Drei-Elektroden-System, bestehend aus einer aus Kohlenstoffpaste siebgedruckten Arbeitselektrode (SPE) (Quasense, Bangkok, Thailand), einer Ag/AgCl-Referenzelektrode (3 M KCl) und einem Platinstab ( 2 mm Durchmesser) Gegenelektrode. Für alle elektrochemischen Untersuchungen wurde die Differential-Pulsvoltammetrie-Technik (DPV) bei 0,02 V Stufenpotential und 0,5 V/s Scanrate von 0,0 bis +1,0 V in 4,5 ml Messpuffer (0,05 M Kaliumphosphatpuffer) bei pH 7 und verwendet Umgebungstemperatur. Alle in dieser Studie gemessenen Potentiale wurden auf eine Ag/AgCl-Elektrode bezogen und homogene Lösungen wurden unter Verwendung eines Beschallungsbades (Elma S30H) hergestellt. Ein Rasterelektronenmikroskop (REM, LEO 1450VP) wurde verwendet, um die Größe und Verteilung der Latexkugeln zu bestimmen.
Methoden
Synthese kolloidaler Gold-Nanopartikel
Kolloidale AuNPs wurden durch Natriumcitrat-Reduktion nach der Turkevich-Methode hergestellt [31]. Kurz gesagt, etwa 10 ml 5 mM HAuCl4 ·3H2 O wurde in 180 ml entionisiertem Wasser gelöst und unter kontinuierlichen Rührbedingungen auf einer kombinierten Heizplatten-Magnetrührer-Vorrichtung gekocht. Zehn Milliliter 0,5 % (w /v ) Trinatriumcitrat wurde dann in die kochende Lösung gegeben, und es wurde beobachtet, dass sich die Farbe der Lösung allmählich von blassrot zu rubinrot änderte.
Vorbereitung von Latexpartikeln
Latexteilchen wurden durch seifenfreie Emulsionscopolymerisationsreaktion hergestellt, wie von Polpanich et al. [23] mit einigen Modifikationen. Kurz gesagt, etwa 190 g entionisiertes Wasser wurden mit Stickstoffgas in einem Dreihalskolben, der in ein Wasserbad eingetaucht war, für ~1 h unter Rühren bei 350 U/min gespült. Zwanzig Gramm St und 0,5 g AA wurden dann zugegeben und die Temperatur wurde bei 70ºC gehalten. Danach wurden etwa 0,2 g APS in 10 ml entionisiertes Wasser gegeben, gefolgt vom Eingießen in die Formulierung im Dreihalskolben, um die radikalische Polymerisationsreaktion zu starten, und der Polymerisationsprozess wurde 8 h fortgesetzt. Die so synthetisierten Carboxyllatex-Kugeln wurden durch Zentrifugation mit entionisiertem Wasser zweimal bei 13.000 U/min für 20 Minuten geerntet [23, 27, 28] und in entionisiertem Wasser bei Raumtemperatur (25 °C) bis zur Verwendung resuspendiert. Die Morphologie und die durchschnittliche Größe der PSA-Latexpartikel wurden durch Rasterelektronenmikroskopie (REM) bestimmt.
Modifikation der SPE-Oberfläche und DNA-Sonden-Immobilisierung
Vor der Oberflächenmodifizierung wurde das Kohlenstoff-SPE gründlich mit entionisiertem Wasser gespült und dann mit der PSA-Suspension mit 3 mg/ml tropfenbeschichtet und bei Umgebungsbedingungen an der Luft trocknen gelassen, gefolgt von einem Tropfenguss mit 5 mg/ml kolloidalen AuNPs . Die elektrochemischen Eigenschaften von Kohlenstoff-SPE vor und nach der Modifizierung mit Latexpartikeln und AuNPs wurden mit der CV-Methode untersucht. Das Latex-AuNPs-modifizierte Kohlenstoff-SPE (PSA-AuNPs-SPE) wurde dann mit entionisiertem Wasser gespült und in 0,1 M Kaliumphosphatpuffer (pH 5) getaucht, der Carbodiimid-Vernetzungsreagenzien enthielt, dh 0,002 M EDC und 0,005 M NHS für 2 h [32], bevor es über Nacht in 0,05 M Kaliumphosphatpuffer (pH 7) mit 5 μM Fangsonde eingeweicht wird. Danach wurde das DNA-modifizierte PSA-AuNPs-SPE (DNA-PSA-AuNPs-SPE) gründlich mit Kaliumphosphatpuffer (0,05 M, pH 7) gewaschen, um die physikalisch adsorbierte Sonde zu entfernen. Die DNA-Elektrode wurde für 1 h in 0,05 M Natriumphosphatpuffer mit linearer Ziel-DNA (1 μM) und AQMS-DNA-Hybridisierungsmarker (5 mM) zur partiellen Hybridisierung getaucht und später in 0,05 M Natriumphosphatpuffer (pH .) getaucht 7) konditioniert mit 1 &mgr;M Reportersonde und 5 mM AQMS für eine weitere Stunde für den vollständigen DNA-Hybridisierungsprozess. Schließlich wurde die DNA-Elektrode mit Kaliumphosphatpuffer (0,05 M, pH 7) gespült, um nicht hybridisierte Oligonukleotidsequenzen zu entfernen. Die elektrochemische Reaktion jeder an die SPE gebundenen verlängerten Substanz wurde mit der DPV-Methode untersucht. Abbildung 1 zeigt das schrittweise Vorgehen zur Entwicklung von V. Cholera DNA-Biosensor basierend auf kolloidalem PSA-AuNPs-Feststoffträger.
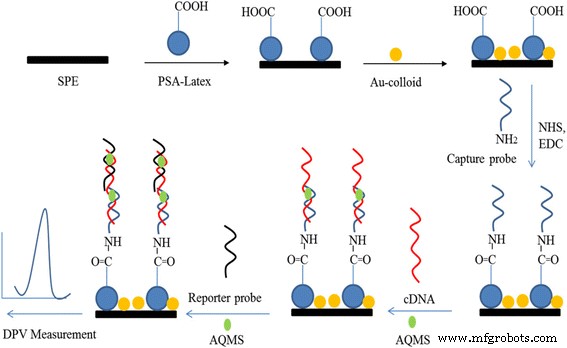
Schematische Darstellung des schrittweisen Herstellungsverfahrens des DNA-Biosensors
Optimierung der Hybridisierung synthetischer Oligonukleotide
Der Einfluss verschiedener Parameter wie DNA-Sonden- und AQMS-Konzentrationen, pH, Ionenstärke und Pufferkapazität auf die Hybridisierungsreaktion der immobilisierten DNA-Sonde mit Signalsonde und Ziel-DNA wurde vor der Bestimmung des dynamischen linearen Bereichs der DNA untersucht Biosensor. Darüber hinaus wurden auch die Dauer der Immobilisierung der Einfangsonde und der DNA-Hybridisierung sowie die Lebensdauer und Regeneration des Biosensors vor dem entwickelten V. Cholera Der DNA-Biosensor war bereit für die Anwendung im Spike-and-Recovery-Experiment. Die Einfangsonden- und AQMS-Beladungen wurden durch Variation ihrer Konzentrationen von 1 bis 6 μM bzw. 0,1–5 mM optimiert, während die Konzentration der Ziel-DNA und der Reportersonde bei 5 μM in 0,05 M Natriumphosphatpuffer (pH 7,0) gehalten wurde. pH-Effekt- und Pufferkonzentrationsstudien wurden durchgeführt, indem der pH und die Konzentration des Natriumphosphatpuffers von pH 5,5 auf 8,0 bzw. 0,001 auf 1,000 M geändert wurden. Die Anwesenheit verschiedener Kationen in der DNA-Hybridisierungsantwort des elektrochemischen DNA-Biosensors wurde durch Zugabe von Na + . durchgeführt , K + , Ca 2+ , und Fe 3+ Ionen mit 1,0 M in den DNA-Hybridisierungspuffer, der 1 mM AQMS und 5 &mgr;M cDNA und eine Detektionssonde von pH 7,0 enthält. Die Wirkung der Ionenstärke wurde untersucht, indem die NaCl-Konzentration über den Bereich von 0,1–3,0 M bei pH 7,0 variiert wurde. Der dynamische Bereich des DNA-Biosensors wurde dann in 1.0 × 10 –21 . bestimmt bis 1,0 × 10 –8 MV. Cholera cDNA unter Verwendung einer konstanten Signalsondenkonzentration bei 5 &mgr;M und pH 7,0. Die Dauer der Immobilisierung der DNA-Sonde wurde bestimmt, indem die DNA-Elektrode zwischen 1 und 13 h in 5 μM Einfangsondenlösung (pH 7,0) getaucht wurde, und die DPV-Antwort wurde alle 1–2 h gemessen. Inzwischen wurde die DNA-Hybridisierungszeit bestimmt, indem man die DNA-Hybridisierungsreaktion zwischen 15 und 180 Minuten ablaufen ließ, und die DNA-Biosensor-Antwort wurde alle 15–30 Minuten aufgezeichnet. Die Haltbarkeit des DNA-Biosensors wurde durch periodisches Messen der DNA-Biosensor-Antwort auf den Nachweis von 5 µM V bestimmt. Cholera cDNA für 120 Tage. Die Analyse wurde in fünf Wiederholungen unter Verwendung einer neuen DNA-Elektrode für jeden Sandwich-Hybridisierungsassay durchgeführt. Die Regeneration der DNA-Elektrode wurde mit 0,1 M NaOH-Lösung für 4 min durchgeführt, und die Rehybridisierung des DNA-Biosensors (60 min) wurde unter Verwendung einer Rehybridisierungslösung mit 5 μM cDNA und Detektionssonde und 1 mM AQMS bei 2,0 M Ionenstärke durchgeführt in 0,05 M Kaliumphosphatpuffer (pH 7,0). Das Regenerationsexperiment wurde in sechs Wiederholungen durchgeführt.
V. Cholera Quantifizierung mit einem auf PSA-AuNPs basierenden elektrochemischen DNA-Biosensor
Verschiedene V. Cholera Bakterienstämme, nämlich J2126-I, J2126-II, J3324-I, J3324-II, J3330-I, J3330-II, CDHI 5294-II und UVC1324 einschließlich Citrobacter freundii (CF-I) und Citrobacter freundii (CF-II) wurden vom Microbiology Laboratory, Faculty of Applied Sciences, AIMST University, Kedah erhalten. Anschließend wurde mit QIAGEN Genomic-tip 500/G eine genomische DNA-Extraktion über diese Bakterien gemäß dem Protokoll des Herstellers durchgeführt. Die extrahierte DNA wurde dann unter Verwendung von Natriumphosphatpuffer (0,05 M, pH 7,0) 100-fach verdünnt. Etwa 300 ml der extrahierten DNA, die 2,0 M NaCl und 1 mM AQMS enthielt, wurden 15 Minuten lang beschallt, um die DNA-Brüche freizugeben. Dann wurde die immobilisierte DNA-Sonde 1 h lang eingeweicht, um den DNA-Hybridisierungsprozess ablaufen zu lassen, und vorsichtig mit 0,05 M Kaliumphosphatpuffer (pH 7,0) gewaschen, um die ungebundene DNA zu entfernen. Die Bewertung der DNA-Biosensorantwort basierend auf dem DPV-Spitzenstrom wurde gemessen und mit der Stromantwort verglichen, die von der DNA-Elektrode ohne Reaktion mit cDNA als Kontrollsignal erzeugt wurde. Jedes Experiment wurde dreifach unter den gleichen experimentellen Bedingungen durchgeführt. Gemeinsames t Der Test wurde verwendet, um einen signifikanten Unterschied zwischen der Reaktion des DNA-Biosensors und der Reaktion der Kontrolle zu bestimmen. Die Wiederherstellung von V. Cholera J3324 und V. Cholera UVC1324-DNAs bei 1.0 × 10 −4 μg μL −1 , 1.0 × 10 −5 μg μL −1 , und 1,0 × 10 −6 μg μL −1 in den Hybridisierungspuffer gespiked wurde dann unter Verwendung des vorgeschlagenen elektrochemischen DNA-Biosensors auf Basis von PSA-AuNPs durchgeführt.
Ergebnisse und Diskussion
Morphologie von Latexpartikeln und elektrochemisches Verhalten von Latex-Gold-Nanopartikel-modifizierter SPE.
Abbildung 2 zeigt die REM-Aufnahme der carboxylierten Latexkugeln mit einer durchschnittlichen Partikelgröße von 186,1 ± 4,6 nm. Die gleichmäßige Größenverteilung der PSA-Kugeln ermöglichte eine homogene Immobilisierung von DNA-Molekülen auf der Latexoberfläche, um die Reproduzierbarkeitsreaktion des DNA-Biosensors zu verbessern. Ein Rasterelektronenmikroskop (REM, LEO 1450VP) wurde verwendet, um die Größe und Verteilung der Latexkugeln zu bestimmen.

REM-Aufnahme der so synthetisierten PSA-Latexkugeln bei 10.000-facher Vergrößerung
Die elektrodynamischen Ergebnisse der modifizierten SPE sind in Tabelle 2 aufgeführt. Die Peakpotentialtrennung (ΔEp) zeigt die Elektronentransferkinetik des Systems. Das PSA-modifizierte SPE (PSA-SPE) zeigte den höchsten ΔEp-Wert aufgrund des langsamen Ladungstransferprozesses in der kolloidalen Copolymerpartikelschicht, der das System in einen quasi-reversiblen Zustand bewegte. Wenn die AuNPs jedoch auf die PSA-SPE geladen wurden, bedeutet die Abnahme von ΔEp eine Verbesserung der Elektronentransferrate an der Elektrodenoberfläche.
Abbildung 3 zeigt die differentiellen Pulsvoltammogramme der AQMS-Oxidationsreaktion auf dem latexmodifizierten SPE und die sequentielle Hybridisierungsreaktion des V. Cholera DNA-Biosensor. Signifikante DPV-Spitzenstromunterschiede wurden zwischen Elektroden beobachtet, die nur latexmodifizierte Mikrokügelchen enthielten und ohne immobilisierte Einfang-DNA (Experimente (a) und (b)) und modifiziert mit immobilisierten Einfang-DNA-Sonden in Gegenwart von cDNA und berichteter Sonde (Experiment (g )). Dies weist darauf hin, dass die aminierten DNA-Einfangsonden erfolgreich auf den beschichteten carboxylierten PSA-Latexkugeln über das EDC/NHS-Kopplungsprotokoll immobilisiert wurden [33]. Experiment (g) zeigt auch eine viel höhere DPV-Reaktion im Vergleich zu DNA-Elektroden in Gegenwart von ncDNA und berichteter Reportersonde (Experiment (d)), in fehlgepaarter DNA und berichteter Reportersonde (Experiment (e)) und Ziel-cDNA mit keine gemeldete Sonde (Experiment (f)). Dies ist auf die vollständige Hybridisierung der Ziel-DNA mit Einfang- und Reportersonden durch Sandwich-Hybridisierungsreaktion auf der DNA-Biosensoroberfläche zurückzuführen, wie in Experiment (g) gezeigt. Dies zeigt auch, dass die Verwendung der beschriebenen Sonde das Signal der DNA-Hybridisierung verstärken könnte. Trotzdem ist der DPV-Strom, der aus der Hybridisierung resultiert, die in Gegenwart von Ziel-DNA ohne Einbau einer berichteten Sonde (Experiment (f)) beobachtet wurde, immer noch höher als die DPV-Stromsignale, die für nicht hybridisierte DNA beobachtet wurden (Experimente (c), (d) und (e)).
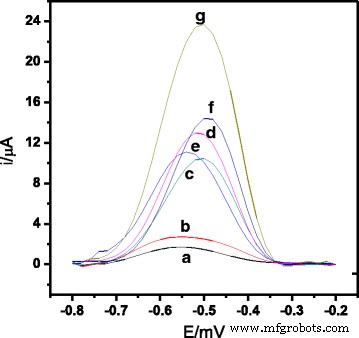
Differenzielles Pulsvoltammogramm-Signal vom AQMS der Elektroden (a ) PSA-SPE, (b ) PSA-AuNPs-SPE, (c ) Fangsonde-PSA-AuNPs-SPE und in Gegenwart von (d ) ncDNA und Reportersonde, (e ) Fehlpaarung von DNA und Reportersonde, (f ) cDNA allein und (g ) cDNA und Reportersonde
Auswirkung der DNA-Sondenbeladung und der AQMS-Konzentration
Die Wirkung der DNA-Sondenkonzentration auf die DNA-Hybridisierungsreaktion wurde durch die elektrochemische AQMS-Oxidationsreaktion beobachtet. Abbildung 4a zeigt, dass die DNA-Biosensor-Antwort allmählich mit der Zunahme der DNA-Sondenmenge, die auf dem PSA-AuNPs-SPE immobilisiert ist, von 1 auf 4 μM zunahm. Dies wurde der zunehmenden Menge an elektroaktiver AQMS zugeschrieben, die in die doppelsträngige DNA (dsDNA) interkaliert ist, um einen Elektronentransfer durch die immobilisierte DNA-Helix zu bewirken. Es wurde beobachtet, dass die DPV-Antwort des DNA-Biosensors zwischen 4 und 6 μM DNA-Sonde fast ein Plateau erreicht, was darauf hindeutet, dass eine optimale DNA-Sondenbeladung auf der Elektrodenoberfläche erreicht wurde [34]. Daher wurde in den nachfolgenden Experimenten eine 4 &mgr;M Einfangsonde als optimale DNA-Sondenbeladung ausgewählt. Auch die Konzentration des AQMS-Markers wurde im Messelektrolyt zwischen 0,1 und 5,0 mM optimiert, und die Konzentration von AQMS bei 1 mM erwies sich als ausreichend für eine optimale DNA-Interkalationsreaktion (Abb. 4b).
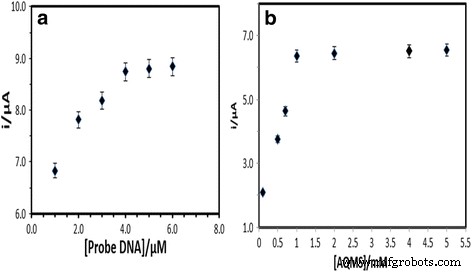
Die Wirkung der Fangsonde (a ) und AQMS-Konzentrationen (b ) zur DNA-Biosensor-Antwort, durchgeführt mit 5 μM cDNA und Signalsonde in 0,05 M Natriumphosphatpuffer (pH 7,0)
Auswirkung von pH, Ionenstärke und Pufferkapazität
Die Geschwindigkeit der DNA-Hybridisierungsreaktion hängt stark vom pH-Wert der Lösung ab. Wie in Fig. 5a zu sehen ist, verringerte in einer saureren Umgebung die Protonierung des Phosphodieter-Rückgrats der DNA die Löslichkeit des DNA-Moleküls, was schließlich die DNA-Hybridisierungsreaktionsgeschwindigkeit verringerte. Im basischen Medium hingegen brach es die schwachen Wasserstoffbrückenbindungen, die die DNA-Basenpaare zusammenhielten. Die optimale DNA-Hybridisierungsreaktion war im neutralen Zustand günstiger, wodurch mehr Einfangsonden zur Hybridisierung mit Ziel-DNA gefördert und anschließend die Interkalation von AQMS-Redoxsonden ermöglicht wurde, um die DNA-Hybridisierungserkennung erfolgreich zu machen. Somit wurden 0,05 M Natriumphosphatpuffer bei pH 7,0 als DNA-Hybridisierungsmedium für nachfolgende DNA-Biosensor-Studien verwendet. Positiv geladene Ionen wie Ca 2+ , Na + , K + , und Fe 3+ Ionen können mit der negativ geladenen Phosphodiesterkette der DNA wechselwirken. Diese ionische Reaktion neutralisiert die Ladung des DNA-Moleküls, wodurch die sterische Abstoßung zwischen DNA-Molekülen verringert wird, um die DNA-Hybridisierungsreaktion zu erleichtern. 5b zeigt die Wirkung einiger Kationen auf die DNA-Hybridisierungsantwort. Es wurde festgestellt, dass die DNA-Hybridisierungsreaktion in Gegenwart von positiv geladenen Ionen in der Größenordnung von Na + . zunimmt> K + > Fe 3+ > Ca 2+ . Sowohl Ca 2+ und Fe 3+ Es wurde festgestellt, dass Ionen die DNA-Hybridisierungsreaktion aufgrund der ionischen Wechselwirkungen von Ca 2+ . erheblich reduzieren und Fe 3+ Ionen mit Phosphationen aus der Pufferlösung, was zur Bildung unlöslicher Phosphatverbindungen führte. Dies hat den Ionengehalt des Mediums verringert und dadurch die elektrostatische Abstoßung zwischen DNA-Molekülen erhöht. Der höchste DNA-Hybridisierungsstrom wurde in Gegenwart von Na + . erhalten -Ion aufgrund seiner geringeren Größe und stärkeren Affinität zum DNA-Zucker-Phosphat-Rückgrat im Vergleich zu K + -Ionen, um die sterische Hinderung und elektrostatische Abstoßung zwischen negativ geladenen Phosphatgruppen von DNAs zu überwinden.
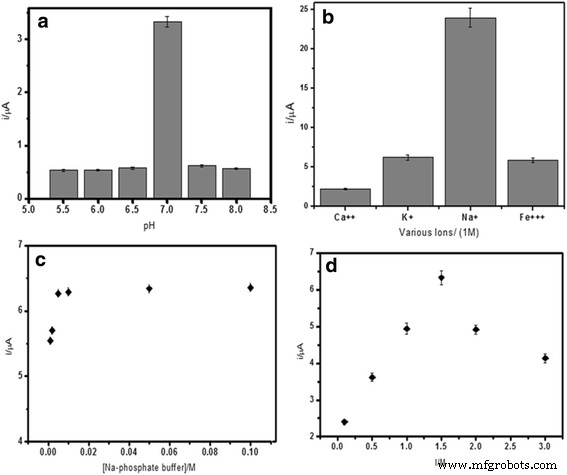
Die Wirkung des pH-Wertes (a ) verschiedene Kationen (b ), Pufferkonzentration (c ) und Ionenstärke (d ) auf die DNA-Hybridisierungsantwort des elektrochemischen V. Cholera DNA-Biosensor. Die Hybridisierung wurde mit 5 μM cDNA und Reportersonde durchgeführt, gefolgt von Interkalation mit 1 mM AQMS
Außerdem würde die Ionenstärke der Lösung auch die Reaktion des DNA-Biosensors beeinflussen. Fig. 5c, d zeigen die optimale Pufferkapazität, und die Ionenstärke wurde unter Verwendung von 0,05 M Natriumphosphatpuffer mit einem auf pH 7,0 bzw. 2,0 M NaCl fixierten pH-Wert erreicht. In diesem Zustand begünstigte es die DNA-Hybridisierungsreaktion in höchstem Maße; daher wurde eine hohe DPV-Antwort erhalten. Bei optimaler Pufferkapazität und Ionenstärke der Lösung nimmt die elektrostatische Abstoßung zwischen DNA-Molekülen ab und verbessert so die DNA-Hybridisierungsreaktion. Wenn dagegen ein zu niedriger oder zu hoher Ionengehalt verwendet wurde, dominierten sterische Hinderung und elektrostatische Abstoßung und schränkten die Hybridisierung von DNA-Molekülen ein.
Einrichtung von V. Cholera DNA-Biosensor-Kalibrierungskurve
Aus dem in Fig. 6a gezeigten Ergebnis geht hervor, dass die Reaktion des DNA-Biosensors proportional mit steigender cDNA-Konzentration von 1,0 × 10 –21 . angestiegen ist bis 1,0 × 10 –8 M (R 2 =0,99) mit einer Nachweisgrenze von 1,0 × 10 −21 M. Die Nachweisgrenze wurde basierend auf der dreifachen Standardabweichung der Biosensor-Antwort bei der Reaktionskurve berechnet, die sich der Nachweisgrenze annähert, geteilt durch die lineare Kalibrierungssteigung. Der breite lineare Nachweisbereich des DNA-Biosensors beruht auf den hoch monodispersen und kugelförmigen PSA-Latexpartikeln im Submikrometerbereich, die als Trägermatrix für die DNA-Immobilisierung verwendet werden. Die acrylsäurereiche Schicht auf der Oberfläche der Latexpartikel bot eine große Bindungsstelle zum Anheften von DNA-Einfangsonden, um eine maximal bedeckte Oberfläche durch die DNA-rezeptive Schicht zu erzeugen. Darüber hinaus verstärkte der Einbau von AuNPs auf dem PSA-modifizierten SPE das analytische Signal der DNA-Hybridisierungsantwort weiter, was zu einer hohen Empfindlichkeit des DNA-Biosensors führte (Abb. 6b).
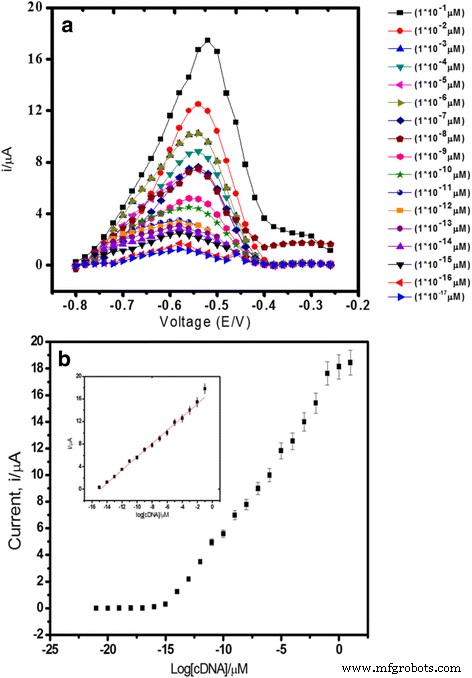
Differential pulse voltammograms (a ) and DNA biosensor linear range (b ) obtained using various cDNA concentrations from 1.0 × 10 −15 to 1.0 × 10 −1 μM V. cholerae target DNA and 5 μM signal probe
DNA Probe Immobilisation and Hybridization Times
It took about 8 h for the capture probe to be immobilised on the PSA copolymer particles surface, as illustrated by the DNA biosensor response in Fig. 7a, which showed a current increment from 1.0 to 8.0 h of capture probe immobilisation time, after which no obvious change in the DPV current was observed. Longer immobilisation time resulted in a higher amount of DNA probes immobilised onto the latex. After 8.0 h of exposure to the DNA probes, the hydrophilic functional latex with reactive carboxyl groups at the surface was presumably fully attached with the DNA probes. DNA hybridisation time, on the other hand, is the rate limiting step, which determines the response time of the DNA biosensor. Based on the DNA biosensor response trend in Fig. 7b, the response time of the V. cholerae DNA biosensor developed in this study was estimated to be about 60 min for the dual hybridisation processes to complete.
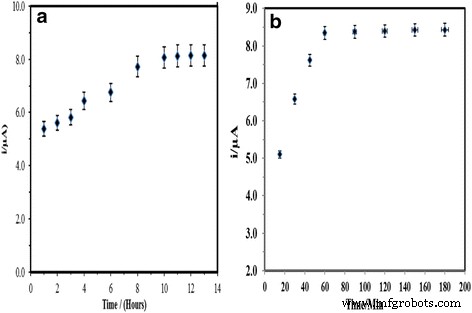
DNA probe immobilisation duration on the immobilised PSA latex colloidal particles (a ) and DNA hybridization duration of the DNA biosensor (b ) in 0.05 M potassium phosphate buffer at pH 7.0 containing 5 μM target DNA and reporter probe and 1 mM AQMS at 2.0 M ionic strength
Long-Term Stability and Regeneration of V.cholerae DNA Biosensor
Figure 8 shows the shelf life of the V. cholerae DNA biosensor. The DNA biosensor showed the highest response to the detection of 5 μM of V. cholerae cDNA for the first month of the experimental period. The electrochemical DNA biosensor was able to retain 95% of its initial DPV current after 58 days of storage period. The DNA hybridisation response was then gradually decreased to about 75% of its original response on the 75th day and exhibited 40% of its initial performance on the 100th operational day. The bioactivity of the immobilised capture probe was finally declined to 30% after 3 months of storage period. The reproducibility of each calibration point, which was repeated on five replicate DNA electrodes, gave satisfactory relative standard deviation (RSD) between 2.4 and 4.5% (n =5).
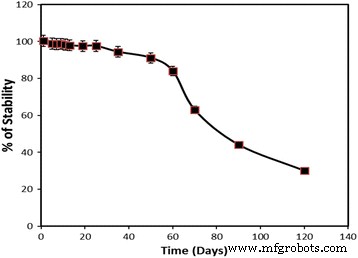
The life span profile of the fabricated V. cholerae DNA sensing electrode. The electrode was stored in 0.05 M potassium phosphate buffer (pH 7.0) at 4 °C after every DPV measurement was taken
Regeneration of biosensor indicates whether the biosensor is reusable for a series of consecutive analyses. The regeneration method used in this study was conducted based on previously reported protocol in other studies [11, 21] with slight modifications. In this study, 0.1 M of NaOH solution was used as the regeneration solution to break the hydrogen bonds between base pairs of hybridised dsDNA. With the result from Fig. 9, it is notable that the DNA biosensor response declined significantly after incubation in 0.1 M of NaOH and the percentage of the DNA biosensor response reduced from 35.1 to 5.2% relative to the DNA biosensor initial response after incubation in 0.1 M of NaOH solution from 30 to 240 s. The DNA biosensor response decreased with the increasing incubation time signifies the hydrogen bonds between hybridised dsDNA were broken up by the alkaline regeneration solution. However, rehybridisation of the DNA biosensor was able to attain almost 100% of its initial response for a consecutive six DNA analyses with a reversibility RSD of 5%.
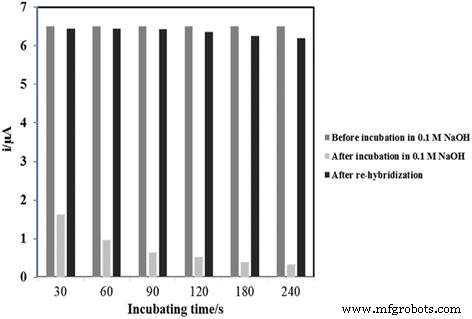
Repeatability of V. cholerae DNA biosensor using 0.1 M NaOH regeneration solution and rehybridization solution containing 5 μM cDNA and detection probe and 1 mM AQMS at 2.0 M ionic strength in 0.05 M potassium phosphate buffer (pH 7.0)
Determination of V. cholerae Bacteria with the Developed DNA Biosensor
The optimised DNA biosensor has been applied to quantify the V. cholerae DNA extracted from various V. cholerae bacterial strains. Table 3 presents the results acquired from DNA tests carried out with the hybridisation medium spiked with different strains of V. cholerae DNAs and other bacterial species at a concentration within the calibration range of the DNA biosensor. The DNA biosensor showed superior selectivity towards V. cholerae J3324–I, V. cholerae J3324–II, and V. cholerae UVC1324 with high DPV current response and low current signals were obtained for the evaluation of both Citrobacter freundii (CF-I) and Citrobacter freundii (CF-II).
Recovery of V. cholerae J3324 and V. cholerae UVC1324 DNAs at three different concentrations spiked into the hybridisation buffer demonstrated 91.4 ± 2.2% to 108.9 ± 4.8% (n =3) of recoveries percentage (Table 4). This result suggests that the proposed PSA-AuNPs-based electrochemical DNA biosensor could be adopted for highly reliable and accurate detection of V. cholerae DNA in environmental and clinical samples.
Performance Comparison with Other Reported V. cholerae DNA Biosensors
Based on the data summarised in Table 5, the proposed electrochemical DNA biosensor based on PSA-AuNPs immobilisation material shows an exceptional broad linear quantification range compared to other planar two-dimensional electrodes as the DNA supporters. This clearly demonstrates the advantage of the micro-sized latex particles where the polymeric PSA is capable to intensify the probe binding capacity with a simple loading method via the classical EDC/NHS coupling compared to avidin-biotin technology [24, 26] and ultra-low detection limit in zeptomolar range with reasonable assay time.
Conclusions
This study reports the development of an electrochemical DNA biosensor for the detection of one of the most devastating high-risk V. cholerae pathogens. The PSA-AuNPs-modified DNA biosensor can be used for direct detection of DNA of interest from the extracted DNA without the need of amplification reaction via conventional PCR method, which is commonly used in those previously reported V. cholerae DNA biosensors. In addition, no further dilution of the extracted DNA is needed as the high-capacity AuNPs-doped latex microspheres-based DNA biosensor is highly sensitive for the quantitation of DNA at extremely low level in sub zeptomolar range. Therefore, the electrochemical DNA biosensor is greatly suitable as a surveillance and diagnostic tool to control the epidemic of the fatal intestinal infection.
Nanomaterialien
- Das Potenzial für die Integration visueller Daten in das IoT
- Demonstration eines flexiblen Graphen-basierten Biosensors für den empfindlichen und schnellen Nachweis von Eierstockkrebszellen
- Der verbesserte photoelektrochemische Nachweis von Harnsäure auf Au-Nanopartikel-modifizierten Glaskohlenstoffelektroden
- Ein hochempfindlicher elektrochemischer DNA-Biosensor aus Acryl-Gold-Nanokomposit zur Bestimmung des Geschlechts von Arowana-Fischen
- Monodisperse Kohlenstoff-Nanosphären mit hierarchischer poröser Struktur als Elektrodenmaterial für Superkondensatoren
- Erhöhte Stabilität magnetischer Goldnanopartikel mit Poly(4-styrolsulfonsäure-co-maleinsäure):Maßgeschneiderte optische Eigenschaften für die Proteindetektion
- Kontrollierte Synthese von BaYF5:Er3+, Yb3+ mit unterschiedlicher Morphologie zur Verbesserung der Aufkonversionslumineszenz
- Ein fluoreszierender Aptasensor auf Graphenoxid-Basis für die Einschalterkennung von CCRF-CEM
- Elektrisch leitfähiger TPU-Nanofaserverbundstoff mit hoher Dehnbarkeit für flexiblen Dehnungssensor
- Impossible Objects arbeitet mit BASF für den 3D-Verbunddruck zusammen



