Gint4.T-modifizierte DNA-Tetraeder, die mit Doxorubicin beladen sind, hemmen die Proliferation von Gliomzellen, indem sie auf PDGFRβ abzielen
Zusammenfassung
Gliom ist aufgrund seines invasiven Wachstums einer der tödlichsten intrinsischen Hirntumore. Die Wirkung der Gliom-Behandlung ist aufgrund des Vorhandenseins der Blut-Hirn-Schranke und der Blut-Tumor-Schranke und der unzureichenden zielgerichteten Arzneimittelwirkung gering. DNA-Tetraeder (TDN) weisen ein großes Potenzial für die Wirkstoffabgabe auf und könnten eine neue therapeutische Strategie für Gliome darstellen. In dieser Studie verwendeten wir TDN, um Doxorubicin (DOX) für die Gliomtherapie zu verabreichen. Gint4.T, ein Aptamer, das den Thrombozyten-Wachstumsfaktor-Rezeptor β auf Tumorzellen erkennen kann, wurde verwendet, um TDN (Apt-TDN) für eine gezielte Wirkstoffabgabe zu modifizieren. Die TDN wurden durch einstufige Synthese selbstorganisiert, die eine geringe Größe (10 nm) und eine negative Ladung aufwies. Der fetale Rinderserumtest zeigte seine Stabilität als Vehikel zur Arzneimittelabgabe. Apt-TDN könnte effektiv von U87MG-Zellen aufgenommen werden. Verglichen mit DOX und DOX@TDN (mit DOX beladenes TDN) zeigte das DOX@Apt-TDN (Gint4.T-modifiziertes TDN mit DOX beladen) eine frühere Apoptoserate, einen höheren Zellzyklusarrest und eine größere Zytotoxizität gegenüber U87MG-Zellen. Zusammenfassend zeigten unsere Ergebnisse, dass DOX@Apt-TDN eine neuartige Therapie mit vielversprechender klinischer Anwendung für Gliompatienten darstellt.
Einführung
Gliom, ein Tumor, der vom Neuroepithel abstammt, ist die häufigste intrakranielle Malignität. Fast 1/3 aller Hirntumore sind Gliome und etwa 4/5 der primären malignen Hirntumore sind Gliome [1, 2, 3, 4]. Derzeit ist die wirksamste Behandlung von Gliomen die chirurgische Resektion und die gleichzeitige postoperative Radiochemotherapie, aber leider bleibt die Prognose für die Patienten schlecht. Herkömmliche Chemotherapien für Gliome zeigen kein gutes Ergebnis aufgrund schlechter Tumor-Targeting, Komplikationen aufgrund der Blut-Hirn-Schranke (BBB) und Blut-Tumor-Barriere (BTB) und unzureichender Wirkstoff-Targeting. Die BBB ist der wichtigste Faktor, der die Abgabe fast aller Makromoleküle (einschließlich Medikamente und Gene) an das Hirnparenchym und deren ordnungsgemäße Funktion verhindert. Medikamente, die die BHS nicht passieren können, müssen in ausreichend hohen Dosen verabreicht werden, um eine wirksame Behandlungskonzentration im interessierenden Bereich zu erreichen. Überschüssige Arzneimittel können jedoch schwere systemische Nebenwirkungen und eine unerwünschte Arzneimittelakkumulation in nicht betroffenen Geweben verursachen. Darüber hinaus haben bestehende konventionelle Anti-Gliom-Medikamente unzureichende Targeting-Fähigkeiten [3, 4]. Nanopartikel haben sich als das vielversprechendste Werkzeug zum Wirkstofftransport herausgestellt. Aufgrund ihres Größenvorteils können Nanopartikel die BHS überwinden und eine antitumorale Wirkung entfalten. Kafaet al. [5] entwarfen chemisch funktionalisierte mehrwandige Kohlenstoffnanoröhren (f-MWNTs), die auf ANG abzielten, und bestätigten ihre Fähigkeit, die BBB durch In-vivo- und In-vitro-Experimente zu passieren. Diese Nanomaterialien können jedoch auf verschiedene Organe im ganzen Körper verteilt werden oder sogar in das zentrale Nervensystem (ZNS) gelangen, wo sie Neurotoxizität verursachen können [6].
DNA ist ein ideales Material für den Aufbau von Nanostrukturen, da ihr Aufbau durch Watson-Crick-Basenpaarung präzise gesteuert werden kann [7]. Bis heute wurde eine Reihe von zweidimensionalen (2D) und dreidimensionalen (3D) DNA-Nanostrukturen entworfen und demonstriert [8,9,10]. Tetraedrische DNA-Nanostrukturen (TDN) haben aufgrund ihrer Biokompatibilität, Stabilität, zahlreichen funktionalisierten Modifikationsstellen und ihrer geringen Immunogenität große Aufmerksamkeit auf sich gezogen [11,12,13]. Turberfieldet al. synthetisierten tetraedrische DNA-Nanostrukturen mit hoher Ausbeute unter Verwendung einer einstufigen Synthesemethode [14]. Walsh et al. fanden, dass ein DNA-Tetraeder, der eine Nukleinsäuresonde enthält, in Säugerzellen eindringen kann, ohne dass ein Transfektionsreagenz erforderlich ist [15]. Leeet al. zeigten, dass selbstorganisierte tetraedrische Nanopartikel für die gezielte siRNA-Lieferung in vivo verwendet werden können [16]. TDN haben ausgezeichnete Anwendungsperspektiven in der Molekulardiagnostik, dem molekularen Transport und der zielgerichteten Arzneimitteltherapie gezeigt. Sie werden auch häufig in der Erforschung von Tumoren in verschiedenen Organen wie Brustkrebs verwendet [17]. In ähnlicher Weise wurden TDN auch bei der Untersuchung von Erkrankungen des Nervensystems verwendet. Tianet al. [18] nutzten DNA-Tetraeder als Grundlage und modifizierten sie mit Angiopep-2 (ANG), um erfolgreich die Nanosonde ANG-TDN zu konstruieren, die auf das Lipoproteinrezeptor-verwandte Protein 1 (LRP-1) mit niedriger Dichte für eine gezielte Bildgebung abzielen könnte. Untersuchungen haben gezeigt, dass ANG-TDN die BBB überschreiten kann. Ma et al. [19] schlugen vor, dass tetraedrische DNA-Nanostrukturen ohne Transfektionsmittel in neuronale Stammzellen (NSCs) eindringen können, wo sie die Migration, Proliferation und Differenzierung von neuralen Stammzellen fördern[20] und ein großes Potenzial für die Reparatur und Regeneration von neuralem Gewebe haben . Daher haben wir die Möglichkeit des Einsatzes von TDN in der Gliomtherapie und der Verbesserung seiner Targeting-Fähigkeit untersucht.
Der Thrombozyten-Wachstumsfaktor-Rezeptor β (PDGFRβ) ist ein wichtiges Mitglied der Tyrosin-Proteinkinase-Familie, das an der zellulären Proliferation, Migration und Angiogenese beteiligt ist. Mehrere Studien haben gezeigt, dass PDGFβ aufgrund seiner Rolle bei der Angiogenese ein vielversprechendes Ziel für Antitumortherapien ist [21, 22]. Aptamere sind kurze, einzelsträngige DNA- oder RNA-Oligonukleotide, die durch die systematische Evolution von Liganden durch exponentielle Anreicherung (SELEX) hergestellt werden. Aptamere ähneln Antikörpern, die eine hohe Affinität und Spezifität gegenüber ihren Zielen aufweisen [23]. Aufgrund ihrer einzigartigen Eigenschaften spielen Aptamere eine wichtige Rolle bei der gezielten Abgabe von Chemotherapeutika, siRNAs und wirkstoffbeladenen Nanopartikeln. Gint4.T, ein RNA-Aptamer, das PDGFRβ spezifisch binden kann, ist ebenfalls ein PDGFRβ-spezifischer Antagonist [24]. Monacoet al. [25] schlugen vor, dass Gint4.T-Aptamere die Blut-Hirn-Schranke (BBB) überwinden und spezifisch PDGFRβ erkennen können. Gint4.T-konjugierte polymere Nanopartikel (PNPs) können leicht von Glioblastom (GBM)-Zellen aufgenommen werden. In dieser Studie berichten wir über ein neuartiges arzneimittelbeladenes System, das das Gint4.T-Aptamer und TDN kombiniert. Das mit Gint4.T modifizierte TDN (Apt-TDN), das mit DOX (DOX@Apt-TDN) beladen war, zeigte eine verbesserte spezifische zelluläre Aufnahme und Zytotoxizität gegenüber U87MG-Zellen.
Methoden
Materialien
Alle DNA-Oligonukleotide und 2’F-Py-RNA-Oligonukleotide wurden von Sangon Biotech (Shanghai, China) bezogen und alle Oligonukleotidsequenzen sind in Tabelle 1 aufgeführt. GelRed DNA-Gelfärbelösung wurde von Sangon Biotech bezogen. Sowohl fötales Rinderserum (FBS) als auch Dulbeccos modifiziertes Eagle-Medium (DMEM) wurden von Thermo Fisher (New York, USA) bezogen. Doxorubicin (DOX) wurde von Mengbio Technology (Chongqing, China) bezogen. U87MG-Zellen wurden von der Zellbibliothek der Shanghai Life Academy of Sciences (Shanghai, China) erworben. DAPI wurde von Zhongshan Golden Bridge Biotechnology (Beijing, China) gekauft.
Herstellung von DNA-Nanostrukturen
Zum Aufbau des DNA-Tetraeders (Tabelle 1) wurden 2 µl jedes Oligonukleotids (S1, S2, S3 und S4) zu 42 µl TM-Puffer (10 µm Tris-HCl, 5 µm MgCl2 , pH =8). Die DNA-Lösung wurde dann 5 Minuten lang auf 95 °C erhitzt und anschließend 2 Minuten lang auf 4 °C abgekühlt, wobei ein Bio-Rad-PCR-Gerät (Kalifornien, USA) verwendet wurde [26, 27]. Die Endkonzentration von TDN betrug 2 µM. TDN’ wurde auf die gleiche Weise hergestellt, außer dass S1 durch S1’ ersetzt wurde. Um das Apt-TDN zu synthetisieren, wurde das Gint4.T-Aptamer in einem gleichen Molverhältnis zu TDN gegeben und das Gemisch wurde 60 Minuten bei 37 °C inkubiert. Vor der Synthese wurde das Aptamer einem kurzen Denaturierungs-Renaturierungsschritt unterzogen (85°C für 5 min, schnelles Abkühlen über 2 min und anschließendes Erwärmen auf 37°C über 10 min) [25].
Agarose-Gelelektrophorese
Ein Agarosegel (3%) wurde in 0,5 × TEB-Puffer bei 100 V für 30 min laufen gelassen. Die Temperatur des Elektrophoresegeräts wurde bei 0°C gehalten, indem das Gerät in ein Eisbad gestellt wurde. Vor der Elektrophorese wurde GelRed dem Agarosegel zugesetzt, um die DNA-Stränge zu färben. Als der Vorgang abgeschlossen war, wurde ein Bio-Rad-Fluoreszenzscanner (Kalifornien, USA) verwendet, um ein Bild des Gels aufzunehmen.
Dynamische Lichtstreuung
Ein Malvern Zetasizer ZS90 (Malvern, UK) wurde verwendet, um die hydrodynamische Größe und das Zetapotential des TDN zu messen. Insgesamt 1 µl des TDN (100 µnM) wurde einer dynamischen Lichtstreuungsanalyse (DLS) unterzogen.
Atomkraftmikroskopische Bildgebung
Die TDN wurden mit TM-Puffer (Tris-HCl-Puffer mit MgCl2 . auf 100 nM verdünnt ). Dann wurden 10 &mgr;l jeder TDN-Probe zu frisch gespaltenem Glimmer hinzugefügt und 10 &mgr;m inkubiert. Die Proben wurden anschließend auf einem Atomic Force Microscopy (AFM)-Instrument im AC-Modus (Agilent 5500, USA) abgebildet.
Messung der Wirkstoffbeladungskapazität des vorbereiteten TDN
Doxorubicin wurde in entionisiertem Wasser gelöst, um eine 500-μM-Aufbewahrungslösung herzustellen. Doxorubicin in verschiedenen Konzentrationen (1 bis 20 µM) wurde mit TDN (100 µM) oder Apt-TDN (100 µM) für 6 Stunden bei Raumtemperatur (24–26 °C) gemischt. Die gemischten Lösungen wurden dann bei 12.000 × g . zentrifugiert für 10&supmin; Minuten, um das arzneimittelbeladene TDN zu erhalten. Dann wurden 50 µl der Überstände entfernt und mit PBS im Verhältnis 1:1 gemischt. Ein Varioskan LUX Mikroplatten-Lesegerät (Kalifornien, USA) wurde verwendet, um die Fluoreszenzintensität von Doxorubicin (λ ex =480 nm und λ em =590 nm), um die Doxorubicin-Menge in den Überständen zu bestimmen [28]. Die Konzentration des im TDN beladenen Doxorubicins wurde anhand der Standardkurve und der Fluoreszenzintensität berechnet. Wir haben auch Doxorubicin mit dem TDN in steigenden Molverhältnissen gemischt.
Serumstabilität des TDN in Vitro
Die TDN wurden mit Komplettmedium gemischt und bei 37 °C für 0, 2, 4, 6, 8, 10, 12 oder 24 h inkubiert. Die TDN-Lösungen wurden mit FBS im Verhältnis 1:1 gemischt und bei 37 °C für 1, 3, 5 oder 7 h inkubiert. Nach der Inkubation wurden die Mischungen auf einem 3% Agarosegel laufen gelassen.
Zytotoxizität des TDN in vitro
Um die Zytotoxizität des TDN zu bestimmen, wurden U87MG-Zellen in einer Konzentration von 1 × 10 4 Zellen/Well wurden auf eine 96-Well-Platte ausgesät. Das Zellkulturmedium wurde entfernt und frisches Medium, das 0–500 nM TDN enthielt, wurde zugegeben und nach der Inkubation über Nacht für weitere 24 h und 48 h inkubiert. Dann wurden 10 &mgr;l CCK-8-Lösung in jede Vertiefung gegeben und die Mischung wurde 1 h lang inkubiert. Die Extinktion bei 450 nm wurde dann mit einem Mikroplatten-Lesegerät gemessen.
Fluoreszenzbildgebung
Die zelluläre Aufnahme von DOX und TDN wurde durch Fluoreszenzmikroskopie (Olympus, Tokio, Japan) untersucht. U87MG-Zellen wurden auf Deckgläsern in 24-Well-Platten mit Medium, das 10 % hitzeinaktiviertes fötales Rinderserum und 1 % Penicillin und Streptomycin enthielt, ausgesät und für mindestens 1 Tag bei 37 °C in einer befeuchteten Atmosphäre mit 5 % CO . gezüchtet 2 bis die Zellen mindestens 75% Konfluenz erreichten. Nach der Inkubation wurden die Kulturmedien entfernt. Vollständige Medien, die 100 &mgr;nM Cy3-TDN und Cy3-Apt-TDN enthielten, wurden zugegeben und 3 h lang inkubiert. TDN und Apt-TDN wurden mit Cy3 markiert, um die interzelluläre Aufnahme von Nanopartikeln nachzuweisen. Um die zelluläre Aufnahme von DOX zu beurteilen, wurden DOX (DOX 2 μM), DOX@TDN (DOX 2 μM) und DOX@Apt-TDN (DOX 2 μM) zu U87MG-Zellen gegeben und für 3 Stunden inkubiert. Nach 3 Stunden Behandlung wurden die Zellen mit 4% Paraformaldehyd für 20 Minuten im Dunkeln fixiert und anschließend mit 4", 6-Diamidino-2-phenylindol (DAPI) für 5 Minuten gefärbt. Die Zellen wurden dreimal mit PBS gewaschen und unter einem Fluoreszenzmikroskop beobachtet.
Durchflusszytometrie
Insgesamt 1 × 10 6 U87MG-Zellen wurden in 6-Well-Platten implantiert. Nach der Inkubation über Nacht wurden die Kulturmedien entfernt und mit 100 &mgr;nM Cy3-TDN, 100 &mgr;nM Cy3-Apt-TDN oder 100 &mgr;nM Cy3-Apt-TDN + 1 &mgr;M freies Apt ergänzte Medien wurden zugegeben und 3 h inkubiert. Dann wurden die Zellen mit 4% Paraformaldehyd für 20 min fixiert und die Durchflusszytometrie wurde verwendet, um die Prozentsätze an Cy-3-positiven Zellen zu analysieren.
Zellzyklus und Apoptose
Nach Behandlung mit DOX, DOX@TDN oder DOX@Apt-TDN für 24 h, 5 × 10 5 Zellen wurden gesammelt und über Nacht in 75% eiskaltem Ethanol fixiert. Dann wurden die Zellen mit RNase und Propidiumiodid 30 min bei 37 °C im Dunkeln inkubiert. Der Zellzyklus wurde durchflusszytometrisch untersucht. Darüber hinaus wurden die Zellen nach den verschiedenen Behandlungen mit Annexin V-FITC/DAPI gefärbt und die frühe Apoptose untersucht.
CCK-8-Assays
Um die Lebensfähigkeit der Zellen zu bestimmen, U87MG-Zellen (5 × 10 3 ) wurden auf 96-Well-Platten mit 100 µl Medium ausgesät und über Nacht bei 37 °C unter einer Atmosphäre mit 5 % CO2 . kultiviert . Das Medium wurde anschließend entfernt und frisches Medium, das DOX, DOX-TDN oder DOX-Apt-TDN enthielt, wurde zugegeben. Nach 24 h Inkubation wurden 10 &mgr;l CCK-8-Lösung zugegeben und die Zellen wurden für weitere 1 h kultiviert. Ein Mikroplatten-Lesegerät wurde verwendet, um die Extinktion bei 450 nm zu messen.
Statistische Analyse
Alle Experimente in dieser Studie wurden dreifach durchgeführt und alle Daten werden als Mittelwert mit seiner Standardabweichung (Mittelwert ± SD) dargestellt. Die statistische Analyse wurde mit dem Programm SPSS 24.0 (IBM, USA) durchgeführt. Signifikante Unterschiede wurden mit Student's t . bestimmt testen, mit P <0,05, was auf signifikante Unterschiede zwischen den Gruppen hinweist.
Ergebnisse
Synthese und Charakterisierung des TDN und Apt-TDN
Das TDN wurde aus vier Oligonukleotiden (Tabelle 1) über eine einstufige Synthese selbstorganisiert, wie zuvor beschrieben [18, 29]. Das tumorzielende Aptamer Gint4.T wurde verwendet, um TDN über Watson-Crick-Basenpaarung zu modifizieren. Das DNA-Tetraeder enthält vier Seiten, wobei jede Seite von einem Oligonukleotid gebildet wird. Somit hybridisierten vier Oligonukleotide miteinander, um ein DNA-Tetraeder zu bilden (Fig. 1a). Die Gelelektrophoreseanalyse zeigte eine einzelne prominente Bande in den Spuren 4 und 5, was darauf hindeutet, dass TDN und Apt-TDN erfolgreich konstruiert wurden. Die Mobilität des Apt-TDN war im Vergleich zu der des TDN verringert, was darauf hindeutet, dass das Gint4.T-Aptamer das TDN erfolgreich modifiziert hat.
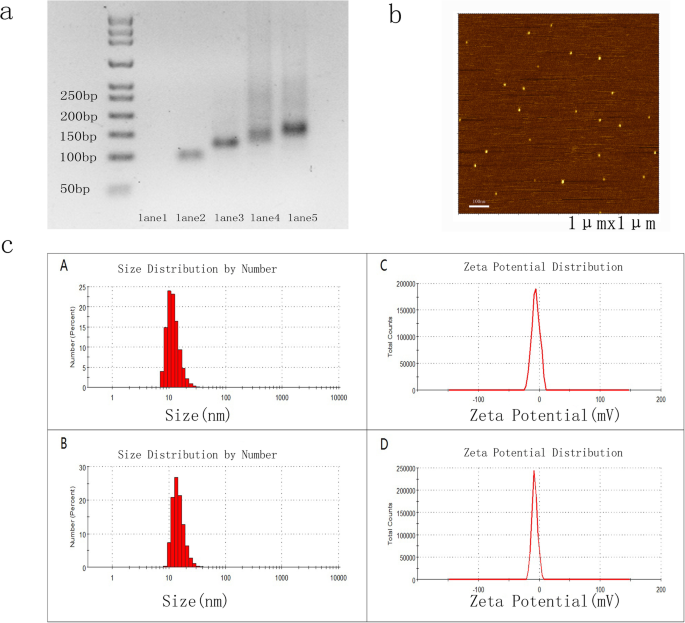
a Synthese des DNA-Tetraeders und Gint4.T-TDN. Spur 1:S1; Bahn 2:S1+S2; Spur 3:S1+S2+S3; Spur 4:S1+S2+S3+S4 (TDN); Spur 5:TDN gemischt mit Apt-Schwanz (Gint4.T); Apt-TDN. Spur 1 war nicht sichtbar, da Nukleinsäurefarbstoffe einzelsträngige DNA nicht richtig anfärben können. b AFM-Bilder zeigten, dass die Höhen von TDN und Apt-TDN ~ 2 nm betrugen. c Bestimmung der Partikelgröße und des Zetapotentials des TDN und Apt-TDN durch dynamische Lichtstreuung (DLS). Die durchschnittlichen Partikelgrößen von TDN und Apt-TDN betrugen 10,10 nm (A) bzw. 13,54 nm (B). Die durchschnittlichen Zetapotentiale von TDN und Apt-TDN betrugen −5,69 mV (C) bzw. −7,3 mV (D)
Die Größen des TDN und Apt-TDN wurden durch DLS und AFM bestimmt. Das TDN und Apt-TDN zeigten Partikelgrößen von 10,1 nm bzw. 13,5 nm, was die Zugabe des Gint4.T-Liganden widerspiegelt (Abb. 1c (A), (B)). Da der hydrodynamische Durchmesser Wassermoleküle umfasst, waren die Partikel größer als ihre theoretischen Größen. Die Höhen sowohl des TDN als auch des Apt-TDN, bestimmt durch AFM-Bilder, betrugen ~ 2 nm (Abb. 1b), was darauf hindeutet, dass die Aptamermodifikation die 3D-Struktur nicht veränderte. Die durchschnittlichen Zetapotentiale von TDN und Apt-TDN betrugen − 5,69 mV (C) bzw. − 7,3 mV (D) (Abb. 1c (C) (D)). Basierend auf diesen Parametern kamen wir zu dem Schluss, dass TDN und Apt-TDN erfolgreich zusammengestellt wurden.
Stabilität und Zytotoxizität des TDN in vitro
Die Gelelektrophorese-Analyse zeigte, dass das TDN intakt blieb, wenn es in Vollmedium für 24 Stunden bei 37 °C inkubiert wurde (Fig. 2a (A)). Wenn die Konzentration des fetalen Rinderserums auf 50 % erhöht wurde, blieb das TDN außerdem mindestens 7 h lang stabil (Abb. 2a (B)), was mit früheren Berichten übereinstimmt [18, 26]. Um die Zytotoxizität der Nanostruktur zu bestimmen, wurde der CCK-8-Assay verwendet, um die Zelllebensfähigkeit von U87MG-Zellen nach Behandlung mit dem TDN in einer Reihe von Konzentrationen zu bestimmen. Wie in 2b gezeigt, wurde keine signifikante Zytotoxizität in U87MG-Zellen beobachtet, die mit TDN bei 0–500 nM für 24 h und 48 h behandelt wurden. Daher können DNA-Nanopartikel als stabile und biosichere Träger für die Wirkstoffabgabe verwendet werden.
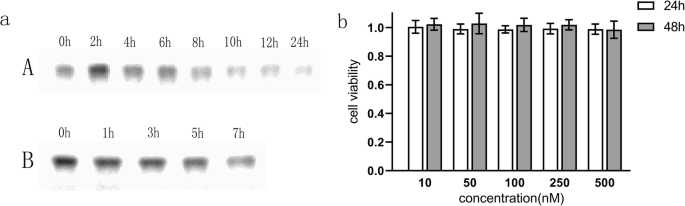
a Die Gelelektrophorese zeigte, dass das TDN im Vollmedium bei 37 °C 24 h lang stabil blieb (A); das TDN blieb 7 Stunden lang stabil, wenn die Konzentration des fötalen Rinderserums auf 50 % erhöht wurde (B). b U87MG-Zellen wurden mit dem TDN in verschiedenen Konzentrationen (10–500 nM) für 24 h und 48 h cokultiviert. Der CCK-8-Test zeigte, dass die Aktivität der U87MG-Zellen nicht beeinflusst wurde, was auf die Biosicherheit des TDN hinweist
Medikamentenladekapazität des TDN und Apt-TDN
Doxorubicin ist ein Chemotherapeutikum mit breitem Spektrum, das in DNA-Doppelstränge interkalieren kann. Wir berechneten eine Standard-Doxorubicin-Kurve (Abb. 3a) und untersuchten dann die Interkalation von Doxorubicin in das TDN. Die Menge an interkaliertem Doxorubicin in TDN und Apt-TDN nahm mit steigender Doxorubicin-Konzentration allmählich zu. Wenn die Doxorubicin-Konzentration 14 µM betrug, erreichte die Menge an Doxorubicin, die in TDN und Apt-TDN interkaliert war, einen Höchststand von 5,5 µM bzw. 6,0 µM und erreichte anschließend ein Plateau (Abb. 3b), was darauf hindeutet, dass die DNA-Stränge vollständig besetzt waren. Inzwischen haben wir Doxorubicin mit dem TDN in steigenden Molverhältnissen gemischt. Das Fluoreszenzspektrum von Doxorubicin wurde zur Analyse gescannt. Wie in 3c gezeigt, wurde das Fluoreszenzspektrum von Doxorubicin mit Doxorubicin in einem Molverhältnis von 0,05 gelöscht. Basierend auf diesen Ergebnissen kamen wir zu dem Schluss, dass ungefähr 55 Moleküle Doxorubicin in einem einzelnen TDN enthalten waren, während 60 Moleküle in einem einzelnen Apt-TDN enthalten waren.
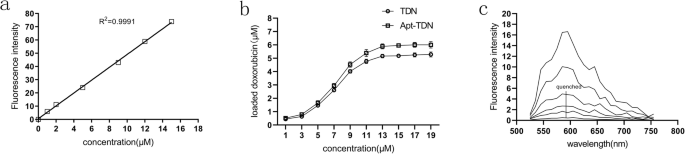
a Eine Standardkurve der DOX-Konzentrationen in PBS-Puffer; λex =480 nm und λem =590 nm. Die vom TDN und Apt-TDN getragene DOX-Menge. b DOX interkalierte in die doppelsträngige DNA von TDN und Apt-TDN. Wenn die DOX-Konzentration 14 µM erreichte und die interkalierte DOX-Konzentration in TDN und Apt-TDN einen Höchststand von 5,5 µM bzw. 6,0 µM erreichte, konnte ein einzelnes DNA-Tetraeder 55 Dox-Moleküle tragen, während ein einzelnes Aptamer-modifiziertes DNA-Tetraeder 60 DOX . trug Moleküle. c Fluoreszenzspektren von DOX im Überstand. Doxorubicin wurde mit dem TDN in steigenden Molverhältnissen (0, 0,0005, 0,001, 0,005, 0,01 und 0,05 von oben nach unten) gemischt. Bei einem Molverhältnis von 1:20 wurde die Fluoreszenz gelöscht
Gezielte Mobilfunkaufnahme des Apt-TDN
DNA ist ein negativ geladenes Makromolekül, das den Eintritt in die negativ geladene Zellmembran erschwert. Typischerweise müssen einzelne DNA-Moleküle mit Hilfe eines Transfektionsreagenzes auf Zellen zugreifen. Hier haben wir TDN und Apt-TDN mit Cy3 markiert, um die intrazelluläre Aufnahme der Nanopartikel zu überwachen. Nach 3 h Inkubation mit U87MG-Zellen trat ein rotes Cy3-Fluoreszenzsignal im zellulären Zytoplasma auf, was darauf hinweist, dass die TDN die Zytomembran banden und ohne Hilfe von Transfektionsmitteln in die Zelle aufgenommen wurden ( 4a ). Das Apt-TDN zeigte eine höhere rote Fluoreszenz, was darauf hindeutet, dass die Anwesenheit des Gint4.T-Aptamers die DNA-Tetraederaufnahme durch U87MG-Zellen signifikant erhöht. Wenn jedoch freies Aptamer zugegeben wurde, nahm die Cy3-Fluoreszenz auf das Niveau ab, das für das TDN allein beobachtet wurde. Aufgrund der kompetitiven Hemmung durch das freie Aptamer folgern wir, dass das Aptamer auf dem TDN die Aufnahme des TDN nicht erleichtern konnte. Basierend auf dieser kompetitiven Hemmung haben wir bewiesen, dass das Apt-TDN auf U87MG-Zellen abzielen kann. Die Durchflusszytometrie zeigte weiterhin, dass der Prozentsatz an Cy3-positiven U87MG-Zellen in der Apt-TDN-Gruppe höher war als in der TDN-Gruppe. Freies Apt verringerte den Prozentsatz an Cy3-positiven U87MG-Zellen in der Apt-TDN-Gruppe (Abb. 4b).
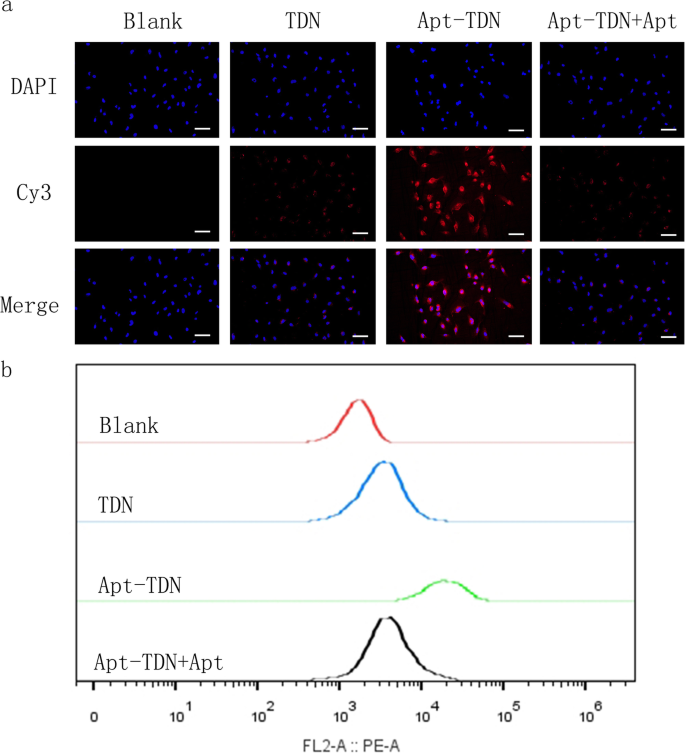
a U87MG-Zellaufnahme von TDN und Apt-TDN (TDN-Gint4.T). Das TDN trat direkt ohne Transfektionsmittel in U87MG-Zellen ein, und die Aufnahme von Apt-TDN (verbunden mit dem Aptamer Gint4.T) wurde signifikant erhöht und durch freies Apt (Gint4.T) kompetitiv gehemmt, was darauf hindeutet, dass das Aptamer Gint4.T a . spielt wichtige Rolle beim zellulären Targeting. Der Maßstabsbalken steht für 50 μm. b Durchflusszytometrie-Kurven zeigen die intrazelluläre Aufnahme von TDN, Apt-TDN und Apt-TDN+Apt nach Inkubation für 3 h
Mobile Aufnahme von DOX@TDN und DOX@Apt-TDN
Wir nutzten das charakteristische Fluoreszenzspektrum von Doxorubicin, um die Wirksamkeit der Wirkstoffaufnahme zu beurteilen. Nach 3 Stunden Behandlung wurde intrazelluläres Doxorubicin durch Fluoreszenzmikroskopie abgebildet (Fig. 5a). Freies Doxorubicin konnte in U87MG-Zellen eindringen und befand sich im Zellkern. Bei Zugabe von DOX@TDN war die Fluoreszenz höher als die von freiem Doxorubicin. Dieses Ergebnis deutet darauf hin, dass DNA-Nanopartikel die zelluläre Aufnahme von Doxorubicin verbesserten. Bei Zugabe von DOX@Apt-TDN war das rote Signal im Zellkern noch höher als bei Zellen, denen DOX@TDN zugesetzt wurde. Die semiquantitative Analyse der intrazellulären Aufnahme von DOX bestätigte weiterhin, dass das Apt-TDN eine mehr als zweifache Erhöhung der intrazellulären DOX-Aufnahme im Vergleich zu der des einzelnen Wirkstoffs ermöglichte. Wir schließen, dass dies auf die Gin4.T-spezifische Bindung an Rezeptoren zurückzuführen ist, wodurch mehr Nanopartikel in die Zelle gelangen können. Nach der Verdauung in Lysosomen kann Doxorubicin in das Zytoplasma freigesetzt werden und im Zellkern weitere Funktionen erfüllen.
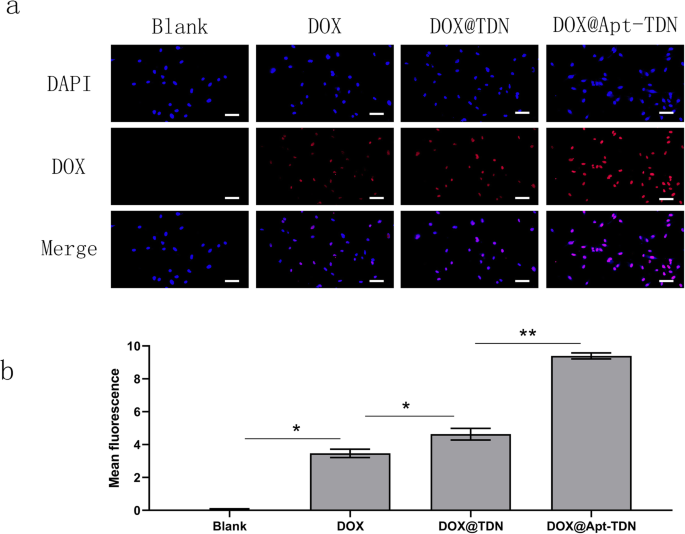
a Zelluläre Aufnahme von DOX, DOX@TDN und DOX@Apt-TDN. Modifiziert mit dem Aptamer Gint4.T könnte das Apt-TDN mehr Doxorubicin an U87MG-Zellen abgeben als das TDN. Darüber hinaus könnte das TDN mehr Arzneimittel zu den Zellen transportieren als das Arzneimittel allein. Die Skalenbalken zeigen 50 μm an. b Semiquantitative Analyse der Fluoreszenzintensität von Doxorubicin mit PBS-, DOX-, DOX@TDN- und DOX@Apt-TDN-Behandlung (im Vergleich zu Leerwert:*p <0,05, **p <0,01)
Zytotoxizität von DOX, DOX@TDN und DOX@Apt-TDN
Für die Zytotoxizitätsstudie wurden drei Gruppen von U87MG-Zellen mit unterschiedlichen Konzentrationen von Doxorubicin behandelt (Abb. 6a). Der IC50 die Werte für Doxorubicin betrugen 13,39 µM für die DOX-Behandlung, 7,826 µM für die DOX@TDN-Behandlung und 4,205 µM für die DOX@Apt-TDN-Behandlung. Unter den drei Behandlungen zeigte das DOX@Apt-TDN die höchste Zytotoxizität nach 24 h, was die Spezifität des Apt-TDN für U87MG-Zellen anzeigte. Nach 24 Stunden wurden Zellen der DOX-, DOX@TDN- und DOX@Apt-TDN-Gruppen gesammelt und verwendet, um die frühe Apoptose zu untersuchen. Unsere Daten zeigten, dass die frühe Apoptoserate in der DOX@Apt-TDN-Gruppe höher war als in den anderen beiden Gruppen (Abb. 6b). Darüber hinaus war der Anteil der Zellen in der G0/G1-Phase in der DOX-Apt-TDN-Gruppe im Vergleich zu den DOX- und DOX-TDN-Gruppen erhöht (p <0,01) und der Anteil der Zellen in der S-Phase war in der DOX-Apt-TDN-Gruppe verringert (p <0,01) (Abb. 6c). Der Prozentsatz der Zellen in der G2-Phase wurde nicht geändert (Daten nicht gezeigt).
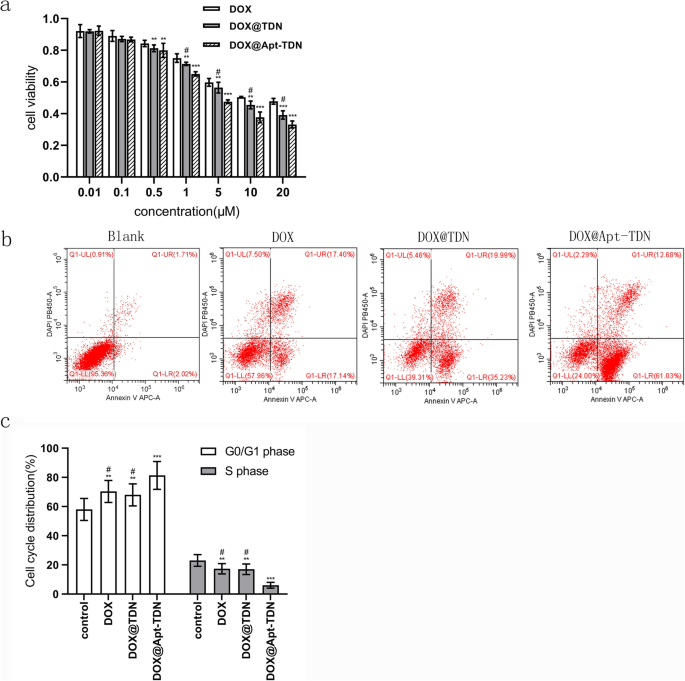
a Zytotoxizität von DOX, DOX@TDN und DOX@Apt-TDN bei verschiedenen Konzentrationen. Die Hemmungsrate der U87MG-Zellen war mit steigender DOX-Konzentration signifikant erhöht, jedoch zeigten die DOX@TDN- und DOX@Apt-TDN-Gruppen eine signifikant erhöhte Zytotoxizität im Vergleich zur DOX-Gruppe. Die Zellhemmungsrate der DOX@Apt-TDN-Gruppe war ebenfalls signifikant höher als die der DOX@TDN-Gruppe (im Vergleich zu DOX, **p < 0,05; im Vergleich zu DOX, ***p <0,01; im Vergleich zu DOX@Apt-TDN, # p <0,05). b Apoptose von U87MG-Zellen nach Inkubation mit PBS, DOX, DOX@TDN und DOX@Apt-TDN für 24 h. c Durchflusszytometrie-Histogramme des U87MG-Zellzyklus nach Inkubation mit PBS, DOX, DOX@TDN und DOX@Apt-TDN für 24 h (im Vergleich zur Kontrolle, **p < 0,05; im Vergleich zur Kontrolle, ***p <0,01; im Vergleich zu DOX@Apt-TDN, # p <0,01)
Diskussion
Ob durch In-vitro- oder In-vivo-Experimente, die Stabilität eines Arzneimittels und seines Trägers muss bestimmt werden. Nach dem Zusammenbau des TDN wurde seine Stabilität zunächst in vitro bestimmt. Diese Studie zeigte, dass die 3D-Struktur von DNA-Nanopartikeln ihre Stabilität im Serum verbessern kann, indem sie die Enzymbindung hemmt. Die biologische Sicherheit der jeweiligen Nanostruktur ist die wichtigste Voraussetzung für deren Anwendung. Keine signifikante Zytotoxizität wurde in U87MG-Zellen beobachtet, die mit verschiedenen Konzentrationen des TDN für 24 h oder 48 h cokultiviert wurden. Keine der in dieser Studie verwendeten DNA-Sequenzen kodiert eine genetische Information, und im Zytotoxizitätstest wurden keine Nebenwirkungen berichtet. Daher kann das TDN als sicherer und stabiler Arzneimittelträger dienen.
Die zielgerichtete Effizienz einer Aptamer-modifizierten Nanostruktur ist entscheidend für die selektive Wirkstoffabgabe an Krebszellen. Im Gegensatz zu Antikörpern sind Aptamere chemisch stabil, kostengünstig und können in Massen hergestellt werden. Darüber hinaus können Aptamere im Gegensatz zu anderen Materialien leicht ein DNA-Tetraeder nach dem Basenkomplementärprinzip binden. Somit legt die Kombination von Aptameren und DNA-Tetraedern die Grundlage für eine gezielte Wirkstoffabgabe und Behandlungen der nächsten Generation. Unsere Ergebnisse zeigten, dass das TDN ohne Transfektionsmittel in Zellen eindringen konnte, was den Ergebnissen von Walsh et al. und Ma et al. [15, 19]. Im Vergleich zu der des TDN war die Aufnahme des Apt-TDN durch U87MG-Zellen signifikant erhöht. Nach Zugabe von freiem Aptamer verschwand dieser Anstieg. Dieses Ergebnis legt nahe, dass Gint4.T ein PDGFRβ-spezifischer Antagonist ist [25]. Unsere Studie zeigte, dass das Gint4.T-Aptamer aufgrund der hohen Expression von PDGGRβ auf der Oberfläche von Gliomzellen auf U87MG-Zellen abzielen könnte. Camoraniet al. [24] zeigten auch, dass das Gint4.T-Aptamer Tumorzellen angreifen kann, indem es spezifisch mit der extrazellulären Domäne von PDGFRβ interagiert. Die Vorteile des Gint4.T-Aptamer-Targetings wurden in gewissem Maße durch In-vitro-Studien nachgewiesen, aber es sind weitere In-vivo-Tests erforderlich, um diese Ergebnisse zu bestätigen. Diese Studie bestätigte auch, dass DOX@Apt-TDN zytotoxischer ist als DOX oder DOX@TDN, was wahrscheinlich auf zwei Faktoren zurückzuführen ist. Erstens kann das Gint4.T-Aptamer spezifisch extrazelluläre PDGFR-Domänen binden, die Tumorzellproliferation blockieren und das Tumorzellwachstum hemmen [24]. Dies stimmt mit unseren experimentellen Ergebnissen überein. Im Vergleich zur Kontrollgruppe stiegen die Veränderungen des U87MG-Zellzyklus nach Behandlung mit DOX, DOX@TDN und DOX@Apt-TDN in den Tumorzellen der G0/G1-Phase signifikant an, verringerten sich in den S-Phasen-Zellen und blockierten Tumorzellen bei G0 /G1-Phase, was darauf hinweist, dass sie den Zellzyklus von U87MG-Zellen hemmen und deren Proliferation hemmen können. Im Vergleich zu den DOX- und DOX@TDN-Gruppen hatte die DOX@Apt-TDN-Gruppe eine signifikant stärkere Fähigkeit, die Proliferation von U87MG-Zellen zu hemmen. Darüber hinaus bindet Gint4.T spezifisch Tumorzellen und verbessert das Zell-Targeting des DOX@Apt-TDN-Komplexes. Auf diese Weise können wir die Wirksamkeit der Wirkstoff-Targeting-Wirkung erhöhen und die systemische Verabreichungsdosis von Antitumor-Medikamenten reduzieren, um deren systemische Nebenwirkungen zu verhindern.
Schlussfolgerungen
Die Modifikation mit Gint4.T erhöhte die Spezifität und Effizienz von TDN bei der Gliombehandlung, indem sie auf das PDGFR&bgr; abzielte, das in großen Mengen auf Gliomzellen exprimiert wird. Darüber hinaus könnte es, wenn es mit Apt-TDN beladen ist, die Anti-Gliom-Wirkung von DOX signifikant fördern. Daher kann DOX@Apt-TDN als vielversprechende therapeutische Strategie gegen Gliome für Patienten dienen. Das Manko dieser Studie ist, dass sie nur in vitro verifiziert wurde. In der späteren Studie werden wir weitere Untersuchungen in Tiermodellen durchführen.
Verfügbarkeit von Daten und Materialien
Die relevanten Daten sind im Artikel enthalten.
Abkürzungen
- BBB:
-
Blut-Hirn-Schranke
- BTB:
-
Bluttumorbarriere
- TDN:
-
Tetraedrische DNA-Nanostrukturen/DNA-Tetraeder
- DOX:
-
Doxorubicin
- PDGFRβ:
-
Thrombozyten-Wachstumsfaktor-Rezeptor β
- Apt-TDN:
-
Gint4.T-modifiziertes TDN
- DOX@ TDN:
-
TDN geladen mit DOX
- DOX@Apt-TDN:
-
Gint4.T-modifiziertes TDN geladen mit DOX
- CNS:
-
Zentralnervensystem
- ANG:
-
Angiopep-2
- LRP-1:
-
Low-density lipoprotein receptor-related protein 1
- SELEX:
-
Exponential enrichment
- PNPs:
-
Polymeric nanoparticles
- GBM:
-
Glioblastoma
- FBS:
-
Foetal bovine serum
- DMEM:
-
Dulbecco's modifiziertes Eagle's Medium
- DLS:
-
Dynamische Lichtstreuung
- AFM:
-
Rasterkraftmikroskopie
- CCK-8:
-
Cell Counting Kit-8
- Cy3:
-
Sulfo-Cyanine3
- DAPI:
-
4′,6-Diamidino-2-phenylindole
Nanomaterialien
- Optimierung der Transfereffizienz mit einer Roboterzelle
- Zielgerichtete Endothelzellen mit multifunktionalen GaN/Fe-Nanopartikeln
- Ultrasensitiver Biosensor zum Nachweis von Vibrio cholerae-DNA mit Polystyrol-Co-Acrylsäure-Komposit-Nanosphären
- Förderung des SH-SY5Y-Zellwachstums durch Goldnanopartikel, die mit 6-Mercaptopurin und einem Neuronen-penetrierenden Peptid modifiziert sind
- Untersuchung einer kristallinen Silizium-Solarzelle mit schwarzer Siliziumschicht auf der Rückseite
- Graphenoxid-basierte Nanokomposite, dekoriert mit Silbernanopartikeln als antibakterielles Mittel
- Herstellung einer 20,19 % effizienten einkristallinen Silizium-Solarzelle mit invertierter Pyramiden-Mikrostruktur
- Auswirkungen von C60-Fulleren auf die Wechselwirkung von Diphenyl-N-(trichloracetyl)-amidophosphat mit DNA in Silico und seine zytotoxische Aktivität gegen menschliche leukämische Zelllinien in vitr…
- Verbesserte photovoltaische Eigenschaften in einer Sb2S3-Planar-Heterojunction-Solarzelle mit einem schnellen Selenylierungsansatz
- Optimierung Ihrer Schweißanwendung mit einer kundenspezifischen RW950-Schweißzelle



