Redox-sensitive Gelatine/Silica-Aptamer-Nanogele für die gezielte siRNA-Lieferung
Zusammenfassung
RNA-Interferenz (RNAi) hat aufgrund ihrer hohen Spezifität und der Fähigkeit, die Zielgenexpression zu hemmen, potenzielle Vorteile gegenüber anderen Gentherapieansätzen. Die Stabilität und gewebespezifische Abgabe von siRNA bleiben jedoch die größten Hindernisse für RNAi-Therapeutika. Hier haben wir ein solches System entwickelt, indem wir Gelatine-basierte Nanogele mit dem auf Nukleolin gerichteten AS1411-Aptamer und Desoxynukleotid-substituierter siRNA (Apt-GS/siRNA) über einen Disulfid-Linker zusammenkonjugieren, um ein vorübergehendes Andocken von siRNA zu erreichen. Diese Apt-GS/siRNA-Nanogele zeigten aufgrund der Disulfidspaltung eine günstige Freisetzung von siRNA unter reduzierenden Bedingungen. Darüber hinaus könnte dieses intelligente System siRNA in Nukleolin-positiven Zellen (A549) durch einen Glutathion-getriggerten Abbau elektiv in das Zytosol freisetzen und anschließend RNAi für Luciferase effizient freisetzen. Außerdem zeigten mit Disulfid ausgestattete Apt-GS-Nanogele in vitro eine gute Biokompatibilität. Zusammengenommen bieten diese redox-responsiven, auf Tumoren ausgerichteten intelligenten Nanogele ein großes Potenzial bei der Nutzung der funktionalisierten siRNA-Lieferung und der Tumortherapie.
Einführung
RNA-Interferenz (RNAi) ist eine sequenzspezifische Stilllegung von Genen und wird derzeit aufgrund ihrer hohen Spezifität und geringen Toxizität auch als vielversprechendste Methode zur Behandlung einer Vielzahl von Krankheiten einschließlich genetischer Störungen, Krebs und Infektionskrankheiten bewertet [1]. Small interfering RNAs (siRNAs) mit doppelsträngigen RNA-Molekülen sind resistenter gegen Nukleaseabbau als Antisense-Oligonukleotide und zeigen daher Vorteile gegenüber der Antisense-Therapie. Der Einbau chemisch modifizierter Nukleotide in siRNAs wurde verwendet, um die Wirksamkeit und die Dauer von RNAi zu erhöhen oder zu verringern. Es hat sich gezeigt, dass das Ersetzen der Ribonukleotide des Sense-Strangs durch Desoxynukleotide die Stabilität des Sense-Strangs erhöhen und eine Wirksamkeit von RNA40% der RNAi beibehalten kann [2, 3]. Die gewebespezifische Abgabe von siRNA bleibt jedoch ein großes Hindernis für ihre Anwendungen, trotz der großen Gen-Knockdown-Potenzen, die in den In-vitro-Studien beobachtet wurden [4, 5].
Die Abgabe auf Nanopartikelbasis hat potenzielle Vorteile bei der effektiven siRNA-Stabilisierung und weiteren Modifikation für die ortsspezifische Abgabe [6,7,8]. Eine produktive ortsgerichtete Abgabe von siRNA kann die Transfektion verbessern und Off-Target-Effekte reduzieren, die für die meisten praktischen Anwendungen erforderlich sind [9]. Nucleolin wird mit diversen biologischen Prozessen in Verbindung gebracht und wild im Kern und Zytoplasma verschiedener normaler Zellen exprimiert. Darüber hinaus wird Nukleolin auf den Plasmamembranen von aktiv proliferierenden Krebszellen im Vergleich zu ihren normalen Gegenstücken stark exprimiert und wird daher als attraktives Ziel für antineoplastische Behandlungen verwendet. Aptamer AS1411 (auch bekannt als AGRO100), ein G-Quartett-DNA-Aptamer, kann stark an Zelloberflächennukleolin binden und den antiapoptotischen Weg in Krebszellen blockieren, indem es mit dem essentiellen nuklearen Faktor-kB-Modulator kombiniert wird erstes Nukleinsäureaptamer-Medikament zur Behandlung von Krebs beim Menschen [10, 11]. Bisher wurde AS1411 erfolgreich an verschiedene Nanopartikel konjugiert und in Krebszellen internalisiert [12,13,14,15]. Gelatine wurde aufgrund ihrer ausgezeichneten Biokompatibilität, biologischen Abbaubarkeit und Gelbildungseigenschaften für die siRNA-Verkapselung verwendet [16,17,18]. Unsere Gruppe hat eine Reihe von Siloxan-vernetzten Gelatine-Nanogelen (GS-NGs) mit kontrollierter Größe und Oberflächenladung untersucht und die Transfektionseffizienz von GS-NGs in vitro und in vivo demonstriert [19, 20].
Neben intrazellulären Barrieren bleiben auch intrazelluläre Barrieren nach der Internalisierung, einschließlich des effizienten Abbaus von siRNA und des endosomalen Entweichens, eine Herausforderung. Die Konjugation von siRNA an einen Träger durch labile oder nicht-labile Bindungen ist eine vielversprechende Methode, um diese Herausforderung beim Transport zu überwinden. Die auf Stimuli ansprechende Freisetzung unter saurem pH und Redoxpotential hat großes Interesse gefunden. Die Disulfidquervernetzung zeigt aufgrund eines höheren Glutathion (GSH)-Spiegels in der extrazellulären Umgebung ein großes Potenzial bei einer Freisetzung von Wirkstoffen über gespaltene Bindungen in Tumorzellen [21, 22]. Es wurde berichtet, dass Poly(milchsäure-co-glycolsäure) (PLGA)/siRNA-Konjugate, die über eine Disulfidbindung gebildet werden, eine verbesserte Einkapselungs- und Abgabeeffizienz aufweisen [23].
In dieser Arbeit wurden hybride Nanogele auf der Basis von GS mit dualen Funktionen der Redox-Reaktionsfähigkeit der Disulfid-Konjugation und dem spezifischen Tumor-Targeting von Aptamer AS1411 als siRNA-Träger für die Krebstherapie untersucht (Schema 1). Unser Ziel ist es zu untersuchen, ob AS1411 funktionelle GS NGs die effektive zelluläre Internalisierung verbessern und eine tumorspezifische Gen-Silencing des Luciferase-Modellgens in vitro erreichen würden. Außerdem wurde die Sicherheit dieses Systems auch in vitro bewertet.
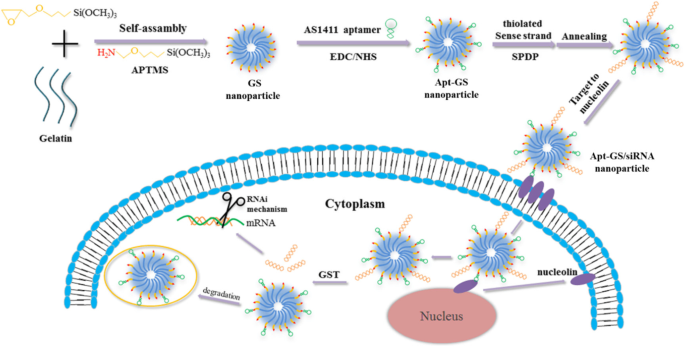
Für die siRNA-Lieferung mit Disulfid-Konjugation und Aptamer AS1411 wurde ein redox-responsives und tumor-targeting Apt-GS NGs entwickelt
Methoden
Materialien
Gelatine (Blütezahl:240–270, pH 4,5–5,5) wurde von BBI Company Inc. (USA) bezogen. N -Succinimidyl-3-(2-pyridyldithio)propionat (SPDP), 3-Glycidoxypropyl-trimethoxysilan (GPSM) und 3-Aminopropyl-trimethoxysilan (APTMS) wurden von Sigma-Aldrich Co. (USA) bezogen. 3-(4,5-Dimethylthiazolyl-2)-2,5-diphenyltetrazoliumbromid (MTT) wurde von Amresco Co. (USA) bezogen. Dulbeccos modifiziertes Eagle-Medium (DMEM), fötales Rinderserum, Penicillin und Streptomycin wurden von Hyclone Co. (USA) bezogen. AS1411-Aptamer, 5'-FAM-markiertes AS1411 und Negativkontroll-DNA (TDO) wurden von Sangon Biotech Co. (China) synthetisiert. Thiol-Sense-Strang der Luc-siRNA, 5′-SH-(CH2 )6 -CTTACGCTGAGTACTTCGATT-3′ (Desoxynukleotide ersetzen Ribonukleotide) und Antisense-Strang, 3'-TTGAAUGCGACUCAUGAAGCU-5′ und FAM-markierter Antisense-Strang am 3'-Ende wurden synthetisiert und mit HPLC von Gene Pharma Co. ( China). Lipofectamine 2000, Luciferase-Plasmid pGL3 und Luciferase-Assay-System wurden von Promega Co. (USA) bezogen. Die malignen menschlichen Lungenadenokarzinom-A549-Zellen und normale NIH-3T3-Fibroblasten wurden verwendet und vom Biomedical Engineering Center der Xiamen University (China) bereitgestellt. Alle verwendeten Materialien waren von analytischer Qualität und ohne weitere Reinigung. Die Glaswaren wurden gründlich gereinigt und mit entionisiertem Wasser gespült.
DNA-Sequenzen sind wie folgt:
AS1411 Aptamer:5′-GGTGGTGGTGGTTGTGGTGGTGGTGGTTTTTTTTTTTT-3′, Kontroll-TDO:5′-CACCGGGAGGATAGTTCGGTGGCTGTTTTTTTTTTTTT-3′
Herstellung und Charakterisierung von Apt-GS/siRNA-Komplexen
Aminofunktionalisierte Gelatine/Silica-Nanogele (GS NGs) wurden zuerst durch ein Sol-Gel-Verfahren hergestellt, wie zuvor beschrieben [19]. Typischerweise wurden 0,2 µg GPSM zu 20 µl einer 0,75%igen Gelatinelösung in HCl-Lösung (pH =~3) bei 60 °C unter 30 Minuten Rühren zugegeben und anschließend 0,08 µg APTMS zugegeben und weitere 24 Stunden inkubiert. Die erhaltenen GS-NGs wurden durch dreimalige Zentrifugation (14.000 U/min, 25 °C, 12 min) gereinigt. Zweitens wurden GS NGs (0,5 µl, 5 µg/l in PBS) mit 25 µl SPDP (20 µm in DMSO) 60 Minuten lang bei Raumtemperatur behandelt, gefolgt von der Zugabe von thioliertem AS1411 (1 µg/ml) und Rühren für 12 Uhr. Apt-GS-Nanogele wurden durch Zentrifugation (14.000 U/min, 25 °C, 12 min) erhalten und dreimal mit entionisiertem Wasser gereinigt. Drittens wurde die erhaltene SPDP-aktivierte Apt-GS NG-Suspension (80 ml, 5 mg/ml) direkt über Nacht bei 4 °C mit dem siRNA-Sense-Strang gemischt. Nach der Reinigung durch Zentrifugation wurde der siRNA-Antisense-Strang hinzugefügt und für 5 Minuten bei 94 °C inkubiert, dann für 20 Minuten bei 47 °C angelagert, um Apt-GS/siRNA-Komplexe zu bilden.
Die Oberflächenmorphologien von AS1411-GS und AS1411-GS/siRNA-Komplexen wurden durch Transmissionselektronenmikroskopie (TEM) (Hitachi S-4300, Japan) untersucht. Die Partikelgröße und das Zetapotential jeder Probe wurden mit einem Nano-ZS Zetasizer dynamischer Lichtstreuungsdetektor (Malvern Instruments, UK) gemessen. Die effektiven Partikeldurchmesser wurden aus der Autokorrelationsfunktion unter Verwendung der Malvern Zetasizer-Software unter Annahme einer logarithmischen Normalverteilung berechnet. Die Konjugationen von GS und AS1411 oder siRNA wurden durch Sammeln und Messen der Konzentration von ungebundener FAM-markierter DNA unter Verwendung von F-7000-Fluoreszenzmikroskopie (Olympus-IX73, Japan) bestimmt. Gesamtaminogruppe (-NH2 )-Niveaus auf der Oberfläche von GS-Nanogelen wurden mithilfe der kolorimetrischen Reaktion von Ninhydrin quantitativ bestimmt. Die Menge betrug etwa 0,642 mmol/g.
Gelelektrophorese-Analyse
Die Gelelektrophorese wurde bei 100 V für 20–30 min unter Verwendung von 2% (w/v) Agarosegel in TBE-Puffer durchgeführt. Die Bilder wurden durch Bestrahlung mit einem Geldokumentationssystem (Tanon GIS-2008, China) beobachtet. Im Apt-GS/siRNA-Verkapselungsassay wurden die nackten siRNA-, GS/siRNA- und Apt-GS/siRNA-Komplexe ohne jegliche Zusätze in das Gel geladen. In redox-responsiven Assays wurden der GS/siRNA- und der Apt-GS/siRNA-Komplex mit 10 mM GSH-Lösung in PBS 2 h vor der Messung inkubiert.
In-vitro-Zytotoxizität
Die Zytotoxizität gegen humane Lungenadenokarzinom-A549-Zellen wurde durch den MTT-Assay bewertet. Kurz gesagt, A549-Zellen (1 × 10 4 Zellen/Vertiefung) wurden in Polystyrol-Kulturplatten mit 96 Vertiefungen ausgesät und 24 h lang bis zur 70%igen Konfluenz inkubiert. Nach dem Entfernen des Kulturmediums wurden 100 µl serumfreies DMEM-Medium mit Nanogelen (100–600 mg/ml) in jede Vertiefung gegeben. Mit Medium behandelte Zellen dienten nur als negative Kontrollgruppe. Nach 24 Stunden Co-Inkubation wurden 100 µl frisches Medium mit 20 µl MTT-Lösung (5 µl/ml in PBS-Puffer) zugegeben und weitere 4 Stunden kultiviert. Dann wurde die MTT-Lösung entfernt und 100 µl Dimethylsulfoxid (DMSO) wurden zugegeben. Nach 30 min Oszillation im Dunkeln wurde die Extinktion jedes Wells mit einem Mikroplatten-Lesegerät (TECAN DNA Export, Swiss) bei einer Wellenlänge von 490 nm gemessen. Alle Experimente wurden dreifach durchgeführt. Die relative Lebensfähigkeit der Zellen (%) wurde als Prozentsatz relativ zu den unbehandelten Kontrollzellen ausgedrückt.
Zellinternalisierung
Die Zellen werden mit 25 µl FAM-markierten Apt-GS- und TDO-GS-Nanogelen (2 µg/ml) für 10 Stunden in serumfreiem Medium bei 37 °C cokultiviert. Nach dreimaligem Waschen mit PBS wurden die Zellen mit Paraformaldehyd (4% in PBS) für 30 min fixiert und dann mit einem konfokalen Laserscanningmikroskop (Leica, Deutschland) beobachtet. Um die zelluläre Aufnahme zu quantifizieren, wurden die Zellen mit 25 µl FAM-markiertem AS1411-GS und TDO-GS (2 µg/ml) für einen Zeitraum (0,5–16 µl) in serumfreiem Medium bei 37 °C behandelt. Um die Rolle von Nukleolin bei der zellulären Aufnahme der Apt-GS NGs zu bewerten, wurde die Mischung der hergestellten Apt-GS NGs und freien AS1411-Aptameren unterschiedlicher Konzentrationen (0,5, 5 und 50 μM) mit den A549-Zellen für 8 . inkubiert h. Dann wurden die Zellen in eiskaltem PBS resuspendiert, und die fluoreszierenden Zellen mit FAM-markierten Nanogelen wurden aus 10.000 Zellen unter Verwendung eines EPICS XL-Durchflusszytometers (Beckman Coulter, USA) gezählt. Die Datenanalyse wurde mit der EPICS XL-Durchflusszytometer-Software durchgeführt und als analytische Gates wurden 1% der Kontrollzellen ausgewählt, die in den positiven Bereich fielen.
Gene Silencing
Die Fähigkeit, Gene zum Schweigen zu bringen, wurde unter Verwendung der Kombination von siRNA für Luciferase und den Luciferase-exprimierenden Zellen untersucht. Luciferase-exprimierendes A549 (2 × 10 5 Zellen/Well) und NIH 3 T3-Fibroblasten (4 × 10 5 Zellen/Well) wurden in 12-Well-Platten ausgesät und über Nacht kultiviert und dann mit 100 µl Testkomplexen (80 µg/ml), die LUC-siRNA (oder Kontroll-siRNA) enthielten, für 8 Stunden behandelt. Die Proben wurden in drei Gruppen eingeteilt:(a) nur Apt-GS/siRNA-Komplexe für 8 h, (b) Apt-GS/siRNA-Komplexe für 8 h, gefolgt von Liposomen für weitere 1 h (bezeichnet als A-GS/si→ Lip-Gruppe) und (c) Co-Inkubation von Apt-GS/siRNA-Komplexen und Liposomen für 8 h (A-GS/si+Lip-Gruppe). Die Zellen ohne jegliche Behandlung und diejenigen, die mit dem Luciferase-Plasmid pGL3 (Madison, WI, USA) transfiziert waren, dienten als Kontrollen. Nach weiteren 40 Stunden Inkubation wurde die Gen-Knockdown-Effizienz durch Quantifizierung der Luciferase-Expression in Zellen unter Verwendung eines Luciferase-Assay-Systems gemäß dem Protokoll des Herstellers untersucht. Die Experimente wurden in dreifacher Ausfertigung durchgeführt und die Daten sind als Mittelwerte ± Standardfehler der Mittelwerte dargestellt.
Ergebnisse und Diskussionen
Synthese und Charakterisierung von GS-AS1411/siRNA-Komplexen
Um den auf Krebs abzielenden siRNA-Träger zu konstruieren, wurde ein Gelatine/Silica-AS1411 (Apt-GS)-Nanogel mithilfe einer Sol-Gel-Methode entwickelt (Schema 1) [19, 20]. Ein typisches TEM-Bild (Abb. 1) zeigte, dass leere GS und Apt-GS dispergierte kugelförmige Strukturen mit durchschnittlichen Durchmessern von 170–200 nm waren. FAM-markierte DNA wurde für die Synthese verwendet, um die Kopplung von AS1411 und siRNA an GS-NGs zu bestätigen. Die Menge an konjugiertem Aptamer auf GS betrug ungefähr 0,65 µmol/g, wie mit Fluoreszenzspektren quantifiziert. Unterdessen ist in den Fluoreszenzbildern leicht eine intensive grüne Fluoreszenz zu beobachten, was auf die Integration von Aptamer AS1411 und siRNA schließen lässt. Wie in Abb. 2 dargestellt, nahm die hydrodynamische Größe von bloßen GS-, Apt-GS- und Apt-GS/siRNA-NGs (162.3 nm, 216.5 nm bzw Oberflächenfunktionsschicht. Andererseits war das Zeta-Potential von Apt-GS weniger positiv (33,9 ± ± 0,5 µmV) als das von bloßen GS-NGs (40,1 ± ± 0,5 µmV) aufgrund des Maskierungseffekts negativer DNA-Aptamere auf der Oberfläche. Nach einer weiteren Konjugation mit siRNA auf der Oberfläche verschiebt sich das Zetapotential von Apt-GS/siRNA NGs aufgrund der überschüssigen freien negativen Phosphatgruppen von siRNA immer noch negativ auf 24.9 mV. Dieses Ergebnis deutet auf eine verringerte biologische Toxizität hin, da die starken positiven Ladungen negativ geladene Proteine im Blut stören und die Zellmembran zerstören könnten [24].
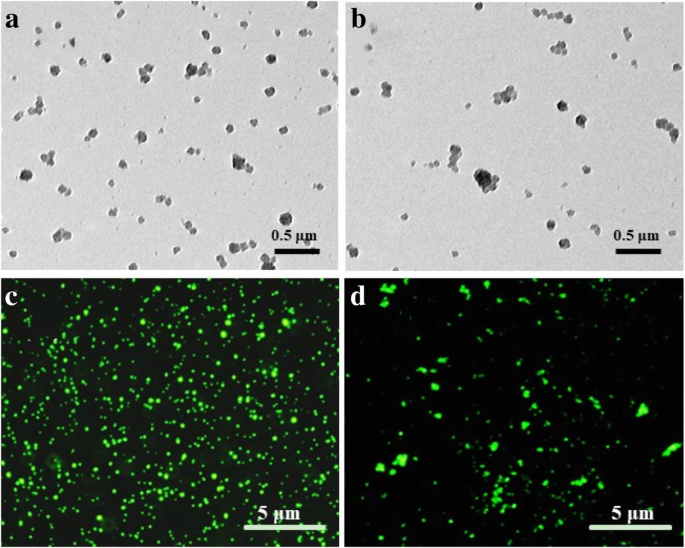
Bilder von Apt-GS (a , c ) und Apt-GS/siRNA-Nanogele (b , d ). a , b TEM-Bilder. Maßstabsbalken repräsentieren 0,5 µm. c , d Fluoreszierende Bilder. Maßstabsbalken repräsentieren 5 μm
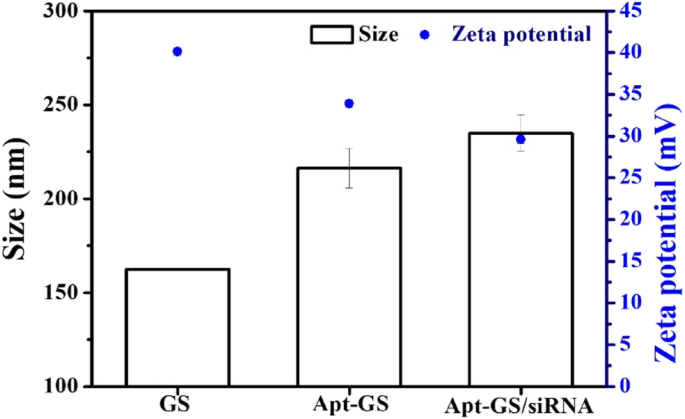
Hydrodynamische Größe und Zetapotentiale von GS-, Apt-GS- und Apt-GS/siRNA-Nanogelen
siRNA Laden und Freigeben
Eine erfolgreiche RNAi kann nur auftreten, wenn siRNA geliefert und schnell von ihrem Träger in das Zytoplasma freigesetzt wird [25]. Tumorzellen haben eine 7–10-fach höhere GSH-Konzentration als normale Zellen [26, 27]. In dieser Studie wurde siRNA durch die Disulfid-Vernetzungsreaktion an aktive Apt-GS-NGs konjugiert. Die Gelelektrophorese (GE) wurde durchgeführt, um die siRNA-Beladung und -Freisetzungsfähigkeit von Apt-GS/siRNA-Komplexen zu bewerten. Während sich die freie pDNA (Abb. 3, Spuren 1–5) in ihre übliche Position bewegte, zeigten Apt-GS/siRNA-Komplexe wie erwartet eine verringerte Mobilität in einem Elektromobilitäts-Shift-Assay (Abb. 3, Spuren 10–11), was die gute Laden von siRNA. Um den intrazellulären Zustand nachzuahmen, wurden 10&supmin;&sup7; mM GSH mit Komplexen 2 Stunden lang behandelt, um die Apt-GS/siRNA-NGs zu reduzieren. Wie in Abb. 3, Spur 6–7 gezeigt, wurden nach der Behandlung von GSH siRNA-Moleküle im Polyacrylamidgel beobachtet, was darauf hindeutet, dass Apt-GS/siRNA-NGs funktionelle siRNA-Moleküle durch Disulfidspaltung in Gegenwart von Glutathion reversibel freisetzen können. Außerdem wurde die Menge an siRNA auf den Nanopartikeln analysiert, indem die Gesamtmenge an siRNA und Kontroll-siRNA in einer 2% Polyacrylamid-Gelelektrophorese verglichen wurde. Die Apt-GS verkapselte siRNA-Moleküle in einem Verhältnis von 3:1∼2:1 pmol pro Mikrogramm. Es ist auch anzumerken, dass die Mischung aus siRNA und Apt-GS aufgrund des physikalischen Einschlusses von negativer siRNA eine schwächere Retention zeigte (Fig. 3, Spur 8).

AGE-Analyse der reversiblen Bindung von siRNA an Apt-GS-Nanogele. Spuren 1–5:freie siRNA mit einer Menge von 50, 40, 30, 20 und 10 pmol. Spuren 6 und 7:GS/siRNA und Apt-GS/siRNA mit 10&supmin;&sup6; mM GSH bei 2&supmin; h. Spur 8:die Mischung aus freier siRNA und Apt-GS-NGs. Bahnen 10 und 11:GS/siRNA- und Apt-GS/siRNA-Komplexe ohne 10 mM GSH als Kontrolle
Zytotoxizität und Nukleolin-Targeting
Die In-vitro-Zytotoxizitätsbewertung wurde durch den MTT-Assay in A549-Zellen untersucht. Der MTT-Assay wird verwendet, um die Zytotoxizität von präparierten Nanogelen zu beurteilen. Als Reporter wurde die nicht-toxische Luciferase siRNA ausgewählt, die keine Abtötungswirkung auf Tumorzellen hat [28, 29]. Daher repräsentierte die Lebensfähigkeit der Zellen die Biokompatibilität des Trägers. Wie in Abb. 4 gezeigt, wurde die Zellproliferation durch GS, Apt-Apt und Apt-Apt/siRNA bei Konzentrationen von 100–600 µg/ml Gruppen (Zelllebensfähigkeit> 80 %) nicht merklich beeinflusst und die Zytotoxizität trat dosiert auf -abhängig. Im Vergleich zu bloßen GS-NGs hatten AS1411-modifizierte Nanogele aufgrund der Hemmung des NF-κB-Signalwegs [27, 30] und der Zerstörung von Organellenmembranen [16] eine leichte Abnahme der Zelllebensfähigkeit. Die gutartige Biokompatibilität von Apt-GS-Nanogelen macht sie als siRNA-Träger in vivo geeignet.
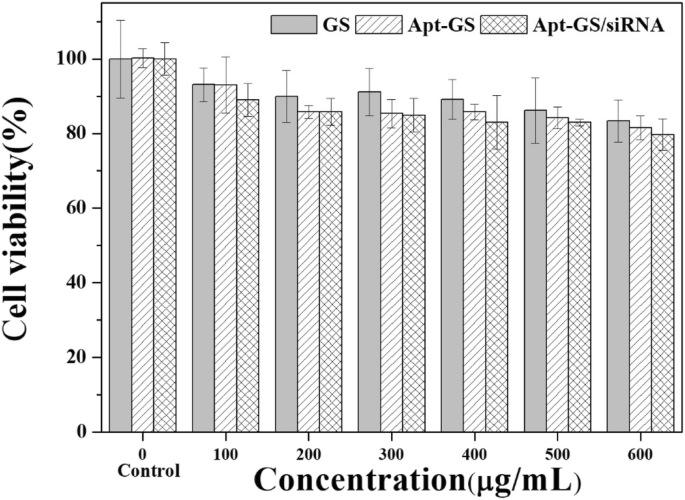
Zelllebensfähigkeit von A549-Zellen mit GS-, Apt-GS- und Apt-GS/siRNA-Nanogelen verschiedener Konzentrationen, bestimmt durch MTT-Assay
Um das Targeting von Apt-GS in Tumorzellen in vitro zu beurteilen, inkubierten wir A549-Zellen und 3T3-Fibroblasten mit den gleichen Mengen an FAM-markiertem Apt-GS oder Kontroll-TDO-GS (GS dekoriert mit einem Kontroll-Oligonukleotid). In 3T3-Zellen oder wenn die Zellen mit Kontroll-TDO-GS behandelt wurden, konnten geringe Fluoreszenzniveaus beobachtet werden (Fig. 5b, c). Im Gegensatz dazu war die von Aptamer AS1411 emittierte grüne Fluoreszenz in A549-Krebszellen deutlich verstärkt ( 5a ), was darauf hindeutet, dass Apt-GS aufgrund der Einführung der auf Nukleolin gerichteten AS1411-Aptamere spezifisch in Tumorzellen akkumuliert wurde. In der quantitativen Analyse durch Durchflusszytometrie (Abb. 6) zeigten Apt-GS-NGs zeitabhängige Fluoreszenzintensitäten in beiden Zelltypen. Von 0,5 auf 16 h verbesserte sich die Fluoreszenzintensität der Internalisierung von Apt-GS in A549-Zellen und 3T3-Zellen um das 13- bzw. 2-fache. Das Zellverhältnis, das NGs enthielt, stieg in den ersten 30 Minuten schnell an und erreichte anscheinend das Maximum der möglichen Sättigung nach 8 Stunden in A549.96 % der A549-Zellen waren innerhalb von 16 Stunden angelagert oder nahmen die Nanogele ein, während nur 52 % der 3T3-Zellen wurde beobachtet. Um die Rolle von Nukleolin bei der zellulären Aufnahme von Nanogelen zu evaluieren, wurde die Mischung der präparierten Apt-GS NGs und freien AS1411-Aptameren unterschiedlicher Konzentrationen (0,5, 5 und 50 µM) mit A549-Zellen für 8 Stunden inkubiert. Die Fluoreszenzintensität und der Prozentsatz der Zellen, die Nanogele enthalten, nahmen um 50 % bzw. 30 % ab, wenn das freie AS1411-Aptamer verwendet wurde, um mit den Apt-GS-NGs um an der Zellmembran exprimiertes Nukleolin zu konkurrieren, was auf eine Nukleolin-induzierte Rezeptor-vermittelte Endozytose schließen lässt ( Abb. 6c). Diese Daten zeigten, dass Apt-GS NGs über eine Nukleolin-vermittelte Internalisierung eine höhere Akkumulation in A549-Krebszellen aufwiesen.
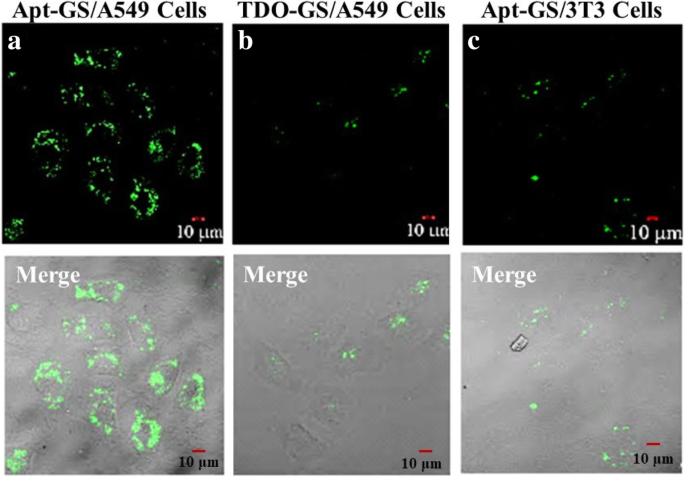
a –c Zellaufnahme von FAM-siRNA, die von Apt-GS- und TDO-GS-Nanogelen geliefert wird. Die Bilder wurden mit CLSM aufgenommen. Grünes Signal:FAM. Maßstabsleiste ist 10 μm
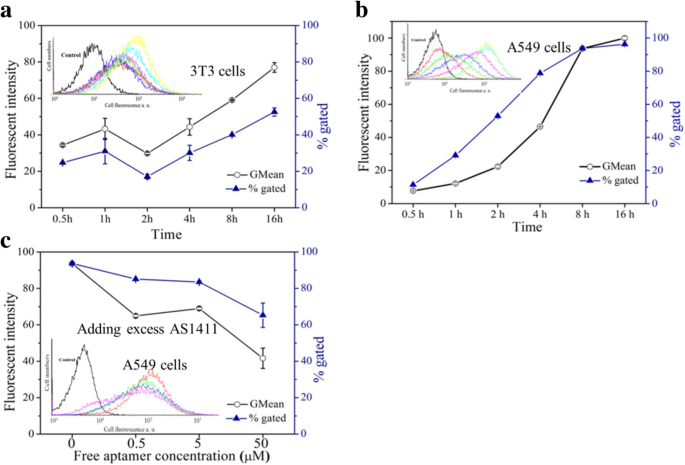
Durchflusszytometrie-Analyse (FCMS) der Zellaufnahme von FAM-markierten Apt-GS- und TDO-GS-Nanogelen in verschiedenen Zelltypen. Einfügen:FACS-Profile, a , b Kurve rot, grün, blau, lila, aqua und gelb zeigen die Inkubationszeit von 0,5 bis 16 h; c Kurve rot, grün, blau und lila bedeuten die Konzentration von AS1411 von 0 bis 16 μM
RNA-Interferenz
Es wird berichtet, dass der Einbau von Desoxynukleotiden in siRNA die Stabilität des Sense-Strangs erhöhen und eine Wirksamkeit von 40% der RNAi aufrechterhalten kann [2, 3]. Um die Knockdown-Effizienz von Test-siRNA-Trägern zu bewerten, verwendeten wir in beiden Zellen ein Ribonukleotid-ersetztes Luciferase-Reportergensystem. Die Gen-Knockdown-(KD)-Effizienz von siRNA wurde anhand der Luciferase-Aktivität nach 48 Stunden bestimmt. Wie in 7a gezeigt, verbessert die Apt-GS/siRNA die 4,6-fache Gen-Knockdown-Effizienz von A549-Zellen im Vergleich zu 6% von normalen 3T3-Zellen, was eine gute Zellselektivität zeigt. Dies deutete darauf hin, dass die Modifikation des Aptamers AS1411 die Transfektionseffizienz durch Erzeugung eines Targeting-Effekts erhöhte. Es wurde nachgewiesen, dass kationische Liposomen die Frachtabgabe unterstützen und aus Lysosomen entweichen [31]. Darüber hinaus trug die nachfolgende Zugabe von Liposomen (A/si-GS →Lip-Gruppe) nicht dazu bei, die siRNA-vermittelte Gen-Silencing-Aktivität (30,4%) trotz Endosomenunterbrechung zu verbessern, was auf die siRNA-Freisetzung in der intrazellulären Umgebung von Glutathion (GSH) durch Disulfidspaltung schließen ließ . Außerdem verbesserte die Co-Transfektion von Liposomen und Apt-GS/siRNA (A/si-GS + Lip-Gruppe) leicht die Knockdown-Effizienz der Luciferase-Expression (41,6 %) in A549, obwohl Liposomen das Entweichen von Lysosomen unterstützten, in Übereinstimmung mit früheren Berichten [27 , 30]. Allerdings hatten A549-Zellen im Vergleich zu 3T3-Zellen eine 2,7- bzw. 1,4-fache Gen-Silencing-Aktivität für die A/si-GS→Lip-Gruppe bzw. für A/si-GS+Lip, was auf eine Abnahme der Zellselektivität schließen lässt.
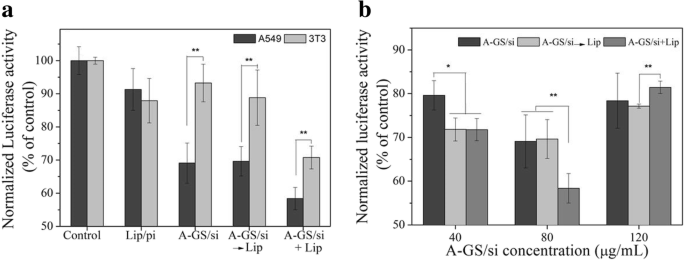
In-vitro-Silencing-Effekt von Anti-Luciferase-siRNA, formuliert in Apt-GS-Nanogelen mit verschiedenen Behandlungen (a ) und Konzentration von Apt-GS/siRNA-Komplexen (A-GS/si) auf Gen-Silencing (b )
Als nächstes untersuchten wir den Konzentrationseffekt von Nanogelen auf die RNAi. Wie in Fig. 7b gezeigt, erreichte die Geninterferenzeffizienz das Maximum bei der Komplexkonzentration von 80 µg/ml. Die verminderte KD-Effizienz bei einer Konzentration von 120 µg/ml kann auf eine erhöhte Zytotoxizität von Aptamer zurückzuführen sein. Es wird auch darauf hingewiesen, dass die Zugabe eines Endosomen-Escape-Agent-Liposoms die RNA-Interferenzeffizienz bei niedrigen Konzentrationen von Apt-GS/siRNA-Nanogelen (40 µg/ml und 80 µg/ml) erhöhen kann, jedoch bei hohen Konzentrationen (120 µg/ml) nicht zu einer Verringerung führte μg/ml). Daher wird angenommen, dass Apt-GS erfolgreich siRNA an die Tumorzellen abgeben und anschließend zu spezifischer RNAi führen kann.
Schlussfolgerungen
Die Stabilität und gewebespezifische Abgabe von siRNA bleiben die größten Hindernisse für RNAi-Therapeutika. Hier wird Desoxynukleotid-substituierte siRNA verwendet, um die Stabilität der siRNA zu verbessern, und der GS-Kern wurde weiter mit AS1411-Aptamer für das Tumor-Targeting und Disulfid-Linker für die redox-responsive Abgabe von siRNA beschichtet. Das Vehikel hat eine kugelförmige Struktur mit einer Größe von ca. 200 nm und besitzt positive Ladungen an der Oberfläche. Diese so hergestellten Apt-GS/siRNA-Nanogele konnten die Ladung schützen und zeigten eine beschleunigte siRNA-Freisetzung unter DTT-Addition durch Disulfidspaltung. Darüber hinaus konnten die neuen Apt-GS-Nanogele Apt-GS mit dem zielgerichteten Aptamer AS1411 effektiv mehr Fracht an das Zytoplasma abgeben und freisetzen, was zu einer signifikanten (~~5-fach in vitro-Verbesserung der Stille-Aktivität in Nukleolin-überexprimierenden A549-Zellen) führt. Insgesamt deuten diese Ergebnisse darauf hin, dass die Abgabe von Desoxynukleotid-substituierter siRNA eine innovative Strategie ist und diese redoxresponsiven, auf Tumoren abzielenden intelligenten Nanogele viel versprechend für die siRNA-Abgabe und Tumortherapie sind.
Verfügbarkeit von Daten und Materialien
Die Datensätze, die die Schlussfolgerungen dieses Artikels unterstützen, sind im Artikel enthalten.
Abkürzungen
- APTMS:
-
3-Aminopropyl-trimethoxysilan
- DMEM:
-
Dulbecco's modifiziertes Eagle's Medium
- DMSO:
-
Dimethylsulfoxid
- FAM:
-
Fluoresceinamidit
- GE:
-
Gelelektrophorese
- GPSM:
-
3-Glycidoxypropyl-trimethoxysilan
- GS-NGs:
-
Siloxan-vernetzte Gelatine-Nanogele
- MTT:
-
3-(4,5-Dimethylthiazolyl-2)-2,5-diphenyltetrazoliumbromid
- RNAi:
-
RNA-Interferenz
- siRNAs:
-
Kleine störende RNAs
- SPDP:
-
N -Succinimidyl-3-(2-pyridyldithio)-propionat
- TDO:
-
Negatives DNA-Oligonukleotid
Nanomaterialien
- Nanofasern und Filamente für eine verbesserte Wirkstoffabgabe
- Umweltverträgliche Metall-organische Gerüste als Wirkstoffabgabesystem für die Tumortherapie
- Zellbasierte Arzneimittelabgabe für Krebsanwendungen
- Zebrafisch:Ein vielversprechendes Echtzeit-Modellsystem für die nanotechnologisch vermittelte neurospezifische Wirkstoffabgabe
- 131I-gespurte PLGA-Lipid-Nanopartikel als Wirkstoffträger für die gezielte chemotherapeutische Behandlung von Melanomen
- Eine neue Methode ohne organisches Lösungsmittel zu zielgerichtetem Nanowirkstoff für eine verbesserte Wirksamkeit bei Krebs
- Silica-Nanopartikel für die intrazelluläre Proteinzufuhr:ein neuartiger Syntheseansatz unter Verwendung von grün fluoreszierendem Protein
- Drei Grundlagen der Routenplanung für große Einzelhändler
- Versand am selben Tag aus dem Geschäft? Fünf Tipps für eine schnellere Lieferung
- Die Zukunft der kontaktlosen Lieferung



