M1-Makrophagen-abgeleitete exosomale MicroRNA-326 unterdrückt die Progression von hepatozellulären Karzinomzellen über die Vermittlung des NF-κB-Signalwegs
Zusammenfassung
Immer mehr Beweise haben gezeigt, dass microRNA (miR), die aus M1-Makrophagen-abgeleiteten Exosomen stammt, das Fortschreiten des hepatozellulären Karzinoms (HCC) regulieren kann. Die Wirkung von miR-326, das von M1-Makrophagen-abgeleiteten Exosomen abgeleitet ist, auf HCC wurde jedoch nicht berichtet. Daher war das Ziel der vorliegenden Studie, den Mechanismus von exosomalem miR-326 aus M1-Makrophagen bei der Regulierung der HCC-Zellprogression zu untersuchen. RT-qPCR wies die Expression von miR-326 in HCC-Zelllinien nach. Die miR-326-Expression in HCC wurde durch Transfektion verändert und die Wirkung von miR-326 auf CD206- und NF-κB-Expression, Zellproliferation, Koloniebildung, Migration, Apoptose und Invasion wurde nachgewiesen. Anschließend wurden Exosomen aus M1-Makrophagen isoliert. RT-qPCR identifizierte die miR-326-Expression in M1-Makrophagen-abgeleiteten Exosomen. Die miR-326-Expression in M1-Makrophagen-abgeleiteten Exosomen wurde durch Transfektion verändert. Von M1-Makrophagen abgeleitete Exosomen wurden zusammen mit HCC-Zellen kultiviert, um ihre Auswirkungen auf den biologischen Fortschritt von HCC-Zellen herauszufinden. Schließlich wurden in-vivo-Experimente durchgeführt, um die in-vitro-Ergebnisse zu überprüfen. MiR-326 war in HCC-Zellen verringert und in M1-Makrophagen-abgeleiteten Exosomen angereichert. Die Hochregulierung von miR-326 würde die HCC-Zellproliferation, Koloniebildung, Migration, Invasion und CD206- und NF-κB-Expression hemmen und die Apoptose fördern und das Wachstum von HCC-Tumoren in vivo hemmen , während die Herunterregulierung von miR-326 gegenteilige Effekte zeigte. Von M1-Makrophagen abgeleitete Exosomen hemmten die HCC-Zellproliferation, Koloniebildung, Migration, Invasion und CD206- und NF-κB-Expression und verstärkten die Apoptose, während die Überexpression von miR-326 die Wirkung von M1-Makrophagen-abgeleiteten Exosomen auf HCC-Zellen verstärkte. Es wurde gezeigt, dass das von M1-Makrophagen abgeleitete exosomale miR-326 die Proliferation, Migration und Invasion unterdrückt sowie die Apoptose von HCC durch Herunterregulieren der NF-κB-Expression fördert.
Einführung
Das hepatozelluläre Karzinom (HCC) ist weltweit der fünfthäufigste Krebs und der häufigste primäre Leberkrebs [1]. Die Daten des China National Cancer Registry zeigen, dass die Sterblichkeitsrate von primärem Leberkrebs an dritter Stelle steht, während die Inzidenz an vierter Stelle unter den häufigen malignen Erkrankungen steht [2]. Die Hauptrisikofaktoren für HCC sind chronische Infektionen mit dem Hepatitis-C-Virus und Hepatitis-B-Virus, aflatoxinbelastete Lebensmittel, Übergewicht, Rauchen, starker Alkoholkonsum und Typ-2-Diabetes [3]. Die transarterielle Chemoembolisation ist eine etablierte Behandlung des HCC im Zwischenstadium, die das Überleben bei der Mehrheit der HCC-Patienten im Zwischen- oder fortgeschrittenen Stadium verbessert [4]. Derzeit hängt die Diagnose des HCC hauptsächlich von Serum-Biomarkern und bildgebenden Verfahren ab [5]. Die HCC-bedingte 5-Jahres-Überlebensrate beträgt nur etwa 60 %, und die Inzidenz steigt in den letzten Jahren von Jahr zu Jahr [6]. Angesichts dessen hat die Erforschung eines genauen therapeutischen Ziels bei der Behandlung von HCC Priorität.
Makrophagen sind Effektorzellen und wichtige Regulatoren des Immunsystems, üben enorme Funktionen bei der Gewebeumbildung und -reparatur aus und führen in fast allen Geweben in vivo orchestrierte Stoffwechselfunktionen aus [7]. Es zeigt sich, dass M1-Makrophagen tumorfördernde Effekte ausüben und die Motilität von HCC-Zellen erhöhen [8]. Das Exosom ist ein scheibenförmiges Vesikel mit einem Durchmesser von 40–150 nm [9]. Nach Xu et al. haben exosomale microRNAs (miRNAs) über die Modulation der Genexpression in den Zielzellen Funktionen auf die Proliferation, Invasivität, Metastasierung und Arzneimittelresistenz von HCC [10]. Eine Studie hat gezeigt, dass miR-326 enthaltende Exosomen ein potenzielles klinisches Ziel bei Multipler Sklerose sein könnten [11]. MiRNAs könnten entweder als Onkogene oder als Tumorinhibitoren wirken, indem sie eine Vielzahl von Protein-kodierten Genexpressionen durch mRNA-Abbau und Translationsblockade in einer bestimmten Sequenz modulieren [12]. In einer Studie wurde diskutiert, dass miR-326 das Wachstum von HCC-Zellen durch eine Störung des Zellzyklusverlaufs sowie durch eine Verbesserung der Apoptose unterdrückt, außerdem die Zellinvasion durch eine Verringerung des epithelial-mesenchymalen Übergangsphänotyps unterdrückt [13]. Eine andere Studie hat berichtet, dass miR-326 ein potenzielles therapeutisches Ziel für die Behandlung von HCC-Patienten sein könnte [14]. Daher diskutierte diese vorliegende Studie den Mechanismus von exosomalem miR-326, der die Invasion und Migration von HCC-Zellen reguliert.
Materialien und Methoden
Ethikerklärung
Alle Tierversuche entsprachen dem Guide for the Care and Use of Laboratory Animal by International Committees. Das Protokoll wurde vom Institutional Animal Care Use Committee des Third Hospital of Jilin University genehmigt.
Induktion und Identifizierung von Makrophagen
Die humane monozytäre Zelllinie THP-1 (Kunming Institute of Zoology, CAS, Kunming, China) wurde in RPMI 1640-Medium (Gibco, CA, USA; Thermo Fisher Scientific, MA, USA) mit 10 % hitzeinaktiviertem fötalem Rinderserum ( FBS). THP-1-Zellen wurden mit 150 ng/ml Phorbol-12-myristat-13-acetat (PMA; P8139, Sigma-Aldrich, SF, CA, USA) umgesetzt und 24 h in RPMI-Medium inkubiert, um M0-Makrophagen zu erhalten. Dann wurde die Zellmorphologie vor und nach der Induktion durch Wright-Färbung beobachtet. THP-1-Zellen und induzierte Makrophagen wurden in 5 &mgr;l PBS resuspendiert, auf den Glasobjektträger getropft, mit Wright-Farblösung gefärbt und mit der Pufferlösung im Verhältnis 1:2 gemischt. 10 min gefärbt und mit fließendem Wasser gespült, wurden die Zellen unter dem Mikroskop beobachtet. Darüber hinaus wurden M0-Makrophagen-Marker (CD68 und CD206) durch quantitative Polymerase-Kettenreaktion (RT-qPCR) der reversen Transkription gemessen. Als nächstes wurden Makrophagen in M1-Makrophagen durch Inkubation mit 20 ng/ml Interferon (IFN)-γ (#285-IF; R&D Systems, MN, USA) und 10 pg/ml LPS (#8630; Sigma-Aldrich) für 18 . induziert h. M1-Makrophagenmarker (IDO1 und IL-12 p35) wurden durch RT-qPCR untersucht [15].
Extraktion von Exosomen
Die Exosomen wurden mit einem Exosomen-Separation-Kit (ExoEasy Maxi Kit, Qiagen, Hilden, Deutschland) extrahiert. Der Makrophagenüberstand wurde unter aseptischen Bedingungen in einem 15-ml-Zentrifugenröhrchen angehäuft und mit einem 0,8-μM-Filterfilm filtriert. Der Zellüberstand in jeder Gruppe wurde mit XBP-Puffer (1:1) angehängt und dann mit einer exoEasy-Membranaffinitätszentrifugationssäule bei 500 g zentrifugiert . Die Zellen wurden mit 10 ml XWP-Puffer ergänzt und mit 3000–5000g . zentrifugiert . Die exoEasy-Membran-Affinitätszentrifugationssäule wurde mit 400 μl XE-Eluierungspuffer ausgebrütet und bei 500 g . zentrifugiert . Der Elutionspuffer wurde auf eine exoEasy-Membran-Affinitätszentrifugationssäule übertragen und bei 500g . zentrifugiert . Der Elutionspuffer wurde 24 h bei 4 °C gelagert und dann zur Identifizierung verwendet. Der Rest wurde bei − 80 °C gelagert.
TEM-Beobachtung und Nanopartikel-Tracking-Analyse (NTA)
Die oben erhaltenen Exosomen wurden in das kohlenstoffgestützte Membrankupfernetz getropft und mit 2% Phosphorwolframsäure versetzt. Die Probe wurde unter einem TEM beobachtet und das optimale Bild wurde gesammelt und analysiert.
Die Verunreinigungen und Partikel in PBS wurden durch einen 0,22-μM-mikroporösen Filter entfernt. Entsprechend der Partikeldichte der Exosomen wurde das gefilterte PBS auf die entsprechende Konzentration verdünnt und mit einem Nanosight NS300 Nanopartikeldetektor (Malvern, Westborough, MA, USA) nachgewiesen.
Nach der Identifizierung wurden von Makrophagen abgeleitete Exosomen, transfiziert mit miR-326-Inhibitor und miR-326-Inhibitor-Negativkontrolle (NC), miR-326-Mimic und miR-326-Mimic-NC mit dem Exosomen-Separation-Kit (Invitrogen) extrahiert.
RT-qPCR
Die Gesamt-RNA wurde mit Trizol Reagent (Thermo Fisher) extrahiert und die Real-Time PCR wurde mit dem SYBR-Green PCR Master Mix (Roche) und dem ABI 7500 Real-Time PCR System (Life Technologies, Grand Island, NY, USA) durchgeführt ). Die Primersequenzen sind in Tabelle 1 aufgeführt. Die quantitative Analyse erfolgte nach der Methode 2 –△△Ct .
Western Blot-Analyse
Das Gesamtprotein von Zellen und Exosomen wurde extrahiert. Die Proteinkonzentration wurde mit einem Bicinchoninsäure (BCA)-Kit (Boster Biological Technology Co. Ltd., Wuhan, Hubei, China) bestimmt. Das Protein wurde mit Probenpuffer angehängt und bei 95 °C gekocht, und jede Vertiefung wurde mit 30 ug beladen. Das Protein wurde mit 10% Polyacrylamid-Gelelektrophorese (Boster Biological Technology) aufgetrennt und auf eine Polyvinylidenfluorid-Membran elektrogeblottet, gefolgt von einer Versiegelung in 5% Rinderserumalbumin (BSA). Die Membran wurde mit primärem Antikörper gegen CD63 (1:1000, Developmental Studies Hybridoma Bank, University of Iowa, Ames, IA, USA), CD181 (1:1000, R&D Systems), GAPDH (1:2000, Jackson ImmunoResearch Laboratories, PA, USA) und mit dem sekundären Antikörper (1:500, Jackson ImmunoResearch Laboratories) markiert durch Meerrettichperoxidase. Die Bilder wurden mit dem Dual-Color-Infrarot-Fluoreszenz-Scan-Bildgebungssystem von Odyssey erhalten, und die Grauwerte der Bänder wurden mit der Bildanalysesoftware Quantity One gemessen.
Zellkultur und Screening
Die normale Leberzelllinie HL-7702 und die humane HCC-Zelllinie BEL-7404, HepG2, SMMC-7721 und QGY-7703 wurden ausgewählt und in Gibco RPMI Media 1640 mit 10 % fötalem Rinderserum (FBS), Penicillin (100 U/ml .) kultiviert ) und Streptomycin (100 mg/ml). Die MiR-326-Expression wurde durch RT-qPCR nachgewiesen und die geeigneten Zelllinien wurden gescreent.
Exosomen-Kennzeichnung und Aufnahme von Exosomen
Die Exosomen wurden mit 250 μL Verdünnungsmittel C resuspendiert und vorsichtig verrieben, um eine Beschädigung der Exosomenmembran zu vermeiden. Der Farbstoff PKH67 (1 µl, Sigma-Aldrich) wurde zu 250 µl Verdünnungsmittel C auf 500 µl gegeben und inkubiert. Die Lösung wurde mit 500 µl 1% BSA versetzt und 1 min bei 37 °C inkubiert. Die Exosomen wurden durch Zentrifugation bei 120.000g . gewonnen 4 °C für 2 h. Die mit PKH67 markierten Exosomen wurden durch Zentrifugation bei 120.000g . gewonnen 4 °C für 2 h. Die Exosomen wurden mit 6 ml RPMI-1640-Medium unter Vermeidung von Licht resuspendiert. Dann wurden die markierten Exosomen mit HCC-Zellen für 12 h cokultiviert. Danach wurde das Kulturmedium entfernt und mit PBS 3 mal 5 min/Zeit gewaschen, und die fluoreszenzmarkierten Exosomen, die nicht intern von HCC-Zellen absorbiert wurden, wurden gründlich abgewaschen. Die Exosomen wurden mit 4% Paraformaldehyd befestigt und mit 4'-6-Diamidino-2-phenylindol gefärbt. Nach dem Versiegeln wurde die Fluoreszenzverteilung mit einem konfokalen Lasermikroskop beobachtet.
Zellgruppierung und -behandlung
HepG2-Zellen und SMMC-7721-Zellen wurden in der 12-Well-Platte bei 0,5–1 × 10 6 . ausgesät Zellen/Well. Bei 50–60% Konfluenz wurden die Zellen mit Lipofectamine 2000 (Invitrogen, Carlsbad, CA) transfiziert. HepG2-Zellen wurden in eine miR-326-nachahmende Gruppe (transfiziert mit miR-326-Nachahmer) und NC-nachahmende Gruppe (transfiziert mit miR-326 nachahmendem NC) verteilt. SMMC-7721-Zellen wurden in eine miR-326-Inhibitor-Gruppe (transfiziert mit miR-326-Inhibitor) und NC-Inhibitor-Gruppe (transfiziert mit miR-326-Inhibitor NC) eingeteilt. miR-326-mimic, miR-326-Inhibitor und ihre NCs wurden zur Transfektion mit Lipofectamine 2000 gemischt. HepG2-Zellen und SMMC-7721-Zellen ohne jegliche Behandlung wurden als leere Gruppe festgelegt. miR-326-mimic, miR-326-Inhibitor und ihr NC wurden von Guangzhou RibBio Co., Ltd. (Guangzhou, China) entwickelt und zusammengestellt (Tabelle 1).
Kokultur von M1-Makrophagen-abgeleiteten Exosomen mit HCC-Zellen
Die Proteinkonzentration der M1-Makrophagen-abgeleiteten Exosomensuspension wurde durch die BCA-Methode bestimmt und das Volumen der entsprechenden Exosomensuspension mit 50 μg Protein wurde berechnet. HepG2-Zellen und SMMC-7721-Zellen wurden in einer 12-Well-Platte bei 1 × 10 5 . ausgesät Zellen/ml pro Vertiefung. HepG2-Zellen wurden in 4 Gruppen verteilt:Kontrollgruppe (HepG2-Zellen, die nicht mit Exosomen kokultiviert wurden), Exosomen (Exo)-Gruppe (HepG2-Zellen, die mit M1-Makrophagen-abgeleiteten Exosomen kokultiviert wurden), Exo-miR-326-nachahmende Gruppe (HepG2 .) Zellen, die mit M1-Makrophagen-abgeleiteten Exosomen kokultiviert wurden, die mit miR-326-Nachahmer transfiziert wurden), Exo-NC-Nachahmungsgruppe (HepG2-Zellen, co-kultiviert mit M1-Makrophagen-abgeleiteten Exosomen, die mit miR-326-Nachahmer-NC transfiziert wurden). SMMC-7721-Zellen wurden auch in 4 Gruppen eingeteilt:Leergruppe (SMMC-7721-Zellen, die nicht mit Exosomen kokultiviert wurden), Exo-Gruppe (SMMC-7721-Zellen, die mit M1-Makrophagen-abgeleiteten Exosomen kokultiviert wurden), Exo-miR-326- Inhibitor-Gruppe (SMMC-7721-Zellen, co-kultiviert mit M1-Makrophagen-abgeleiteten Exosomen, die mit miR-326-Inhibitor transfiziert wurden), Exo-NC-Inhibitor-Gruppe (SMMC-7721-Zellen, co-kultiviert mit M1-Makrophagen-abgeleiteten Exosomen, die mit miR- 326 Inhibitor NC).
3-(4,5-Dimethylthiazol-2-yl)-2, 5-Diphenyltetrazoliumbromid (MTT)-Assay
Die Zellen wurden mit Trypsin abgelöst und auf 96-Well-Platten mit einer Zelldichte von 4 × 10 4 . ausgesät Zellen pro Vertiefung. Das Kulturmedium wurde nach 12, 24, 36, 48 bzw. 60 Stunden Kultivierung aufgegeben. Inkubiert mit 500 µL 0,5 g/L MTT-Lösung wurden die Zellen mit 200 µL Dimethylsulfoxid-Lösung versetzt, verrieben und ausgebrütet. Die Werte der optischen Dichte (OD, 490 nm) wurden mit einem Mikroplatten-Lesegerät gemessen.
Kolonie-Bildungs-Assay
24 h kultiviert und mit Trypsin abgelöst, wurden die Zellen in eine 35 mm kleine Schale mit 300 Zellen pro Schale ausgesät. Die Lösung wurde alle 3 d ersetzt. Nach 10 Tagen Kultur wurden die Zellen mit 40 g/L -1 . fixiert Paraformaldehyd und gefärbt mit 1 g/L −1 Kristallviolettlösung und getrocknet. Koloniezahl (mehr als 50 Zellen) wurde unter einem Mikroskop berechnet.
Transwell-Assay
Zellen (1 × 10 5 ) wurden mit 200 µL Leernährboden suspendiert. Die Experimente wurden in Übereinstimmung mit den Anweisungen der Transwell-Kammer (Corning Glass Works, Corning, N.Y., USA) durchgeführt (Matrigel wurde für das Invasionsexperiment benötigt, aber nicht für das Migrationsexperiment). RPIM 1640 (10% FBS, 600 &mgr;l) wurde in die untere Kammer gegeben. Die obere und untere Kammer wurden durch eine mit Matrigel vorbeschichtete Transwell-Membran (BD Biosciences, Franklin Lakes, NJ, USA) getrennt. 24 h kultiviert, wurde die Kammer mit 95 % Alkohol fixiert. Nach der Färbung mit Kristallviolettlösung wurden die Zellen in fünf Gesichtsfeldern unter dem Mikroskop beobachtet.
Durchflusszytometrie
Zellzyklus:Zellen wurden durch Trypsin abgelöst. Zellen (1 × 10 6 ) wurden mit 0,5 ml PBS suspendiert und zu einer Einzelsuspension verrieben. Vermischt mit 4,5 ml vorgekühltem 70 % Ethanol auf Eis wurden die Zellen bei 3000g . zentrifugiert , mit 5 mL PBS gespült und erneut bei 3000g . zentrifugiert . Anschließend wurden die Zellen mit 1 ml PI/Triton X-100 Färbelösung (20 µg PI/0,1% Triton X-100) enthaltend 0,2 mg RnaseA suspendiert. Der Zellzyklus wurde durch Durchflusszytometrie nachgewiesen.
Zellapoptose:Trypsinisierte Zellen (1 × 10 6 ) wurden mit 1 mL PBS suspendiert, verrieben und bei 3000g . zentrifugiert . Die Zellen wurden wiederum mit Inkubationspuffer (10 mmol/L Hepes/NaOH, pH 7,4, 140 mmol/L NaCl, 5 mmol/L CaCl2 . gespült ) und zentrifugiert bei 3000g . Als nächstes wurden die Zellen mit 100 μl Markierungslösung inkubiert (FITC-Annexin V und PI wurden dem Inkubationspuffer hinzugefügt, um 1 μg/ml zu erreichen), zentrifugiert bei 3000 g , einmal mit Inkubationspuffer gewaschen und mit fluoreszierender (SA-FLOUS) Lösung ausgebrütet. Die Zellapoptose wurde durch Durchflusszytometrie nachgewiesen. Die Wellenlänge der Durchflusszytometrie betrug 488 nm, und die FITC-Fluoreszenz wurde durch einen Bandfilter bei 515 nm detektiert, während PI mit einer Wellenlänge von mehr als 560 nm gemessen wurde. Die Ergebnisse wurden automatisch per Computer analysiert.
Tumor-Xenotransplantat bei Nacktmäusen
Vierzig Mäuse (College of Animal Science, Jilin University, Jilin, China) im Alter von 4–6 w wurden zufällig in 8 Gruppen mit 5 Mäusen in jeder Gruppe verteilt. Die Mäuse wurden 1 Woche lang in einem speziellen pathogenfreien Tierlabor gefüttert und Futter, Kissen und Wasserflasche wurden rechtzeitig ersetzt. Der Gesundheitszustand von Mäusen sollte täglich beobachtet werden. Eine Woche später wurden HCC-Zellen in Zellsuspension präpariert und mit 0,1 ml Zellsuspension (1 × 10 6 .) subkutan in Nacken und Rücken injiziert ). Das Wachstum des Tumors wurde nach 3–5 d beobachtet. Die Nacktmäuse wurden alle 4 Tage gewogen und das Tumorvolumen gemessen. Die Nacktmäuse wurden 20 Tage nach der Injektion euthanasiert.
Statistische Analyse
Alle Daten wurden mit der Software SPSS 17.0 (SPSS Statistics, Chicago, IL, USA) interpretiert. Die Messdaten wurden als Mittelwert ± Standardabweichung angegeben. Vergleiche zwischen zwei Gruppen wurden von der t . formuliert -Test, während Vergleiche zwischen mehreren Gruppen durch Einweg-Varianzanalyse (ANOVA) bewertet wurden. P Wert < 0,05 war ein Hinweis auf einen statistisch signifikanten Unterschied.
Ergebnisse
Identifizierung von M1-Makrophagen und -Exosomen
Die Wright-Färbung wurde verwendet, um die Morphologie der durch PMA induzierten THP-1-Zellen zu beobachten. Es zeigte sich, dass das Volumen der THP-1-Zellen vor der Induktion klein und der Anteil an Karyoplasma höher war; die Morphologie der Zellen nach der Induktion war unregelmäßig, das Volumen wurde größer und der Anteil des Karyoplasmas nahm ab; das Zytoplasma war reicher und hellblau, mit reichlich Partikeln und einigen Vakuolen; der Zellkern war purpurrot und oft zur Seite geneigt, was zeigt, dass die Zellen die typischen morphologischen Merkmale von Makrophagen aufwiesen (Abb. 1a).
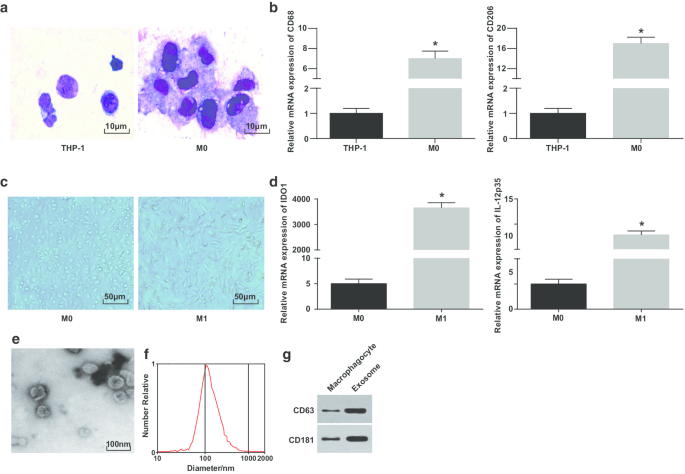
Identifizierung von M1-Makrophagen und Exosomen. a Wright-Färbung zur Beobachtung der Morphologie von THP-1-Zellen vor und nach der PMA-Behandlung. b CD68- und CD206-Expression in THP-1-Zellen und PMA-behandelten THP-1-Zellen, nachgewiesen durch RT-qPCR; c Morphologie von Makrophagen und M1-Makrophagen. d Expression von IDO1 und IL-12 p35 in M0-Makrophagen und LPS- und INF-γ-behandelten M0-Makrophagen, nachgewiesen durch RT-qPCR. e TEM zur Exosomenbeobachtung. f Nachweis der Partikelgrößenverteilung von Exosomen durch NTA. g Proteinbanden von CD63 und CD181. In Feld b, *P < 0,05 gegenüber THP-1-Zellen; In Feld d, *P < 0,05 gegenüber M0-Makrophagen. Die Messdaten wurden als Mittelwert ± Standardabweichung (N = 3), Vergleiche zwischen zwei Gruppen wurden durch t-Test durchgeführt
Um die erfolgreiche Induktion von Makrophagen weiter zu verifizieren, wurde die Expression von CD68 und CD206 vor und nach der Induktion durch RT-qPCR getestet. Es zeigte sich, dass die Expression von CD68 und CD206 seit der PMA-Induktion erhöht war, was anzeigt, dass die PMA erfolgreich THP-1-Zellen in M0-Makrophagen induzierte ( 1b ). Dann wurden M0-Makrophagen durch Induktion von LPS und INF-γ in M1-Makrophagen polarisiert. Die Morphologie und die M1-Typ-Makrophagen-Oberflächenmarker IDO1 und IL-12 p35-Expression von Makrophagen wurden beobachtet und getestet. M0-Makrophagen zeigten verschiedene und unregelmäßige adhärente Morphologien mit runder, elliptischer oder spindelförmiger Form. Die Morphologie von Makrophagen, die durch IFN-γ stimuliert wurden, zeigte sich mit mehr Pseudopodien und Vorsprüngen und Fusiformis (Abb. 1c). RT-qPCR zeigte (Abb. 1d), dass M1-Makrophagen nach LPS- und INF-γ-Behandlung einen Anstieg ihrer Marker (IDO1 und IL-12p35) zeigten.
Anschließend wurden die von Makrophagen stammenden Exosomen mit dem TEM beobachtet. Es zeigte sich, dass die von Makrophagen stammenden Exosomen reich und die Form rund oder oval war, mit membranöser Struktur, einheitlicher Größe und weniger Schadstoffen (Abb. 1e). NTA zeigte, dass Exosomen mit einem zentralisierten Peak der MODE-Kurve und glatter Linearität einen konzentrierteren Durchmesser und weniger Verunreinigungen aufwiesen ( 1f ). Western-Blot-Analysen berichteten, dass im Vergleich zu Makrophagen die Expression der spezifischen Markerproteine CD63 und CD181 in Exosomen, die von Makrophagen stammten, erhöht war ( 1g ). Diese Ergebnisse zeigen, dass wir erfolgreich Monozyten zur Differenzierung in Makrophagen induziert und sie in M1-Makrophagen polarisiert haben.
M1-Makrophagen-abgeleitete Exosomen liefern miR-326 an HCC-Zellen und beeinflussen die miR-326-Expression in HCC-Zellen
Um zu überprüfen, ob von M1-Makrophagen abgeleitete Exosomen miR-326 zu HCC-Zellen transportierten, wurden HepG2- und SMMC-7721-Zellen zusammen mit Exosomen kultiviert. Es konnte erkannt werden, dass eine große Anzahl von Exosomen nach 4-stündiger Transfektion unter dem Fluoreszenzmikroskop von HepG2- und SMMC-7721-Zellen assimiliert wurde (Abb. 2a, b).
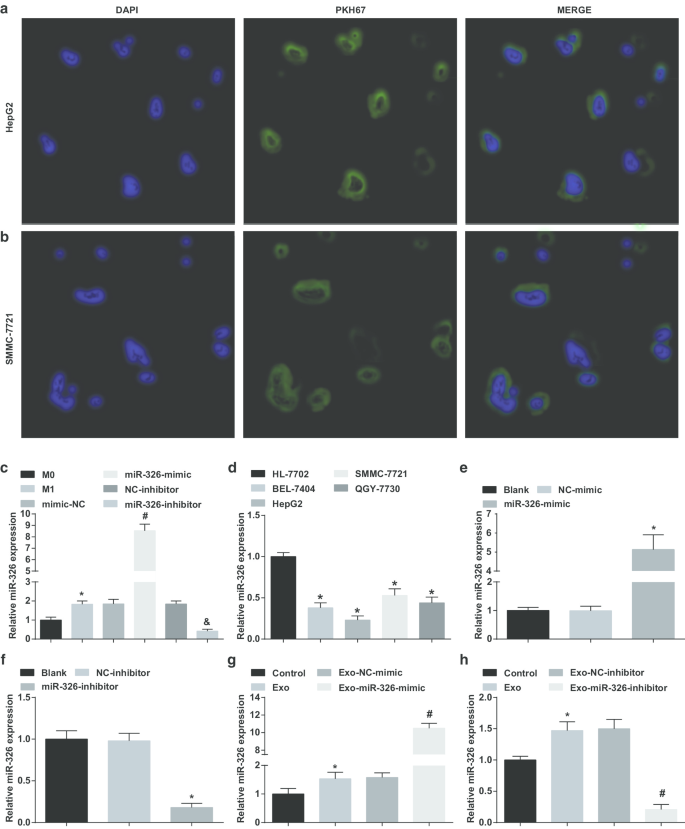
Von M1-Makrophagen abgeleitete Exosomen liefern miR-326 an HCC-Zellen und beeinflussen die miR-326-Expression in HCC-Zellen. a Aufnahme von M1-Makrophagen abgeleiteten Exosomen durch HepG2-Zellen. b Aufnahme von M1-Makrophagen-abgeleiteten Exosomen durch SMMC-7721-Zellen. c Vergleich der miR-326-Expression von Makrophagen-Exosomen in jeder Gruppe vor und nach INF-γ- und LPS-Induktion. d miR-326-Expression in HCC-Zelllinien (BEL-7404, HepG2, SMMC-7721, QGY-7703) und menschlicher normaler Hepatozyten-HL-7702-Zelllinie, nachgewiesen durch RT-qPCR. e RT-qPCR wies die Wirkung der miR-326-Mimik auf die miR-326-Expression in HepG2-Zellen nach. f RT-qPCR wies die Wirkung des miR-326-Inhibitors auf die miR-326-Expression in SMMC-7721-Zellen nach. g RT-qPCR wies die Auswirkungen von miR-326-mimiktransfizierten M1-Makrophagen-abgeleiteten Exosomen auf die miR-326-Expression in HepG2-Zellen nach. h RT-qPCR wies die Auswirkungen von miR-326-Inhibitor-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die miR-326-Expression in SMMC-7721-Zellen nach. In Feld c, *P <0,05 gegenüber dem M0-Makrophagen; In Feld d, *P <0,05 gegenüber HL-7702-Zellen; In Feld e, *P <0,05 gegenüber der NC-nachahmenden Gruppe; In Feld f, *P <0,05 gegenüber der NC-Inhibitor-Gruppe; In Feld g, *P <0,05 gegenüber der Kontrollgruppe, #P <0,05 gegenüber der Exo-NC-nachahmenden Gruppe; In Feld h, *P <0,05 gegenüber der Kontrollgruppe, #P <0,05 gegenüber der Exo-NC-Inhibitor-Gruppe. Die Messdaten wurden als Mittelwert ± Standardabweichung (N = 3), Vergleiche zwischen mehreren Gruppen wurden durch eine einseitige Varianzanalyse durchgeführt
Anschließend wurde miR-326-mimic/inhibitor in Makrophagen transfiziert und die miR-326-Expression in ihren Exosomen vor und nach der Transfektion überprüft. Es wurde gezeigt, dass im Vergleich zu den Exosomen von M0-Makrophagen die miR-326-Expression in von M1-Makrophagen abgeleiteten Exosomen erhöht war. Erhöhte miR-326-Expression wurde in den von M1-Makrophagen abgeleiteten Exosomen mit miR-326-nachahmender Behandlung beobachtet. MiR-326-Expression in M1-Makrophagen-abgeleiteten Exosomen mit miR-326-Inhibitor-Behandlung reduziert (Abb. 2c).
Dann wurde die miR-326-Expression in HCC-Zelllinien getestet. Wie sich zeigte, war die miR-326-Expression in BEL-7404-, HepG2-, SMMC-7721- und QGY-7703-Zellen im Vergleich zu HL-7702-Zellen verringert, darunter HepG2-Zellen mit der niedrigsten Expression, während SMMC-7721-Zellen mit der höchsten Expression (Abb. 2d).
Anschließend wurden miR-326-mimic und miR-326-inhibitor in HepG2- bzw. SSMC-7721-Zellen transfiziert und deren Auswirkungen auf die miR-326-Expression untersucht. MiR-326 imitiert eine erhöhte miR-326-Expression in HepG2-Zellen, während der miR-326-Inhibitor die miR-326-Expression in SSMC-7721-Zellen reduzierte (Abb. 2e, f).
Als nächstes wurden die Exosomen von M1-Makrophagen, die mit miR-326-mimic und miR-326-Inhibitor transfiziert waren, mit HepG2- bzw. SSMC-7721-Zellen co-kultiviert. Es wurde hervorgehoben, dass die Co-Kultur mit Exosomen die miR-326-Expression in HCC-Zellen erhöht, miR-326-mimic-transfizierte M1-Makrophagen-abgeleitete Exosomen die miR-326-Expression in HepG2-Zellen weiter erhöhen, während miR-326-Inhibitor-transfizierte M1-Makrophagen -abgeleitete Exosomen degradierten die miR-326-Expression in SSMC-7721-Zellen ( 2g , h). Es wird vermutet, dass M1-Makrophagen-abgeleitete Exosomen miR-326 an HCC-Zellen liefern und die miR-326-Expression in HCC-Zellen beeinflussen.
M1-Makrophagen-abgeleitetes exosomales miR-326 reduziert die Zellproliferation und die Fähigkeit zur Koloniebildung in HCC-Zellen
Bei der Untersuchung der Wirkung von exosomalem miR-326 auf die Proliferation von HCC-Zellen wurden MTT- und Koloniebildungsassays durchgeführt, um die Proliferation von HCC-Zellen zu untersuchen. Es wurde vorgeschlagen, dass in HepG2-Zellen die Wiederherstellung von miR-326 die Zellproliferation und die Fähigkeit zur Koloniebildung beeinträchtigte ( 3a , e, f). Von M1-Makrophagen abgeleitete Exosomen hinderten HepG2-Zellen daran, sich zu vermehren und Kolonien zu bilden. MiR-326 mimisch transfizierte M1-Makrophagen-abgeleitete Exosomen beeinträchtigten die Zellproliferation und die Fähigkeit zur Koloniebildung weiter (Abb. 3c, i, j).
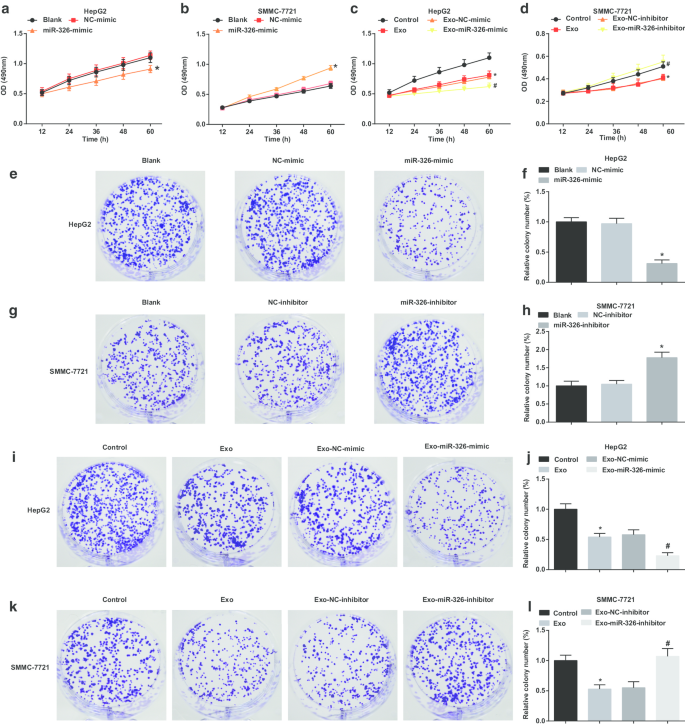
M1-Makrophagen exosomal miR-326 hemmt die Zellproliferation und Koloniebildung in HCC-Zellen. a Der MTT-Assay wies die Wirkung der Transfektion von miR-326-Mimetika auf die Proliferation von HepG2-Zellen nach. b Der MTT-Assay wies die Wirkung der Transfektion des miR-326-Inhibitors auf die Proliferation von SMMC-7721-Zellen nach. c Der MTT-Assay wies die Wirkung einer Co-Kultur mit miR-326 mimisch-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die Proliferation von HepG2-Zellen nach. d Der MTT-Assay zeigte die Wirkung einer Kokultur mit miR-326-Inhibitor-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die Proliferation von SMMC-7721-Zellen. e Der Koloniebildungsassay wies die Wirkung der Transfektion von miR-326-Mimetika auf die Koloniebildungsfähigkeit von HepG2-Zellen nach. f Koloniezahlen von HepG2-Zellen. g Der Koloniebildungsassay wies die Wirkung der Transfektion des miR-326-Inhibitors auf die Koloniebildungsfähigkeit von SMMC-7721-Zellen nach. h Koloniezahlen von SMMC-7721-Zellen. ich Der Koloniebildungsassay wies die Wirkung einer Kokultur mit miR-326 mimisch-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die Koloniebildungsfähigkeit von HepG2-Zellen nach. j Koloniezahlen von HepG2-Zellen, die mit Exosomen behandelt wurden. k Der Koloniebildungsassay wies die Wirkung der Kokultur von miR-326-Inhibitor-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die Koloniebildungsfähigkeit von SMMC-7721-Zellen nach. l Koloniezahlen von SMMC-7721-Zellen, die mit Exosomen behandelt wurden. In Feld a und f, *P <0,05 gegenüber der NC-nachahmenden Gruppe; In den Feldern b und h *P <0,05 gegenüber der NC-Inhibitor-Gruppe; In den Feldern c und j, *P <0,05 gegenüber der Kontrollgruppe, #P <0,05 gegenüber der Exo-NC-nachahmenden Gruppe; In den Feldern d und l, *P <0,05 gegenüber der Kontrollgruppe, #P <0,05 gegenüber der Exo-NC-Inhibitor-Gruppe. Die Messdaten wurden als Mittelwert ± Standardabweichung (N = 3), Vergleiche zwischen mehreren Gruppen wurden durch eine einseitige Varianzanalyse durchgeführt
In SMMC-7721-Zellen verstärkte der miR-326-Knockdown die Zellproliferation und die Fähigkeit zur Koloniebildung (Fig. 3b, g, h). In SMMC-7721-Zellen, die mit M1-Makrophagen-abgeleiteten Exosomen behandelt wurden, waren die Zellproliferation und die Fähigkeit zur Koloniebildung verringert. MiR-326-Inhibitor-transfizierte M1-Makrophagen-abgeleitete Exosomen förderten weiter die Zellproliferation und die Fähigkeit zur Koloniebildung (Fig. 3d, k, l). Es wird angedeutet, dass miR-326, abgeleitet von M1-Makrophagen-abgeleiteten Exosomen, die Proliferation von HCC-Zellen behindert.
M1-Makrophagen-abgeleitetes exosomales miR-326 unterdrückt die Migration und Invasion von HCC-Zellen
Anschließend wurde die Wirkung von exosomalem miR-326 auf die Invasion und Migration von HCC-Zellen untersucht. Es wurde gezeigt, dass in HepG2-Zellen die Wiederherstellung von miR-326 die Invasion und Migration einschränkte (Abb. 4a–c). Von M1-Makrophagen abgeleitete Exosomen störten HepG2-Zellen beim Eindringen und Wandern. Invasion und Migration wurden weiter abgebaut, wenn HepG2-Zellen mit miR-326-mimic-transfizierten M1-Makrophagen-abgeleiteten Exosomen cokultiviert wurden (Abb. 4g–i).
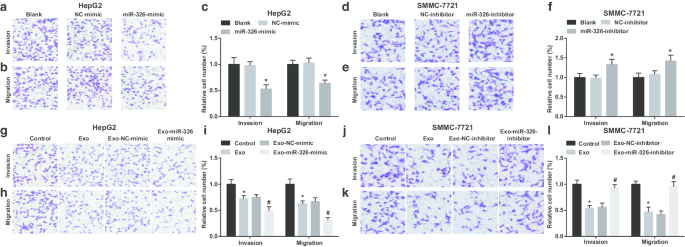
M1-Makrophagen exosomal miR-326 hemmt die Migration und Invasion von HCC-Zellen. a Der Transwell-Assay wies die Wirkung der Transfektion von miR-326-Mimetika auf die Invasion von HepG2-Zellen nach. b Der Transwell-Assay wies die Wirkung der Transfektion von miR-326 nach, die die Migration von HepG2-Zellen nachahmt. c Die Zahl der Invasion und Migration von HepG2-Zellen. d Der Transwell-Assay wies die Wirkung der Transfektion des miR-326-Inhibitors auf die Invasion von SMMC-7721-Zellen nach. e Der Transwell-Assay zeigte die Wirkung der Transfektion des miR-326-Inhibitors auf die Migration von SMMC-7721-Zellen. f Die Zahl der Invasion und Migration von SMMC-7721-Zellen. g Der Transwell-Assay wies die Wirkung einer Kokultur mit miR-326 mimisch-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die Invasion von HepG2-Zellen nach. h Der Transwell-Assay wies die Wirkung einer Kokultur mit miR-326 mimisch-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die Migration von HepG2-Zellen nach. ich Die Anzahl der Invasion und Migration von HepG2-Zellen, die mit M1-Makrophagen-abgeleiteten Exosomen cokultiviert wurden. j Der Transwell-Assay zeigte die Wirkung einer Kokultur mit miR-326-Inhibitor-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die Invasion von SMMC-7721-Zellen. k Der Transwell-Assay zeigte die Wirkung einer Kokultur mit miR-326-Inhibitor-transfizierten M1-Makrophagen-abgeleiteten Exosomen auf die Migration von SMMC-7721-Zellen. l Die Zahl der Invasion und Migration von SMMC-7721-Zellen, die mit M1-Makrophagen-abgeleiteten Exosomen cokultiviert wurden. In Feld c, *P <0,05 gegenüber der NC-nachahmenden Gruppe; In Feld f, *P <0,05 gegenüber der NC-Inhibitor-Gruppe; In Panel i, *P <0,05 gegenüber der Kontrollgruppe, #P <0,05 gegenüber der Exo-NC-nachahmenden Gruppe; In Feld l, *P <0,05 gegenüber der Kontrollgruppe, #P <0,05 gegenüber der Exo-NC-Inhibitor-Gruppe. Die Messdaten wurden als Mittelwert ± Standardabweichung (N = 3), Vergleiche zwischen mehreren Gruppen wurden durch eine einseitige Varianzanalyse durchgeführt
MiR-326 knockdown resulted in enhancements in SMMC-7721 cell invasion and migration (Fig. 4d–f). When treated with M1 macrophages-derived exosomes, SMMC-7721 cells were exhibited with decreased invasion and migration. However, SMMC-7721 cell invasion and migration were boosted upon co-culture with miR-326-inhibitor-transfected M1 macrophages-derived exosomes (Fig. 4j–l). It is implied that miR-326 derived from M1 macrophage exosomes impedes the invasion and migration of HCC cells.
M1 Macrophage-Derived Exosomal miR-326 Promotes Apoptosis of HCC Cells
When examining the effect of exosomal miR-326 on the cell cycle and apoptosis of HCC cells, PI single staining and Annexin V-FITC/PI double staining were applied. It was illustrated that miR-326 overexpression increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases and raised apoptosis in HepG2 cells (Fig. 5a–d). Co-culturing with M1 macrophages-derived exosomes increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases and raised cell apoptosis of HepG2 cells. Co-cultivation with exosomes from M1 macrophages transfected with miR-326-mimic further enhanced these effects (Fig. 5i–l).
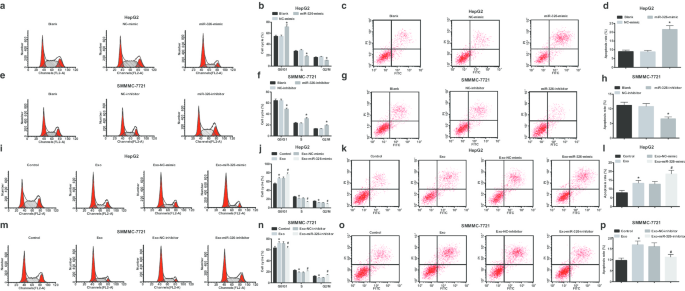
M1 macrophage exosomal miR-326 promotes apoptosis of HCC cells. a , b Flow cytometry detected the effect of transfection of miR-326 mimic on HepG2 cell cycle; c , d flow cytometry detected the effect of transfection of miR-326 mimic on HepG2 cell apoptosis; e , f flow cytometry detected the effect of transfection of miR-326 inhibitor on SMMC-7721 cell cycle; g , h flow cytometry detected the effect of transfection of miR-326 inhibitor on SMMC-7721 cell apoptosis; i , j flow cytometry detected the effects of co-culture of with miR-326 mimic-transfected M1 macrophage-derived exosomes on HepG2 cell cycle; k , l flow cytometry detected the effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on HepG2 cell apoptosis; m , n flow cytometry detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on SMMC-7721 cell cycle; o , p , flow cytometry detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on SMMC-7721 cell apoptosis. In panel b and d, *P <0.05 versus the NC-mimic group; In panel f and h, *P <0.05 versus the NC-inhibitor group; In panel j and l, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel n and p, *P <0.05 versus the controlgroup, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (N = 3), comparisons among multiple groups were conducted by one-way analysis of variance
In SMMC-7721 cells, miR-326 down-regulation reduced cells arrested at G0/G1 phase, elevated cells arrested at S and G2/M phases, and declined cell apoptosis (Fig. 5e–h). Untransfected M1 macrophages-derived exosomes increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases, and heightened cell apoptosis. MiR-326-inhibitor-transfected M1 macrophages-derived exosomes degraded cells arrested at G0/G1 phase, elevated cells arrested at S and G2/M phases, and decreased cell apoptosis (Fig. 5m–p). Briefly, it is summarized that miR-326 derived from M1 macrophage exosomes arrests cell cycle in G0/G1 phase and induces cell apoptosis in HCC.
M1 Macrophage-Derived Exosomal miR-326 Declines CD206 and NF-κB Expression in HCC Cells
Next, the potential mechanism of miR-326 derived from M1 macrophage exosomes in the biological progress of HCC cells was explored. NF-κB is the key link between inflammation and cancer. Many regulatory proteins and miRNAs could inhibit the excessively activated NF-κB signaling to suppress cancer [16]. Such a beneficial effect may include the polarization of M2 macrophages into M1 macrophages. CD206 and NF-κB expression in HepG2 and SMMC-7721 cells was tested by RT-qPCR. It was suggested that miR-326 restoration decreased CD206 and NF-κB expression in HepG2 cells, while miR-326 knockdown enhanced CD206 and NF-κB expression in SMMC-7721 cells (Fig. 6a, b). Moreover, co-culture with M1 macrophage exosomes significantly reduced CD206 and NF-κB expression in HepG2 cells, while co-culture with M1 macrophage exosomes-overexpressing miR-326 further decreased CD206 and NF-κB expression. Treated with untransfected M1 macrophages-derived exosomes, CD206 and NF-κB expression was decreased in SMMC-7721 cells. Co-cultured with miR-326-inhibitor-transfected M1 macrophages-derived exosomes, SMMC-7721 cells were featured by heightened CD206 and NF-κB expression (Fig. 6c, d). It was concluded that miR-326 from M1 macrophage exosomes played a tumor suppressor by inhibiting NF-κB in HCC cells.
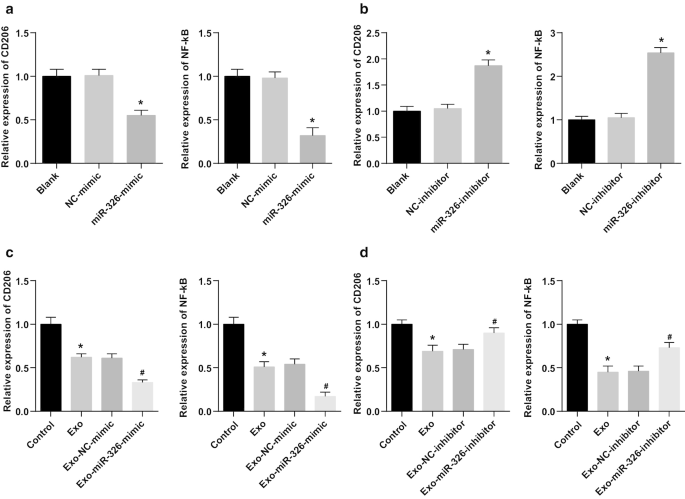
M1 macrophage exosomal miR-326 declines CD206 and NF-κB expression in HCC cells. a RT-qPCR detected the effect of transfection of miR-326 mimic on the expression of CD206 and NF-κB in HepG2 cells; b RT-qPCR detected the effect of transfection of miR-326 inhibitor on the expression of CD206 and NF-κB in SMMC-7721 cells. c RT-qPCR detected the effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on the expression of CD206 and NF-κB in HepG2 cells; d RT-qPCR detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on the expression of CD206 and NF-κB in SMMC-7721 cells. In panel a, *P <0.05 versus the NC-mimic group; In panel b, *P <0.05 versus the NC-inhibitor group; In panel c, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel d, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (N = 3), comparisons among multiple groups were conducted by one-way analysis of variance
miR-326 from M1 Macrophage Exosomes Inhibits HCC Tumor Growth In Vivo
Finally, the in vivo results were validated through tumor xenografts. As displayed, miR-326 overexpression decreased volume and weight of tumors in HepG2 cells (Fig. 7a–c). In mice transplanted with HepG2 cells co-cultured with exosomes, the treatment with M1 macrophage exosomes significantly reduced the tumor volume and weight of HepG2 cells, while treatment with M1 macrophages-overexpressing miR-326 further reduced the tumor volume and weight of HepG2 cells (Fig. 7g–i).
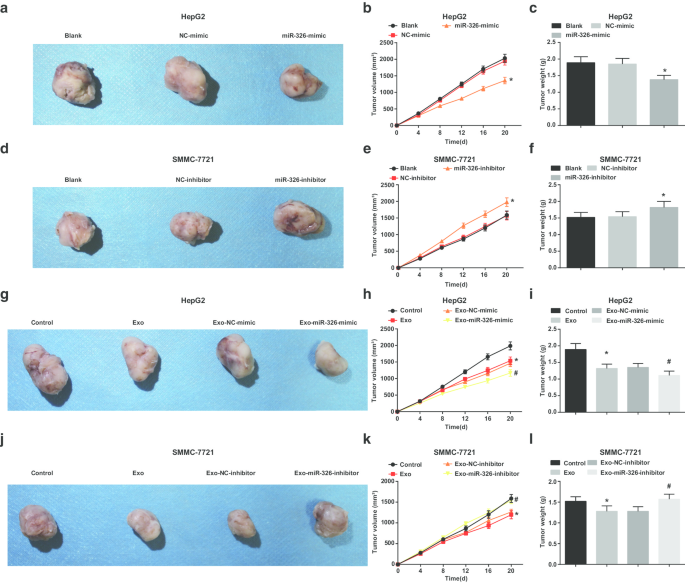
M1 macrophage exosomal miR-326 reduces the volume and weight of HCC tumor in vivo. a –c The effect of transfection of miR-326 mimic on the tumor volume and tumor of nude mice xenografted with HepG2 cells. d –f The effect of transfection of miR-326 inhibitor on the tumor volume and tumors of nude mice xenografted with SMMC-7721 cells. g –ich The effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on the tumor volume and tumor of nude mice xenografted with HepG2 cells. j –l The effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on the tumor volume and tumor of nude mice xenografted with SMMC-7721 cells. In panel b and c, *P <0.05 versus the NC-mimic group; In panel e and f, *P <0.05 versus the NC-inhibitor group; In panel h and i, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel k and l, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (n = 5), comparisons among multiple groups were conducted by one-way analysis of variance
In SMMC-7721 cells, miR-326 suppression increased volume and weight of tumors (Fig. 7d–f). Untransfected M1 macrophages-derived exosomes obstructed tumor growth in volume and weight of SMMC-7721 cells. Co-cultured with miR-326 inhibitor-transfected M1 macrophages-derived exosomes, SMMC-7721 cells were injected into mice and caused elevations in tumor volume and weight (Fig. 7j–l). Summarily, miR-326 derived from M1 macrophage exosomes depressed tumor growth of HCC in vivo.
Discussion
HCC is a common cancer that is characterized with high morbidity and mortality, difficult early diagnosis and treatment, poor prognosis and 5-year survival rate [17]. Recently, a study has highlighted that the lowly expressed lncRNA cox-2 declines the ability of M1 macrophages to suppress HCC cell growth, invasiveness, angiogenesis migration and promote apoptosis [18]. Hu et al. have discussed that miR-326 is obviously degraded in HCC tissues and cell lines, while down-regulated miR-326 is connected to the TNM stage, lymph node metastasis and differentiation of HCC patients [14]. It is customarily considered that HCC cells-derived exosomes can form a fertile environment to facilitate HCC cells growth, invasiveness and metastasis as well as development of drug resistance [19]. The current study was designed to explore the mechanism of exosomal miR-326 in regulating invasion and migration of HCC cells.
Our results indicated that miR-326 expression decreased in HCC cells but increased in exosomes. A recent study has pointed out that miR-326 expression is declined in HCC tissues [20]. Another study has presented miR-326 expression is notably reduced in HCC cell lines and tissues and its down-regulation predicts a poor prognosis in HCC [13]. It is reported that miR-326 acts a tumor-suppression role and is greatly depressed in HCC cells [21]. All these aforementioned evidences are in line with our findings. A study has purported that in comparison with the controls, miR-326 expression is raised in Tconv-derived exosomes which is observed in relapsing–remitting multiple sclerosis patients [11].
Other results emerge from our data that highly expressed exosomal miR-326 reduced cell proliferation, colony formation, migration and invasion as well as facilitated apoptosis of HCC cells in vitro and reduced the volume and weight of HCC tumor in vivo. It has been suggested previously that HCC cell growth can be suppressed via overexpression of miR-326, and HCC cell migration and invasion ability are markedly attenuated through elevating miR-326 [21]. It is reported that the up-regulated miR-326 expression suppresses HCC cell growth and invasiveness as well as stimulates cell apoptosis in vitro [14]. Besides that, a prior study has verified that overexpression of miR-326 declined tumor growth in vivo [13]. A study has revealed that ectopic expressed miR-326 markedly attenuates cell growth, and suppresses cellular migration and invasiveness in non-small cell lung cancer cell lines [22]. Moreover, it is found that miR-326 decreases profibrotic genes like MMP-9, implying its repressive function in cancer cell proliferation [23]. Also, it is presented that miR-326 represses Bcl-2 protein expression and elevates Bax expression so as to affect the apoptosis [24]. Similar to our findings, there are some miRNAs interacting with exosomes to play a role in HCC development. It is displayed that highly expressed exosomal miR-638 can repress the proliferation of HCC cells, involving the potential impact on carcinogenesis [25]. Another study also proves that HCC cells-derived exosomal miR-451a suppresses tumor angiogenesis via disrupting endothelial functions as apoptosis, tube formation, migration and permeability [26]. A prior research generally confirms that when treated with the overexpression of miR-744 exosomes, the proliferation of HCC cells is dramatically suppressed [27].
Conclusion
To briefly conclude, our study provides evidence that M1 macrophage-derived exosomal miR-326 suppresses proliferation, migration and invasion as well as advances apoptosis of HCC cells, supplying a new insight in a novel target therapy for HCC. Due to the limited sample size and limited known researches, the exact mechanism of miR-326 is not fully elucidated, and therefore, further large-scale studies are required to illustrate the underlying mechanism.
Abkürzungen
- HCC:
-
Hepatocellular carcinoma
- miRNAs:
-
MicroRNAs
- FBS:
-
Fetal bovine serum
- M/CSF:
-
Macrophage colony-stimulating factor
- PBS:
-
Phosphate buffered saline
- DMEM:
-
Dulbecco’s modified eagle medium
- PPAR:
-
Peroxisome proliferator-activated receptor
- TEM:
-
Transmissionselektronenmikroskop
- NTA:
-
Nanoparticle tracking analysis
- NC:
-
Negative control
- RT-qPCR:
-
Reverse transcription quantitative polymerase chain reaction
- BCA:
-
Bicinchoninic acid
- BSA:
-
Bovine serum albumin
- Exo:
-
Exosomes
- ANOVA:
-
Einseitige Varianzanalyse
Nanomaterialien
- Vollständige Terahertz-Polarisationskontrolle mit erweiterter Bandbreite über dielektrische Metaoberflächen
- In-vitro-Studie zum Einfluss von Au-Nanopartikeln auf HT29- und SPEV-Zelllinien
- Effekt einer schräg gesputterten ITO-Elektrode in MAPbI3-Perowskit-Solarzellenstrukturen
- Herstellung und Charakterisierung eines neuen anodischen Tio2-Kohlenstoff-Nanofaser-Verbundkatalysators für eine Direkt-Methanol-Brennstoffzelle mittels Elektrospinnverfahren
- Verbesserte Antitumorwirksamkeit und Pharmakokinetik von Bufalin durch PEGylierte Liposomen
- Abstimmung des Gleit-Roll-Bewegungsmodus von Kohlenstoffnanoröhren über Hydroxylgruppen
- Bewertung der durch Goldnanopartikel gehemmten Aktivität von Cytochrom P450 3A4 und der molekularen Mechanismen, die seiner zellulären Toxizität in der humanen hepatozellulären Karzinomzelllinie C…
- Winzige Seltenerd-Fluorid-Nanopartikel aktivieren das Wachstum von Tumorzellen über elektrische polare Wechselwirkungen
- Wechsel des Fehlermechanismus in einer programmierbaren Metallisierungszelle auf Zinkperoxidbasis
- Autophagie-Inhibitor (LY294002) und 5-Fluorouracil (5-FU) Kombinations-basiertes Nanoliposom für verbesserte Wirksamkeit gegen Plattenepithelkarzinom des Ösophagus



