Knochenmark-mesenchymale Stammzellen-abgeleitete exosomale MicroRNA-133a hemmt Myokardfibrose und Epithel-Mesenchym-Übergang bei Ratten mit viraler Myokarditis durch Unterdrückung von MAML1
Zusammenfassung
Myokarditis ist eine Krankheit, die durch eine lokalisierte oder diffuse Entzündung des Myokards ohne wirksame Behandlung gekennzeichnet ist. Diese Studie untersuchte den Regulationsmechanismus von microRNA-133 (miR-133), die aus mesenchymalen Stammzellen-abgeleiteten Exosomen des Knochenmarks (BMSC-Exo) auf Myokardfibrose und epithelial-mesenchymale Transition (EMT) bei Ratten mit viraler Myokarditis (VMC) durch Regulation . sezerniert wird Mastermind-like 1 (MAML1). BMSCs in Ratten wurden isoliert und kultiviert, um ihren Immunphänotyp und ihre osteogene und adipogene Fähigkeit zu identifizieren, und BMSC-Exo wurde extrahiert und identifiziert. Exosomen wurden durch Ultrazentrifugation erhalten, die durch Transmissionselektronenmikroskop und Western-Blot-Analyse identifiziert wurden. Den Ratten wurde das Coxsackie-B3-Virus zur Herstellung des VMC-Modells injiziert, und die Kardiomyozyten wurden isoliert, kultiviert und wie im Tierversuch gruppiert (NC Exo , Ad-miR-133a Exo , Adas-miR-133a Exo ). In-vivo- und in-vitro-Experimente wurden durchgeführt, um die Rolle von exosomalem miR-133a und MAML1 bei Entzündungen, Apoptose, EMT, Fibrose und Zelllebensfähigkeit herauszufinden. Die Targeting-Beziehung zwischen miR-133a und MAML1 wurde durch einen dualen Luciferase-Reportergen-Assay verifiziert. BMSC-Exo erhöhte die miR-133a-Expression in VMC-Ratten und verbesserte effektiv die Herzfunktion von VMC-Ratten und Myokardfibrose, erhöhte die Lebensfähigkeit der Kardiomyozyten und hemmte den EMT-Prozess. Erhöhte miR-133a in Exosomen verstärkten die Verbesserungen. Silenced miR-133a kehrte die Auswirkungen von BMSC-Exo auf VMC-Ratten effektiv um. miR-133a zielte auf MAML1 ab. Die Hemmung von MAML1 verbesserte die Herzfunktion und Myokardfibrose bei VMC-Ratten und könnte die Wirkung von miR-133a-stummgeschalteten Exosomen bei VMC-Ratten umkehren. Unsere Studie legt nahe, dass ein erhöhtes exosomales miR-133a Myokardfibrose und EMT bei Ratten mit VMC durch Herunterregulieren von MAML1 unterdrückt und dadurch das Fortschreiten der Myokarditis hemmt.
Einführung
Myokarditis gilt als entzündliche Erkrankung der Herzmuskelzellen [1]. Myokarditis ist offensichtlich bei Männern bekannter als bei Frauen [2]. Virale Myokarditis (VMC) ist ein Hauptfaktor, der bei jungen Menschen zu dilatativer Kardiomyopathie (DCM) und plötzlichem Tod führt [3]. Die klinische Leistungsfähigkeit der Myokarditis ist vielfältig und reicht von asymptomatischen Zuständen mit mehrdeutigen Anzeichen und Symptomen bis hin zu ernsthafter Myokardzerstörung durch Viren und Immunzellen, die an kardiogenem Schock und Arrhythmien leiden [1]. Myokarditis kann durch eine Vielzahl infektiöser Elemente, bestehend aus Viren, Bakterien, Chlamydien, Rickettsien, Pilzen und Protozoen, zusammen mit nichtinfektiösen Induktoren induziert werden. Unter anderem war eine Virusinfektion die häufigste Ursache, insbesondere bei Kindern [4]. Das Coxsackie-B3-Virus (CVB3) kann als das wichtigste Virus, das zu Myokarditis führt, bei der Pathogenese von VMC zu einer oxidativen Stressreaktion und Apoptose führen, eine spezielle Behandlung von VMC wurde jedoch noch nicht beschrieben [5]. Zudem ist die Pathogenese der VMC nicht gut dokumentiert und auch eine exakte klinische Therapie fehlt [3]. Daher werden dringend neue Targets benötigt, um die Prognose der Krankheit zu verbessern.
MicroRNAs (miRNAs) sind endogene nichtkodierende RNAs, die die Expression proteinkodierender Gene regulieren können [6]. MiR-133a ist als eine der herzspezifischen miRNAs an der Herzentwicklung und einigen kardiovaskulären Erkrankungen beteiligt, die einen Myokardinfarkt (MI) enthalten [7]. Darüber hinaus wird miR-133a bei der chronischen Kardiomyopathie der Chagas-Krankheit anomal exprimiert [8]. Darüber hinaus ist der miR-133a-Spiegel im Myokard mit Entzündungen, der linksventrikulären Funktion und dem klinischen Ergebnis bei entzündlicher Kardiomyopathie verbunden [9]. MiRNAs wurden in Exosomen gefunden, die aus Maus- und menschlichen Mastzellen stammen [10]. Exosomen, nanoskalige Vesikel, die von den meisten Zellarten freigesetzt werden, finden sich in verschiedenen biologischen Flüssigkeiten [11]. Exosomen können ihre Fracht auf Empfängerzellen übertragen, was nachweislich die biochemische Zusammensetzung und Signalwege der Empfängerzellen verändert [12, 13]. Es gibt Hinweise darauf, dass veränderte exosomale miRNAs mit der Pathogenese der CVB3-induzierten Myokarditis in Verbindung stehen [14]. Es wurde angesprochen, dass exosomales miR-125b-5p aus Hypoxie-konditionierten mesenchymalen Knochenmarkstammzellen die Apoptose von Kardiomyozyten reduziert und die ischämische Herzreparatur verbessert [15]. Darüber hinaus lindert exosomales miR-25-3p aus MSCs den MI, indem es die Apoptose der Kardiomyozyten und die Entzündungsreaktion reduziert [16]. Von Interesse ist exosomales kardiales exprimiertes miR-133a mit dem kardialen Troponinkardialen Troponin-I [17]. Mastermind-like 1 (MAML1) war in unserer Studie ein kreuzgescreentes Downstream-Gen von miR-133a, von dem berichtet wurde, dass es an einer Verletzung von Myokardischämie/Reperfusion (I/R) beteiligt ist [18]. Eine kürzlich durchgeführte Studie hat auch erwähnt, dass der MAML1-Knockdown die antifibrotische Funktion bei Leberfibrose besitzt [19].
Durch frühere Studien beleuchtet, stellt sich die Frage, ob von BMSCs abgeleitetes exosomales miR-133a eine Myokarditis vermitteln könnte. Daher wurde diese Studie mit der Hypothese begonnen, dass miR-133, das von einem BMSC-derived Exosom (BMSC-Exo) transportiert wird, die Myokardfibrose und den epithelial-mesenchymalen Übergang (EMT) bei VMC-Ratten durch Regulation von MAML1 verbessert.
Materialien und Methoden
Ethische Genehmigung
Die Studie wurde vom Institutional Animal Care and Use Committee des Vierten angegliederten Krankenhauses der Medizinischen Fakultät der Zhejiang University genehmigt. Tiere wurden artgerecht behandelt.
Isolation von BMSCs
Die Versuchstiere waren männliche Sprague-Dawley (SD)-Ratten (Experimental Animal Center of Zhejiang University School of Medicine, Zhejiang, China). Die Ratten wurden durch intraperitoneale Injektion mit Pentobarbital-Natrium euthanasiert und mit 75% Alkohol sterilisiert. Femur und Tibia wurden auf einem ultrareinen Tisch herausgenommen, Muskel- und Bindegewebe wurden entfernt und die Markhöhle wurde wiederholt mit Dulbecco modifiziertem Eagle-Medium (DMEM) mit niedrigem Glucosegehalt gespült. Die Flüssigkeit wurde zentrifugiert, um die Präzipitate zu sammeln, die resuspendiert und für 24 h inkubiert wurden (das Medium wurde alle 2–3 d gewechselt). Beim Anwachsen bis zur logarithmischen Phase wurden BMSCs mit 0,25% Trypsin (Gibco, Carlsbad, Kalifornien, USA) abgelöst, zentrifugiert und in MSC-Kulturlösung (Cyagen Biosciences Inc., Guangzhou, China) resuspendiert. Die Suspension wurde im Verhältnis 1:2 durchgeleitet. Der obige Vorgang wurde wiederholt und die BMSCs der 4. Passage wurden für nachfolgende Experimente verwendet.
Identifizierung von BMSCs
Die Oberflächenantigene der BMSCs der 4. Passage in logarithmischem Wachstum wurden durch Durchflusszytometrie identifiziert. Die BMSCs wurden mit 0,25% Trypsin (1 ml), das Ethylendiamintetraessigsäure enthielt, abgelöst, zentrifugiert, mit geeigneter phosphatgepufferter Kochsalzlösung (PBS) resuspendiert und bei 151 g zentrifugiert. Die BMSCs wurden dann mit PBS resuspendiert, das 2% frisches fötales Rinderserum (FBS) (Gibco) enthielt, um eine Einzelzellsuspension herzustellen. FITC-CD34, PE-CD29 und PE-CD44 monoklonaler Antikörper (jeweils 5 µL, BD Biosciences, Franklin Lakes, NJ, USA) wurden mit der Zellsuspension (100 µL) inkubiert, bei 151 g zentrifugiert, mit 500 . resuspendiert μL PBS mit 1% Paraformaldehyd und 30 min fixiert. Die Hintergrundmarker wurden mithilfe von monoklonalen Antikörpern aus der Homotypkontrolle identifiziert.
Durchflusszytometrie:Die Einzelzellsuspension wurde fixiert und bei 151 g zentrifugiert. Dann wurden die BMSCs mit PBS mit 1 % Paraformaldehyd resuspendiert, auf dem MACS Quart Durchflusszytometer getestet und mit der entsprechenden Software analysiert.
Induktion der Osteogenese und Adipogenese von BMSCs
BMSCs in der 4. Passage wurden in 6-Well-Platten mit 200 Zellen/ml ausgesät. Die Osteoblasten-Induktionslösung und die adipogene Induktionslösung (Cyagen Biosciences Inc.) wurden in BMSCs mit 60–70% Konfluenz zugegeben. BMSCs in den anderen beiden Vertiefungen wurden nicht mit Induktionsflüssigkeiten als Kontrollen hinzugefügt. Die BMSCs wurden für 14 Tage induziert und mit 4% Paraformaldehyd fixiert. Dann wurden die differenzierten Osteoblasten und Adipozyten mit Alizarin-Rot-Färbung und Öl-Rot-O-Färbung (Wuhan Pulande Biological Technology Co., Ltd., Wuhan, China) umgesetzt und unter einem Mikroskop beobachtet.
Exosomenisolierung und -identifizierung
BMSCs in der 4. Passage wurden 48 h lang kultiviert, um den Überstand zu ernten, der dann zentrifugiert wurde (800 g und 2000 g), mit 0,22 &mgr;m und 100000 MW Filtermembranen filtriert und zentrifugiert (100000 g), um die Präzipitate zu sammeln. Dann wurden die Präzipitate mit PBS resuspendiert, erneut bei 100.000 g zentrifugiert, um eine Exosomenpräzipitation zu erhalten. Die BMSC-Exo-Suspension in PBS wurde einem Konzentrationsnachweis durch Bicinchoninsäure (BCA) und einem Nachweis des Exosom-Maker-Proteins (CD63, CD81 und CD9) durch Western-Blot-Analyse (Proteintech, Chicago, IL, USA) unterzogen.
Rekombinante Adenovirus-Infektion vermittelt miR-133a-Genmodifikation von BMSCs
BMSCs wurden über Nacht passagiert. Die normale Kontrolle (gleiche Menge an PBS), die miR-133a-Negativkontrolle (NC), die miR-133a-Überexpression (Ad-miR-133a) und die miR-133a-Niedrigexpression (Adas-miR-133a) ( Hanbio Biotechnology Co., Ltd., Shanghai, China) wurden mit BMSCs gemäß 100 Infektionsmultiplizität (MOI) transfiziert. Die BMSCs wurden kultiviert und die entsprechenden Exosomen (NC Exo , NC Exo , Ad-miR-133a Exo , und Adas-miR-133a Exo ) wurden durch Ultrazentrifugation erhalten [20].
Etablierung eines VMC-Modells bei Ratten und Gruppierung von Versuchstieren
Reife männliche SD-Ratten vom SPF-Grad wurden in 10 Gruppen mit jeweils acht Ratten eingeteilt. Coxsackievirus B3 (CVB3) wurde vom Institute of Medical Biotechnology, Chinese Academy of Medical Sciences (Beijing, China) bereitgestellt.
CVB3 (10 mg/kg) wurde Ratten intraperitoneal injiziert, während PBS oder BMSC-Exo (100 &mgr;g) durch die Schwanzvene injiziert wurde. Normalen Ratten als Kontrollen wurde CVB3-Kulturlösung und PBS injiziert. Ratten, denen 10 mg/kg CVB3 injiziert wurden, erhielten zusätzlich PBS, MSC exo . , NC Exo , Adas-miR-133a Exo , Ad-miR-133a Exo , si-NC oder si-MAML1 (RIBOBIO, Guangzhou, China).
Den Ratten wurde 7 Tage lang kontinuierlich injiziert und das Augapfelblut wurde entnommen. Das Blut wurde zentrifugiert und das Serum wurde gesammelt, unterverpackt und bei – 20 °C gelagert. Nach der Euthanasie der Ratten wurden die Herzproben entnommen, mit 10 % Formaldehyd fixiert, mit Gradientenalkohol dehydriert, mit Xylol geklärt und mit Paraffin eingebettet, zur histologischen Untersuchung geschnitten. Ein Teil der Schnitte wurde bei − 80 °C als Material für molekularbiologische Experimente platziert.
Echokardiographie
Am 7. Tag nach der Virusinjektion wurde den Ratten intraperitoneal Pentobarbitalnatrium 25 mg/kg injiziert. Nach vollständiger Anästhesie wurde die Gliedmaßenleitung des Elektrokardiogrammgeräts, die mit der Elektrodennadel verbunden war, subkutan an den Enden der Gliedmaßen der Ratten eingeführt und das Elektrokardiogramm der Gliedmaßenleitung wurde aufgezeichnet. Dann wurden die Ratten in Rückenlage leicht nach links fixiert, der Brustkorb wurde enthaart und das Elektrokardiogramm der zweiten Ableitung wurde angeschlossen, um das Doppler-Spektrum des Blutflusspulses der Aorta im parasternalen Vierkammer-Herzabschnitt zu erhalten. Zu den Indikatoren gehörten die linksventrikuläre Hinterwanddicke (LVPW), der linksventrikuläre endsystolische Durchmesser (LVIDs), die linksventrikuläre Verkürzungsfraktion (FS) und die linksventrikuläre Ejektionsfraktion (LVEF).
Hämatoxylin-Eosin (HE)-Färbung
Die Gewebe wurden mit 4% Paraformaldehyd fixiert, dehydriert, geklärt und mit Paraffin eingebettet. Dann wurden die 4 µm-Schnitte entwachst, mit Hämatoxylin (Servicebio, Wuhan, China) gefärbt, mit 1% Salzsäurealkohol differenziert, wieder blau gefärbt und mit Eosin gefärbt, dehydratisiert, mit Xylol geklärt, mit neutralem Gummi versiegelt, und unter einem optischen Mikroskop (Olympus, Tokio, Japan) beobachtet.
Masson-Kollagenfärbung
Paraffinschnitte wurden entwachst, mit Hämatoxylin weniger als 2 min gefärbt, mit Lichun-Magenta-Lösung gefärbt und schnell mit 0,5% Eisessig-Lösung gespült. Dann wurden die Schnitte mit 1%iger wässriger Aluminiumphosphatlösung gefärbt, von dunkelrot über hellrot bis pink gefärbt und unter einem Mikroskop betrachtet. Dann wurden die Schnitte mit Anilinblau (Pulande) gefärbt, konventionell mit Xylol entwässert und versiegelt. Die medizinische Bildanalysesoftware Image-Proplus 6.0 wurde verwendet, um die positive Färbefläche von Kollagenfasern und die Kollagenvolumenfraktion (CVF) =Kollagenfläche/Gesamtfeldfläche zu messen. Der Ort der Färbung und die Farbe der kollagenen Fasern wurden unterschieden (die Kardiomyozyten waren rot und die kollagenen Fasern waren blau gebänderte oder homogene Strukturen im Interzellularraum).
terminale Desoxynukleotidyltransferase-vermittelte Desoxyuridintriphosphat-Biotin Nick End-Labeling (TUNEL)-Färbung
Paraffinschnitte wurden entwachst, in den Citratpuffer gegeben und 10 min bei 350 W gebacken. Die Schnitte wurden mit 50 µl TUNEL-Lösung versetzt, mit 50 µl Umwandlungsmittel-Peroxidase verbunden, mit DAB entwickelt und unter dem Mikroskop beobachtet. Die Schnitte wurden in Hämatoxylin gelegt, in 95 % Ethanol I–II getaucht, mit wasserfreiem Ethanol I–II, Xylol I–II verbunden und versiegelt. Die Ergebnisse wurden unter einem optischen Mikroskop analysiert.
Enzyme-Linked Immunosorbent Assay (ELISA)
Tumornekrosefaktor α (TNF-α), Interleukin (IL)-1β und IL-6 wurden durch ELISA-Kits (BOSTER Biological Technology Co. Ltd., Wuhan, China) nachgewiesen. Das Augapfelblut wurde bei 604 g zentrifugiert, um das obere Serum zu sammeln. Der durch Zentrifugation aus Zellkulturmedium gewonnene Überstand wurde im Zellexperiment nachgewiesen. Es gab sieben Konzentrationsgradienten im Verdünnungsstandard der Proben. Die leere Vertiefung wurde mit Probenverdünner verbunden und eine weitere Vertiefung wurde mit Tetramethylbenzidin (TMB) hinzugefügt, zwei Duplikate wurden für jede Konzentration eingestellt. Die Probenvertiefungen wurden mit 50 &mgr;l Probenverdünner verbunden und die Proben der Reihe nach. Jede Vertiefung wurde mit 100 µl primärem Antikörper (außer TMB-Vertiefung) 1 h lang sowie mit 300 µl 0,01 M Tris-gepufferter Kochsalzlösung (TBS) und 100 µl Avidin-Biotin-Peroxidase-Komplex-Arbeitslösung (außer TMB-Vertiefung) umgesetzt ). Dann wurde jede Vertiefung mit 300 &mgr;l 0,01 M TBS versetzt und mit 100 &mgr;l TMB inkubiert. Der Wert der optischen Dichte (OD) und die Konzentration jeder Vertiefung wurden sofort gemessen und die Standardkurve wurde gezeichnet.
Quantitative Reverse Transkription der Polymerase-Kettenreaktion (RT-qPCR)
MiR-133a, Collagen Ι, Collagen III, α-SMA, TGF-β1, CTGF, E-Cadherin und FSP-1-Expression in Myokardgeweben und Kardiomyozyten wurden mittels RT-qPCR nachgewiesen. Die Gesamt-RNA wurde aus Kardiomyozyten oder Myokardgewebe extrahiert und über einen RNA-Extraktionskit (Takara, Dalian, China) in cDNA revers transkribiert und RT-PCR-Primer wurden über Invitrogen (Guangzhou, China) synthetisiert, die Sequenzen sind in Tabelle 1 gezeigt. Die relative quantitative Genexpression wurde unter Verwendung von Glyceraldehyd-3-Phosphat-Dehydrogenase (GAPDH) oder U6 als Ladekontrollgene gemäß 2 −△△Ct . analysiert Methode.
Western Blot-Analyse
Die Ratten wurden unter Narkose euthanasiert. Die Myokardgewebe wurden eingefroren und in flüssigem Stickstoff gemahlen. Dann wurde die Stammlösung der Protease-Inhibitoren Phenylmethansulfonylfluorid mit Zelllysepuffer in einem Verhältnis von 1:100 (Beyotime Biotechnology Co., Ltd., Shanghai, China) gemischt. Die Proben wurden mit gemischter Lösung lysiert und Proteine der Zellen wurden extrahiert. Die Gesamtproteinkonzentration wurde mit dem BCA-Kit nachgewiesen. Die Proben wurden mit 5 × Beladungspuffer bei 4 :1 gemischt, in einem siedenden Wasserbad für 10 min umgesetzt, eisgebadet und zentrifugiert. Es wurde eine Elektrophorese-Trennung durchgeführt, und die Proteine wurden mit einer elektrischen Transferlösung auf eine Polyvinylidenfluorid-(Servicebio)-Membran übertragen. Dann wurde die Membran mit 5% Magermilchpulver blockiert und mit den Primärantikörpern CD63, CD81 und CD9 (polyklonale Kaninchen-Anti-Ratte-Antikörper von Proteintech, 1:100), MAML1 (ab65090, Abcam, MA, USA, 1:1000) und GAPDH (Santa Cruz Biotechnology, Inc., Santa Cruz, CA, USA, 1:1000). Dann wurde die Membran mit dem sekundären Antikörper, Meerrettichperoxidase-markiertem IgG (Cell Signaling Technology, Beverly, MA, USA, 1:1000) betropft und in eine verstärkte Chemilumineszenz-Reaktionslösung (Pierce, Rockford, IL, USA) getaucht. Mit GAPDH als Ladekontrolle wurden Protein-Imprinting-Bilder mit der ImageJ2x-Software analysiert.
Kardiomyozytenkultur und -passage
Es wurden SD-Ratten im Alter von 3–5 Tagen (Experimental Animal Center of Zhejiang University School of Medicine, Zhejiang, China) entnommen. Der ventrikuläre Teil wurde mit vorgekühlter Hank's Balanced Salt Solution gespült, in kleine Stücke geschnitten und mit 0,25% Trypsin abgelöst. Die Stücke wurden mit einer geeigneten Menge von 10 % Vollmedium zur Beendigung der Ablösung versetzt und bei 151 g zentrifugiert. DMEM, das 20 % FBS enthielt, wurde zur Zellresuspension verwendet. Kardiomyozyten wurden durch die differentielle Adhäsionsmethode gereinigt und die Überlebensrate wurde durch Trypanblau-Färbung beobachtet, die überlebten Kardiomyozyten wurden kultiviert. Nach 24 h hafteten die Kardiomyozyten an der Wand und begannen zu pulsieren. Nach 72 Stunden breiteten sich die Pseudopodien aus.
Konstruktion des VMC-Modells von Kardiomyozyten
Kardiomyozyten in der 4. Passage in der logarithmischen Wachstumsphase wurden selektiert und mit MSC Exo . infiziert , NC Exo , Adas-miR-133a Exo , und Ad-miR-133a Exo . Die 100 Tcid50 CVB3-Viruslösung (100 &mgr;l) wurde in die Zellen gegeben, um ein Zell-VMC-Modell zu induzieren. Gleichzeitig wurde den Zellen eine gleiche Menge an Erhaltungslösung als Kontrolle zugesetzt, und die entsprechenden Exosomen wurden nach 1-stündiger Infektion für 47-stündige Kultur zu Kardiomyozyten zusammengefügt.
Cell Counting Kit (CCK)-8 Assay
Das CCK-8-Zellnachweis-Kit (Beyotime) wurde verwendet, um die Überlebensrate von Kardiomyozyten nachzuweisen. Beim Wachstum bis zur logarithmischen Phase wurden die Zellen mit 0,25% Trypsin abgelöst und in eine 96-Well-Zellkulturplatte bei 2,5 × 10 4 . ausgesät Zellen/pro Vertiefung. Zusammen mit CCK-8-Lösung (10 μl/Well) wurden die Zellen kontinuierlich 1–4 h lang kultiviert und die OD450 nm Wert wurde mit einem Mikroplatten-Lesegerät gemessen.
Durchflusszytometrie
Die Doppelfärbungsmethode AnnexinV-APC/Propidiumiodid (PI) wurde angewendet, um Zellapoptose nachzuweisen. Die Zellen wurden zentrifugiert, mit 250 μl Bindungspuffer (4 ml Bindungspuffer + 12 ml entionisiertes Wasser) resuspendiert und auf 1 × 10 6 . eingestellt Zellen/ml. Die 100-μl-Zellsuspension wurde mit 5 μl Annexin V-APC (BD Biosciences) und 5 μl PI-Lösung (BD Biosciences) versetzt, auf das Durchflusszytometer geladen und automatisch von einem Computer analysiert.
Dualer Luciferase-Reporter-Gen-Assay
Die Wildtyp-(wt)- oder Mutanten-Typ-(mut)-Sequenz der MAML1-3-untranslatierten Region (UTR) wurde in den pGL3-M-Vektor (Promega, WI, USA) kloniert, dann MAML1-3-UTR-wt oder MAML1- 3-UTR-mut wurden generiert. Die Vektoren wurden zusammen mit miR-133a-Mimic oder NC über Lipofectamine 2000 in Kardiomyozyten cotransfiziert. Die Luciferase-Aktivität wurde 48 h später durch das duale Luciferase-Reportergensystem (Promega) getestet [21]
RNA-Immunpräzipitation (RIP)-Assay
Der RIP-Kit (Millipore, USA) wurde verwendet, um die Bindung von MAML1 und miR-133a nachzuweisen. Die Zellen wurden durch Radioimmunpräzipitationsassaypuffer (P0013B, Beyotime, Shanghai, China) lysiert, bei 1400 g zentrifugiert und mit Antikörpern zur Copräzipitation inkubiert. Magnetische Beads (50 µl) wurden in 100 µl RIP-Waschpuffer resuspendiert und mit 5 g Anti-MAML1-Antikörper (1 g/ml, ab155786) oder IgG (1:100, ab172730) inkubiert. Der magnetische Bead-Antikörper-Komplex wurde in 900 μl RIP-Waschpuffer resuspendiert, mit 100 μl Zellextrakt interagiert, mit Proteinase K verdaut und durch RT-qPCR nachgewiesen [22].
Statistische Analyse
Zur Analyse der Daten wurde die Statistiksoftware SPSS 21.0 (IBM Corp. Armonk, NY, USA) verwendet. Die Messdaten wurden als Mittelwert ± Standardabweichung ausgedrückt. Der t-Test wurde auf Vergleiche zwischen zwei Gruppen angewendet. Für den Vergleich zwischen den Gruppen wurde eine Einweg-Varianzanalyse (ANOVA) und für paarweise Vergleiche der Post-hoc-Test von Tukey verwendet. Prädiktoren wurden beibehalten, wenn sie bei einem P . signifikant waren Wert von 0,05 oder kleiner.
Ergebnisse
Identifizierung von BMSCs und BMSC-Exo
Mikroskopisch waren BMSCs spindelförmig und abgerundet und hafteten in einem Wirbel- oder Radialmuster an der Wand (Abb. 1A). Nach der Ölrot-O-Färbung der Adipogenese-Induktion waren die Lipidtröpfchen der 4. BMSCs rot und es gab runde Lipidtröpfchen unterschiedlicher Größe ( 1B ). Nach Induktion der Osteogenese zeigten die Zellen, die kalzifizierte Knötchen exprimierten, rot nach Alizarinrot-Färbung und eine ungleichmäßige Verteilung von kalzifizierten Knötchen und überlappenden Zellen ( 1C ). Die Durchflusszytometrie zeigte, dass die MSC-Marker CD29 und CD44 (> ~ 95 %) exprimiert wurden, aber das Oberflächenantigen CD34 der hämatopoetischen Stammzellen (<~ 95 %) wurde nicht exprimiert ( 1D ). Diese Ergebnisse zeigten, dass die BMSCs von hoher Reinheit waren und den MSC-Standards der International Society of Cell Therapy entsprachen.
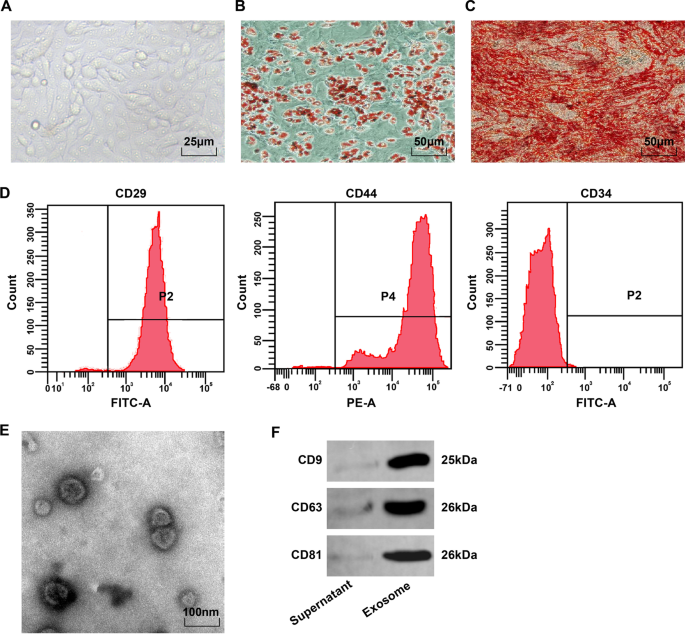
BMSC-Phänotyp-Beobachtung und BMSC-Exo-Identifizierung. A Morphologische Beobachtung von BMSCs in der 4. Passage; B Die Ergebnisse der Ölrot-O-Färbung von Adipozyten; C Die Ergebnisse der Alizarinrot-Färbung von Osteoblasten; D Der Phänotyp von BMSC wurde mittels Durchflusszytometrie nachgewiesen. E Elektronenmikroskopische Beobachtung von BMSC-Exo; F Proteinbanden von CD9, CD63 und CD81
Transmissionselektronenmikroskop beobachtete, dass BMSC-Exos ovale Vesikel mit klarer peripherer Membranstruktur, unterschiedlicher Größe und einem Durchmesser von 40–100 nm waren (Abb. 1E). Western-Blot-Analyse zeigte, dass die extrahierten Produkte, die Exosomen exprimierten, von charakteristischen Proteinen von CD9, CD63 und CD81 stammten (Fig. 1F).
Erhöhtes exosomales miR-133a verbessert die Symptome einer Myokarditis
Die Transfektion von BMSCs mit rekombinantem Adenovirus miR-133a wurde beobachtet ( 2A ). Unter dem invertierten Fluoreszenzmikroskop wurde eine große Anzahl grüner Fluoreszenzexpressionen von NC, Ad-miR-133a und Adas-miR-133a beobachtet, was darauf hindeutet, dass der rekombinante Adenovirus-Vektor BMSCs effektiv transfizieren kann. Um die Transfektionseffizienz von miR-133a zu testen, wurde die miR-133a-Expression in BMSCs und ihren Exosomen durch RT-qPCR gemessen. Es wurde entdeckt, dass die Hochregulation von miR-133a die Expression von miR-133a erhöht, während die Herunterregulierung von miR-133a die Expression von miR-133a verringert ( 2B ). Anschließend injizierten wir Exosomen, die miR-133a enthielten, in Ratten. Unter dem invertierten Fluoreszenzmikroskop wurde bei VMC-Ratten nach der Behandlung von NC Exo . eine grüne Fluoreszenzexpression beobachtet , Ad-miR-133a Exo , oder Adas-miR-133a Exo , was darauf hinweist, dass der rekombinante Adenovirus-Vektor Myokardgewebe von Ratten infizierte ( 2C ). Das RT-qPCR-Experiment ergab auch, dass die miR-133a-Expression bei VMC-Ratten anscheinend verringert war; Die miR-133a-Expression war offensichtlich bei VMC-Ratten erhöht, denen Ad-miR-133a Exo . injiziert wurde aber verringert bei VMC-Ratten, denen Adas-miR-133a Exo . injiziert wurde (Abb. 2D). Bezüglich des Allgemeinzustands von Ratten wurde beobachtet, dass der Allgemeinzustand normaler Kontrollratten normal war und die Charakteristika von VMC offensichtlich bei VMC-Ratten und VMC-Ratten, denen Adas-miR-133a Exo , wie raues und unordentliches Haar, Dyspnoe und wenig Diät. Bei VMC-Ratten, die mit MSC Exo . behandelt wurden , NC Exo , und Ad-miR-133a Exo , diese Zeichen wurden in unterschiedlichem Maße verbessert. Das Gewicht der VMC-Ratten wurde 1 Tag nach der Infektion und der Injektion von MSC Exo . kontinuierlich verringert , NC Exo , oder Ad-miR-133a Exo erhöhte das Gewicht der Ratten. Das Gewicht der mit Ad-miR-133a Exo . behandelten VMC-Ratten wurde offensichtlich erhöht und das Gewicht der VMC-Ratten mit Adas-miR-133a Exo . injiziert wurde deutlich verringert (Abb. 2E).
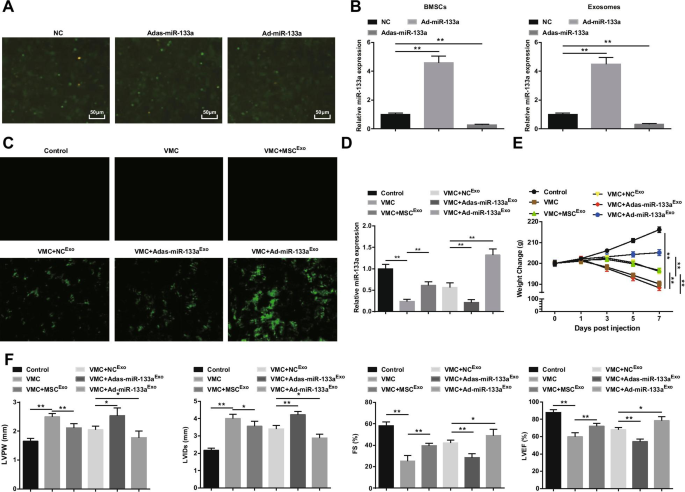
Hochreguliertes exosomales miR-133a lindert Myokarditis. A BMSCs-Transfektion von miR-133a rekombinantem Adenovirus; B RT-qPCR-Nachweis der miR-133a-Expression in BMSCs und deren Exosomen nach Regulierung von miR-133a; C miR-133a-Transfektionseffizienz getestet mit einem invertierten Fluoreszenzmikroskop; D Die relative Expression von miR-133a in Myokardgeweben wurde mittels RT-qPCR getestet; E Gewichtsveränderung der Ratten in jeder Gruppe; F Bestimmung von LVPW, LVIDs, FS und LVEF bei Ratten jeder Gruppe. *P <0,05; **P <0,001
Die Beobachtung der Myokardfunktion legte nahe, dass ( 2F ) die VMC-Ratten erhöhte LVPW und LVIDs hatten und verringerten FS und LVEF. Nach Exosom-Injektion zeigten sich bei VMC-Ratten eine Abnahme von LVPW und LVIDs und offensichtlich erhöhte FS und LVEF. Adas-miR-133a Exo Behandlung beeinträchtigt während Ad-miR-133a Exo verbesserte Myokardfunktion bei VMC-Ratten.
Hochreguliertes exosomales miR-133a hemmt Entzündungen im Myokardgewebe von VMC-Ratten
Die HE-Färbung zeigte, dass die Myokardfasern bei normalen Kontrollratten eng angeordnet waren und es keine Entzündungszellinfiltration in das Mesenchym gab. Die Kardiomyozyten der VMC-Ratten waren desorganisiert und das Mesenchym war von einer Vielzahl von Entzündungszellen infiltriert. Die Kardiomyozyten von VMC-Ratten, denen MSC Exo . injiziert wurde oder NC Exo waren geordnet angeordnet, wobei eine kleine Menge entzündlicher Zellen in das Mesenchym eindrang. Die Kardiomyozyten bei VMC-Ratten nach Adas-miR-133a Exo Behandlung war ungeordnet angeordnet und die Entzündungszellen im Mesenchym wurden infiltriert. Die Kardiomyozyten bei VMC-Ratten, die mit Ad-miR-133a Exo . behandelt wurden wurden ohne offensichtliche Infiltration von Entzündungszellen geordnet angeordnet (Abb. 3A).
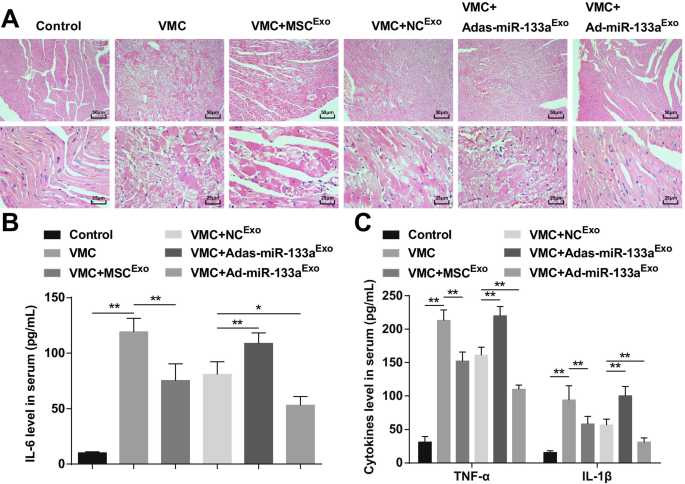
Erhöhtes exosomales miR-133a hemmt Entzündungen im Myokardgewebe mit VMC. A HE-Färbung von Rattenmyokardgewebe in jeder Gruppe; B Die Expression von IL-6 im Serum wurde mittels ELISA getestet; C Die Expression von TNF-α und IL-1β im Serum wurde mittels ELISA getestet. *P <0,05; **P <0,001
ELISA zeigte, dass ( 3B , C) Entzündungsfaktoren (TNF-&agr;, IL-1&bgr; und IL-6) bei VMC-Ratten offensichtlich erhöht waren. Den VMC-Ratten wurde Ad-miR-133a Exo . injiziert hatte reduzierte Entzündungsfaktoren. Adas-miR-133a Exo Behandlung verursachte erhöhte Entzündungsfaktoren bei VMC-Ratten.
Erhöhtes exosomales miR-133a verringert CVF im Myokardgewebe von Ratten mit VMC
Die Masson-Färbung ergab, dass die Myokardfasern bei normalen Ratten eng angeordnet waren und fast keine blauen Kollagenfasern aufwiesen. Nach CVB3-Injektion waren die Kardiomyozyten hypertroph, mit Bindegewebshyperplasie und einer großen Anzahl blauer kollagener Fasern, und CVF war offensichtlich erhöht. Mit Exosomen behandelt, waren die Kardiomyozyten geordnet angeordnet, die interzelluläre Bindegewebshyperplasie war verringert, blaue Kollagenfasern und CVF waren deutlich verringert. Der myokardiale Interzellularraum von VMC-Ratten, denen Adas-miR-133a Exo . injiziert wurde war verbreitert, die Zellen waren offensichtlich vergrößert, die blauen Kollagenfasern und CVF waren deutlich erhöht; der Interzellularraum war verringert, die Verteilung der blauen Kollagenfasern und der CVF waren bei VMC-Ratten mit Ad-miR-133a Exo . verringert Behandlung (Abb. 4A, B).
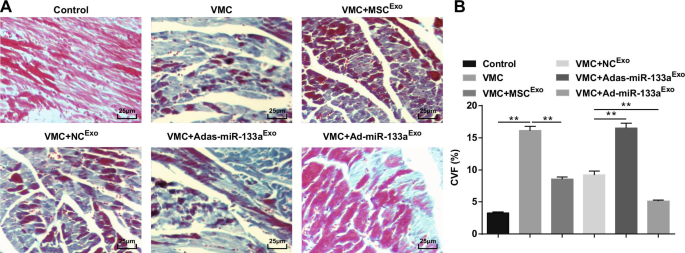
Hochreguliertes exosomales miR-133a verringert CVF im Myokardgewebe von Ratten mit VMC. A Masson-Färbung von Myokardgewebe bei Ratten; B Kollagenvolumenfraktion von Ratten in jeder Gruppe. **P <0,001
Erhöhtes exosomales miR-133a reduziert die Expression von Collagen I, Collagen III, TGF-β1 und CTGF im Myokardgewebe von Ratten mit VMC
Kollagen I und Kollagen III sind die Hauptbestandteile von Kollagen, die hauptsächlich in Zellverbindungen und Zellmembranen, in der Interzellularsubstanz und im Zytoplasma verteilt sind. TGF-β1 und CTGF sind die charakteristischen Proteine der Fibrose. Die Ergebnisse der RT-qPCR zeigten, dass die Expressionsspiegel von Collagen I, Collagen III, TGF-β1 und CTGF-mRNA bei VMC-Ratten erhöht waren, aber nach Exosomenbehandlung abnahmen. Die mit Ad-miR-133a Exo . behandelten VMC-Ratten hatten verringerte mRNA-Expressionsspiegel von Kollagen I, Kollagen III, TGF-β1 und CTGF, während die VMC-Ratten nach Adas-miR-133a Exo Behandlung zeigte die gegenteilige Situation (Abb. 5A–C).
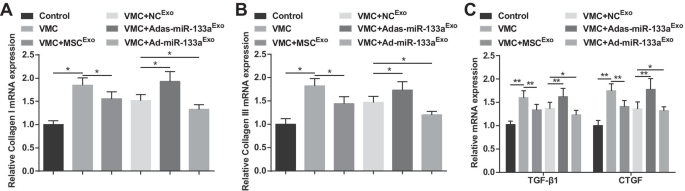
Elevated exosomal miR-133a reduces the mRNA expression of collagen I, collagen III, TGF-β1 and CTGF in myocardial tissues of rats with VMC. A Collagen I mRNA expression in myocardial tissues of rats was detected by RT-qPCR; B Collagen III mRNA expression in myocardial tissues of rats was detected by RT-qPCR; C TGF-β1 and CTGF mRNA expression in myocardial tissues of rats was detected by RT-qPCR. *P < 0.05; **P <0,001
Up-regulated Exosomal miR-133a Inhibits the Cardiomyocyte Apoptosis in Myocardial Tissues of Rats with VMC
TUNEL staining showed that the apoptotic cardiomyocytes were brownish black or brownish yellow with nuclear condensation. The number of apoptotic cells was increased in VMC rats which would be attenuated by exosome treatment. The VMC rats injected with Ad-miR-133a Exo had reduced number of apoptotic cells and those injected with Adas-miR-133a Exo had increased number of apoptotic cells (Fig. 6A, B).
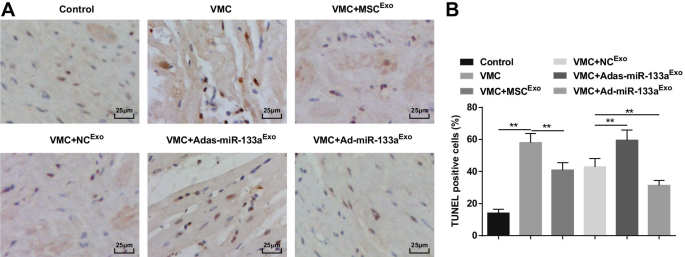
Increased exosomal miR-133a inhibits the cardiomyocyte apoptosis in myocardial tissues of rats with VMC. A TUNEL staining of rat myocardial tissues in each group; B The number of TUNEL positive cells in each group. **P <0,001
Elevated Exosomal miR-133a Depresses EMT in Myocardial Tissues of Rats with VMC
E-cadherin, α-SMA, and FSP-1 are key indicators of EMT. Results of RT-qPCR demonstrated that α-SMA and FSP-1 mRNA expression levels were elevated and E-cadherin mRNA expression level was decreased in VMC rats. In addition, α-SMA and FSP-1 mRNA expression levels were reduced and E-cadherin mRNA expression level was increased in VMC rats after exosome treatment. α-SMA and FSP-1 mRNA expression levels were elevated and E-cadherin mRNA expression level was decreased in VMC rats treated with Adas-miR-133a Exo , while the expression of these indicators was opposite in VMC rats injected with Ad-miR-133a Exo (Fig. 7A, B).
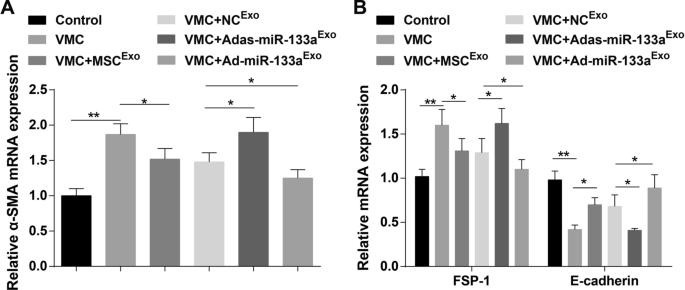
Up-regulated exosomal miR-133a represses EMT in myocardial tissues of rats with VMC. A The mRNA expression of α-SMA in rat myocardial tissues in each group was detected by RT-qPCR; B The mRNA expression of FSP-1 and E-cadherin in rat myocardial tissues in each group was detected by RT-qPCR. *P < 0.05; **P <0,001
Up-regulated Exosomal miR-133a Depresses Inflammation of Cardiomyocytes in VMC
As a result, fluorescence microscopy captured green fluorescent expression in VMC rats treated with NC Exo , Ad-miR-133a Exo , or Adas-miR-133a Exo , indicating that the recombinant adenovirus vector infected cardiomyocytes of rats (Fig. 8A). RT-qPCR and ELISA discovered that (Fig. 8B, D) miR-133a expression was reduced and inflammatory factors (TNF-α, IL-1β, and IL-6) were increased in VMC rats, which would be reversed by exosome treatment. The VMC rats treated with Ad-miR-133a Exo had up-regulated miR-133a and decreased inflammatory factors in VMC rats, while those treated with Adas-miR-133a Exo presented decreased miR-133a and increased levels of inflammatory factors in VMC rats.
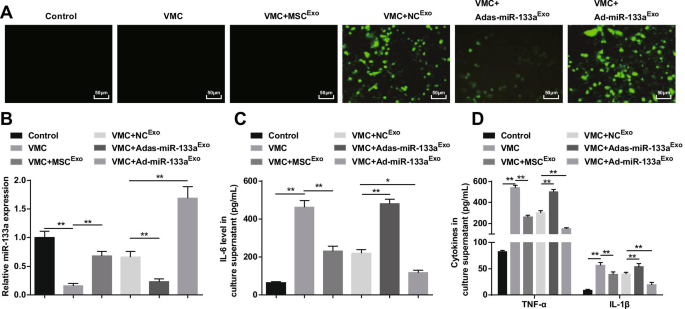
Elevated exosomal miR-133a restrains inflammation of cardiomyocytes in VMC. A miR-133a transfection efficiency tested via inverted fluorescence microscope; B The relative expression of miR-133a in cardiomyocytes of rats in each group; C IL-6 expression in culture supernatant of cardiomyocytes in each group; D TNF-α and IL-1β expression in culture supernatant of cardiomyocytes in each group. *P < 0.05; **P <0,001
Elevated Exosomal miR-133a Promotes Cell Viability, and Represses Apoptosis of Cardiomyocytes in VMC
The apoptosis and the cell viability were detected via AnnexinV-APC/PI double staining and CCK-8 assay. The results revealed that there was an obvious increase in apoptosis rate, a decrease in cell viability of cardiomyocytes in VMC rats. Exosome treatment reduced apoptosis rate and enhanced the viability of cardiomyocytes. Adas-miR-133a Exo enhanced the apoptosis rate and disrupted the viability of cardiomyocytes in VMC rats. Ad-miR-133a Exo treatment functioned the opposite effects on cardiomyocytes of VMC rats (Fig. 9A–C).
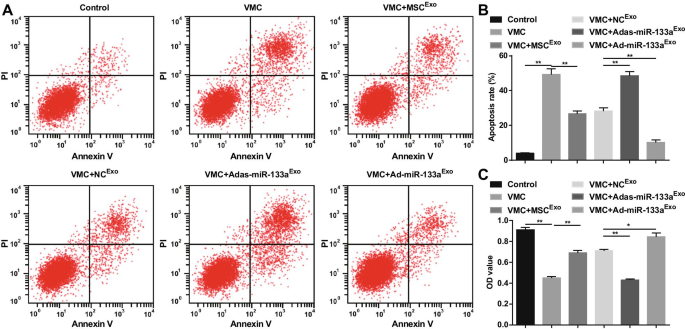
Increased exosomal miR-133a promotes viability and represses apoptosis in cardiomyocytes in VMC. A The cardiomyocytes apoptosis detected via flow cytometry; B Quantification results of A; C The cell viability detected via CCK-8 assay. *P < 0.05; **P <0,001
miR-133a Targets MAML1
It has been reported that up-regulated miRNA-193b reduces myocardial I/R damage by targeting MAML1 [18]. Based on that, we cross-screened downstream genes of miR-133a through bioinformatics websites PITA, miRanda, PicTar, microT and miRmap, and selected MAML1 as a target of miR-133a (Fig. 10A). We constructed MAML1-wt or MAML1-mut, and co-transfected cardiomyocytes with miR-133a mimic or NC. The results showed that miR-133a mimic reduced the luciferase activity of MAML1-wt (Fig. 10B). The RIP experiment further verified the targeting relationship between miR-133a and MAML1 (Fig. 10C). RT-qPCR and Western blot detection of MAML1 expression showed that MAML1 expression was decreased in cardiomyocytes transfected with miR-133a mimic (Fig. 10D, E).
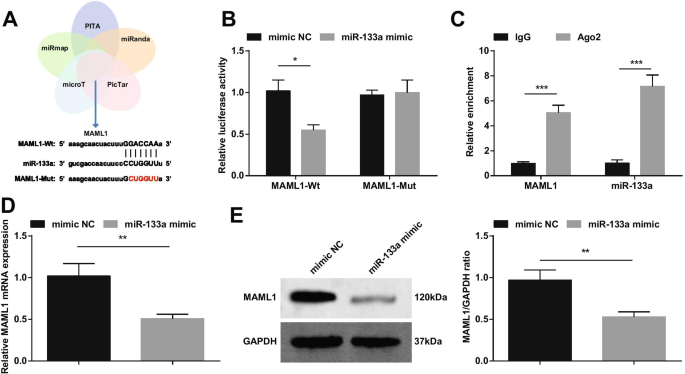
miR-133a targets MAML1. A miR-133a’s targets predicted on bioinformatics websites; B The targeting relationship between miR-133a and MAML1 verified by dual luciferase reporter gene experiment; C The targeting relationship between miR-133a and MAML1 verified by RIP experiment; D /E MAML1 expression changes after up-regulation of miR-133a detected by RT-qPCR and Western blot. *P < 0.05; **P < 0.01; ***P <0,001
Inhibition of MAML1 has a Protective Effect on Rats with Myocarditis and Reverses the Effect of miR-133a-Inhibited Exosomes on Rats with VMC
To further study the effect of miR-133a-regulated MAML1 on rats with VMC, we injected si-MAML1 or si-NC adenovirus into VMC rats or VMC rats that had been treated with miR-133a-silenced exosomes. The injection success was validated by RT-qPCR and Western blot (Fig. 11A, B). The results manifested that injection of si-MAML1, the weight of VMC rats was increased (Fig. 11C), cardiac function was improved (Fig. 11D–G), myocardial tissue pathology and fibrosis were attenuated (Fig. 12A–C), serum inflammation (Fig. 12D, E) and cardiomyocyte apoptosis (Fig. 13A–G) were inhibited. Also, the deleterious effects of miR-133a-silenced exosomes in VMC rats were reversed after injection of si-MAML1.
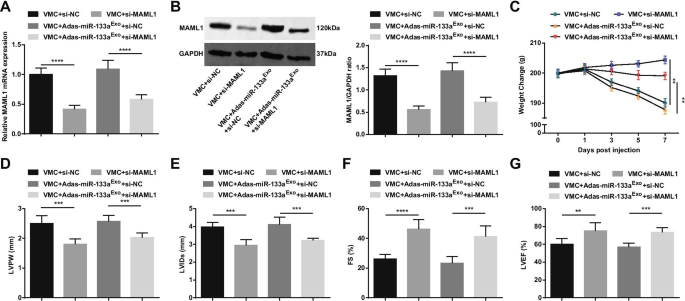
Inhibition of MAML1 has a protective effect on myocarditis rats and can reverse the effect of miR-133a-silenced exosomes on rats with VMC. A /B MAML1 expression in myocardial tissue of rats detected by RT-qPCR and Western blot; C. Weight change of rats; D –G Determination of LVPW, LVIDs, FS and LVEF in rats; **P < 0.01; ***P < 0,001; ****P < 0.0001
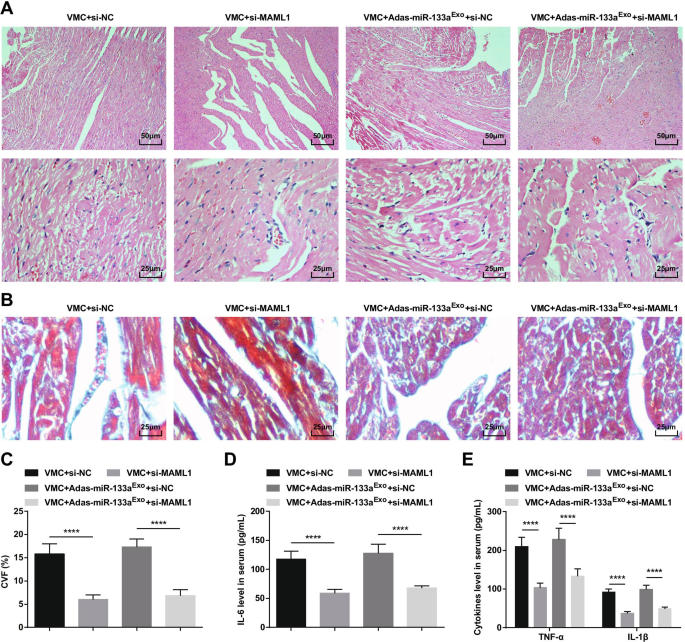
Inhibition of MAML1 can reverse the effect of miR-133a-silenced exosomes on rats with VMC. A HE staining of rat myocardial tissue; B Masson staining of myocardial tissues in rats; C CVF of rats; D The expression of IL-6 in serum tested via ELISA; E The expression of TNF-α and IL-1β in serum tested via ELISA.****P < 0.0001
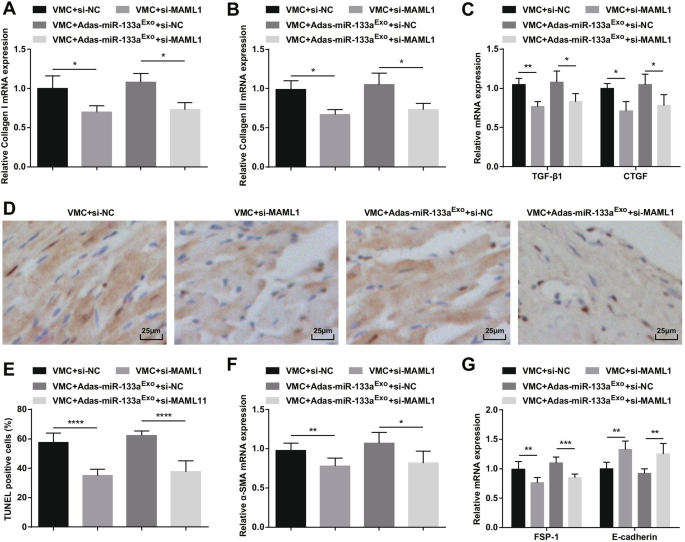
Inhibition of MAML1 can reverse the effect of miR-133a-inhibiting exosomes on rats with VMC. A Collagen I mRNA expression in myocardial tissues of rats was detected by RT-qPCR; B Collagen III mRNA expression in myocardial tissues of rats was detected by RT-qPCR; C TGF-β1 and CTGF mRNA expression in myocardial tissues of rats was detected by RT-qPCR; D TUNEL staining of rat myocardial tissues in each group; E The number of TUNEL positive cells in each group. F The mRNA expression of α-SMA in rat myocardial tissues in each group was detected by RT-qPCR; G The mRNA expression of FSP-1 and E-cadherin in rat myocardial tissues in each group was detected by RT-qPCR. *P < 0.05; **P < 0.01; ****P < 0.0001
Discussion
Myocarditis is an inflammatory heart illness resulting in DCM and heart failure and is most frequently induced by viral infections such as CVB3 [2]. A study has revealed that miR-133 relieves cardiomyocyte apoptosis and electrical remodeling in mice with VMC [23]. Additionally, changed exosomal miRNAs are also found to be linked with the pathogenesis of CVB3-induced myocarditis [14]. Exosomes derived from cardiac progenitor cells ease CVB3-induced apoptosis via restraining the proliferation of CVB3 in VMC [24]. This study explored the regulatory mechanism of BMSC-derived exosomal miR-133 on myocardial fibrosis and EMT in VMC rats (Additional file 1:Fig. 1).
The study found that the expression of miR-133a was decreased in VMC. As demonstrated before, miR-133a expression is decreased in MI [7]. A study has also suggested that the relative expression of miR-133 in mouse hearts of the VMC is obviously decreased with contrast to the controls [23]. There are some connections of miRNAs with exosomes. The differential expression of exosomes and of exosomal miRNAs in illness has been regarded as biomarkers of disease with performance of noninvasive clinical diagnosis together with their therapeutic potentials [25]. Lin et al. have found that miR-133 is specially sorted into hypoxia/reoxygenation (H/R)-caused human endothelial progenitor cells-derived exosomes to increase fibroblast angiogenesis and EMT [26]. Another study has revealed that MSCs exhibits a communication with brain parenchymal cells and may modulate neurite outgrowth by transfer of miR-133b to neural cells via exosomes [27].
The major finding of this work manifested that up-regulated exosomal miR-133a promoted cell viability, inhibited inflammation, apoptosis, EMT, and fibrosis in rats with VMC. They suits well with a former research that miR-133a silence reverses the Astragalus polysaccharides treatment-induced osteosarcoma MG63 cell proliferation inhibition, together with cell apoptosis promotion [28]. Another study has revealed that overexpressed miR-133a suppresses angiogenesis, apoptosis, fibrosis, and inflammation, while accelerating therapeutic cardiac remodeling in ischemic myocardial illnesses [29]. Similar to our study, Li et al. have stated that miR-133 inhibits cardiomyocyte apoptosis by regulating the expression of apoptosis-related genes in the hearts of VMC mice [23]. The over-expressed miR-133a has been reported to depress hypoxia-induced apoptosis and strengthen cardiomyocyte survival [30]. Meanwhile, the up-regulated serum exosomal miR-30a and miR-181d may have the potentials to be applied as biomarkers for VMC diagnosis [14].
Another finding in our study was that up-regulated exosomal miR-133a decreased CVF, reduced the expression of collagen I and collagen III in rats with VMC. A article has elucidated that released fibroblast growth factor-18 from a collagen membrane causes osteoblastic activity participating in down-regulated miR-133a [31]. In vitro excessive expression of miR-133a depresses cardiomyocyte hypertrophy and reduces collagen expression [32], as evidenced in another study. CVF equals the ratio of collagen area to the sum of myocardial area and collagen area, and the mean value shows the CVF of the section [33]. This finding is also reported by Wang et al. that VMC mice model is successfully constructed by CVB3 infection, manifesting apparent higher CVF expression in contrast with the control group [34]. Moreover, the finding is consistent with that of Ferreira et al. who demonstrates that miR-133a may take on a major role in the modulation of gene expression in chronic Chagas disease cardiomyopathy pathogenesis, with potential link as diagnostic and prognostic tools [8]. Furthermore, evidence has shown that knocking down MAML1 can reduce the hypertrophy of pre-treated cardiomyocytes [35]. In our study, we found that MAML1 was the target gene of miR-133a and inhibition of MAML1 reversed the effects of miR-133a-silenced exosomes on rats with VMC. In myocardial ischemia–reperfusion injury, miR-193b-mediated down-regulation of MAML1 could in part reduce infarction and myocardial enzymes, as well as attenuate apoptosis of cardiomyocytes [18]. Also, there is a report suggesting that deficiency of MAML1 could relieve hepatic fibrogenesis [19].
Conclusion
In conclusion, this present study offers evidence that miR-133a is down-regulated in rats with VMC, and elevated exosomal miR-133a improves cardiac function and restrains myocardial fibrosis and EMT in rats with VMC, as well as enhances viability and represses apoptosis of cardiomyocytes in VMC through targeting MAML1. Our study also suggests that inhibition of MAML1 has a protective effect on rats with myocarditis and reverses the effect of miR-133a-inhibited exosomes on rats with VMC. The identification of the exosomal miR-133a in myocardial fibrosis and EMT of myocarditis may potentially widen our understanding of mechanisms underpinning myocarditis and also bear clinical value as a novel molecular target. More researches should be undertaken for making inroads into the treatment of this disease.
Abkürzungen
- miR-133:
-
MicroRNA-133
- BMSC-Exo:
-
Bone marrow mesenchymal stem cell-derived exosome
- EMT:
-
Epithelial–mesenchymal transition
- VMC:
-
Viral myocarditis
- CVF:
-
Collagen volume fraction
- DCM:
-
Dilated cardiomyopathy
- CVB3:
-
Coxsackie B3 virus
- miRNAs:
-
MicroRNAs
- MI:
-
Myocardial infarction
- SPF:
-
Specific pathogen-free
- SD:
-
Sprague–Dawley
- DMEM:
-
Dulbecco’s Modified Eagle Medium
- PBS:
-
Phosphate-buffered saline
- FBS:
-
Fötales Rinderserum
- NC:
-
Negative control
- MOI:
-
Multiplicity of infection
- LVPW:
-
Left ventricular posterior wall thickness
- LVIDs:
-
Left ventricular end-systolic diameter
- LVEF:
-
Left ventricular ejection fraction
- HE:
-
Hematoxylin–eosin
- TUNEL:
-
Terminal deoxynucleotidyl transferase-mediated deoxyuridine triphosphate-biotin nick end-labelin
- ELISA:
-
Enzyme-linked Immunosorbent Assay
- TMB:
-
Tetramethylbenzidine
- TBS:
-
Tris-buffered saline
- ABC:
-
Avidin–Biotin-Peroxidase Complex
- OD:
-
Optische Dichte
- RT-qPCR:
-
Reverse transcription quantitative polymerase chain reaction
- GAPDH:
-
Glyceraldehyde-3-phosphate dehydrogenase
- CTGF:
-
Connective tissue growth factor
- CCK:
-
Cell counting kit
- PI:
-
Propidium iodide
- ANOVA:
-
Analysis of variance
Nanomaterialien
- Schwarze Phosphor-Nanopartikel fördern die osteogene Differenzierung von EMSCs durch hochregulierte TG2-Expression
- Struktur und elektronische Eigenschaften von übergangsmetalldotiertem Kaolinit-Nanoton
- Aktivkohlefasern, die von Ag Nanohair durch Selbstmontage und schnelles thermisches Glühen „dick überwachsen“ sind
- Untersuchung der Adhäsionskraft und des Glasübergangs dünner Polystyrolfolien durch Rasterkraftmikroskopie
- TEM- und STEM-Studien zu den Querschnittsmorphologien von Dual-/Tri-Layer-Breitband-SiO2-Antireflexfolien
- Eine vergleichende In-vivo-Untersuchung biosynthetisierter Kupfer- und Zinkoxid-Nanopartikel durch intraperitoneale und intravenöse Verabreichungswege bei Ratten
- Entwicklung und Charakterisierung von Sr-haltigen Glaskeramik-Kompositen auf Basis von biogenem Hydroxyapatit
- Materialien der Graphenfamilie in der Knochengeweberegeneration:Perspektiven und Herausforderungen
- Rasterkraftmikroskopie-basierte Nanoskopie von chondrogen differenzierenden menschlichen Fettstammzellen:Nanostruktur und Integrin-β1-Expression
- Preußisch-Blau-Nanopartikel-markierte mesenchymale Stammzellen:Bewertung von Zelllebensfähigkeit, Proliferation, Migration, Differenzierung, Zytoskelett und Proteinexpression in vitro



