SOX2 reguliert die lncRNA CCAT1/MicroRNA-185-3p/FOXP3-Achse, um die Proliferation und Selbsterneuerung von Gebärmutterhalskrebs-Stammzellen zu beeinflussen
Zusammenfassung
Es wurde die Rolle von langen nicht-kodierenden RNAs (lncRNAs) bei Gebärmutterhalskrebs (CC) dargestellt. Unser Ziel ist es, die Wirkung der geschlechtsbestimmenden Region Y-Box 2 (SOX2)/lncRNA Dickdarmkrebs-assoziiertes Transkript-1 (CCAT1)/microRNA-185-3p (miR-185-3p)/Forkhead Box Protein 3 (FOXP3 .) zu diskutieren ) zur Proliferation und Selbsterneuerungsfähigkeit von CC-Stammzellen. Die Expressionen von MiR-185-3p, SOX2, CCAT1 und FOXP3 wurden in CC-Geweben und -Zellen getestet. Die Beziehung zwischen SOX2/CCAT1-Expression und klinisch-pathologischen Merkmalen bei CC-Patienten wurde überprüft. Funktionsverlust- und Funktionsgewinnuntersuchungen wurden bei CD44 + . durchgeführt HeLa-Zellen, um biologische Funktionen und Selbsterneuerungskapazität zu diskutieren. Schließlich wurden die Beziehungen zwischen SOX2, CCAT1, FOXP3 und miR-185-3p überprüft. Die miR-185-3p-Expression war verringert, während die SOX2-, CCAT1- und FOXP3-Expression in CC-Geweben und -Zellen erhöht war. SOX2- und CCAT1-Expressionen wurden mit der Tumorgröße, Lymphknotenmetastasierung und dem CC-Stadium der Internationalen Föderation für Gynäkologie und Geburtshilfe in Verbindung gebracht. Das Herunterregulieren von SOX2 oder CCAT1 und das Hochregulieren von miR-185-3p führten zu einer Hemmung der Proliferation, Invasion, Migration und Zellsphärenzahl sowie zu einer Beschleunigung der Apoptose von CD44 + HeLa-Zellen. SOX2 konnte an CCAT1 binden, was die Expression von miR-185-3p beeinflusste, und FOXP3 wurde von miR-185-3p angegriffen.
Einführung
Gebärmutterhalskrebs (CC) ist mit geschätzten 570.000 Fällen und 311.000 Todesfällen weltweit im Jahr 2018 die vierthäufigste Todesursache bei Frauen [1]. Diese komplexe Krankheit ist an einer Vielzahl von Faktoren beteiligt, darunter genetische Auswirkungen und Virusinfektionen [2]. Mit der Entwicklung des Human-Papillomavirus-Co-Tests und der Human-Papillomavirus-Impfung führen Frühdiagnostiken von zervikaler Dysplasie und Krebs zu einer Verringerung der Inzidenz, Morbidität und Mortalität von CC [3]. Bei frühen CC-Patienten wird eine Operation empfohlen, wie z. B. fertilitätserhaltende Chirurgie, Konusbiopsie, radikale Trachlektomie, Beckenlymphknotendissektion, Beckenbestrahlung und Brachytherapie [4]. Aufgrund von Metastasen oder Rezidiven bei fortgeschrittenen CC-Patienten ist die Prognose noch immer schlecht [5]. Daher ist es nach wie vor dringend erforderlich, neue und wirksame prognostische Marker und therapeutische Strategien zu identifizieren, um die Behandlung von CC zu verbessern.
Die geschlechtsbestimmende Region Y-Box 2 (SOX2) ist ein kritisches Mitglied der Transkriptionsfaktor-SOX-Familie und wird hauptsächlich in embryonalen und adulten Stammzellen manifestiert und auch in Tumorstammzellen exprimiert [6]. Es hat sich gezeigt, dass SOX2 die Radioresistenz bei CC durch den Hedgehog-Signalweg moduliert [7]. Eine andere Studie hat gezeigt, dass SOX2 entscheidend für den Erhalt der Subpopulation von Krebsstammzellen in CC-Zelllinien ist [8]. Lange nicht-kodierende RNAs (lncRNAs) sind eine Klasse von RNA-Molekülen mit einer Länge von 200 Nukleotiden [9]. Das LncRNA-Kolonkarzinom-assoziierte Transkript-1 (CCAT1) befindet sich auf dem menschlichen Chromosom 8q24.21 und gilt als „Hot Spot“, der zu genetischen Mutationen bei Dickdarmkrebs führt [10]. Eine Studie hat berichtet, dass CCAT1 die Zellproliferation und Invasion von CC beschleunigt [11]. Laut Jia et al. verstärkt CCAT1 dramatisch die Proliferation, Migration und Invasion von CC-Zellen [12]. Darüber hinaus hat eine andere Studie gezeigt, dass CCAT1 den malignen Grad einer entzündlichen Darmerkrankung erhöht, indem es die Darmbarriere zerstört, indem es microRNA-185-3p (miR-185-3p) reduziert [13]. MiRNA kann die Expression von Genen umgekehrt kontrollieren, indem sie mRNA reduziert und die Translation unterdrückt [14]. In-vitro-Experimente in einer früheren Studie haben gezeigt, dass miR-185-3p die Strahlenresistenz von Nasopharynxkarzinomen moduliert [15]. Eine andere Studie hat impliziert, dass miR-185 in vivo und in vitro an der Cisplatin-Resistenz von Eierstockkrebs beteiligt ist [16]. Forkhead Box Protein 3 (FOXP3) ist ein Transkriptionsfaktor aus der Familie der FOX-Proteine, der zuerst in regulatorischen T (Treg)-Zellen gefunden wird und eine entscheidende Rolle bei der Erhaltung und dem Prozess von Treg-Zellen spielt [17]. Eine Studie berichtet, dass FOXP3 mit der Lymphangiogenese von CC in Verbindung steht [18]. Eine andere Studie zeigt, dass der FOXP3-Spiegel dramatisch mit dem Stadium der Internationalen Föderation für Gynäkologie und Geburtshilfe (FIGO-Stadium) und der Tumorgröße von CC verbunden ist [19]. In dieser Studie haben wir daher die Auswirkungen der SOX2/CCAT1/miR-185-3p/FOXP3-Achse auf die Proliferation und die Selbsterneuerungsfähigkeit von CC-Stammzellen untersucht.
Materialien und Methoden
Ethik-Genehmigung und Zustimmung zur Teilnahme
Die Experimente mit Menschen wurden in Übereinstimmung mit den in der Deklaration von Helsinki formulierten Prinzipien durchgeführt. Die Studie wurde vom Institutional Review Board des First Hospital der Jilin University genehmigt. Alle Teilnehmer haben eine Einwilligungserklärung unterschrieben.
Studienfächer
Von Dezember 2016 bis Dezember 2018 wurden 39 Fälle von CC-Geweben und entsprechenden angrenzenden Normalgeweben von CC-Patienten entnommen und in flüssigem Stickstoff konserviert. Die Einschlusskriterien waren wie folgt:(1) Patienten wurden als CC durch Pathologie von Biopsien in transzervikalen und zervikalen Kanälen, Zervikalabstrichzytologie, Zervikaljodtest, Vaginalspekulum und Halswirbelresektion bestätigt. (2) Die Patienten erhielten 2 Wochen vor der Operation keine Strahlen- und Chemotherapie. Die Ausschlusskriterien waren wie folgt:(1) Patienten, die sich einer Strahlen- oder Chemotherapie unterziehen, (2) Patienten waren mit der Probenentnahme nicht einverstanden und (3) Patienten mit Erkrankungen des Immunsystems.
Zellauswahl und -kultur
CC-Zelllinien (SiHa, HeLa, CaSki, HCC94 und C33A) und immortalisierte humane zervikale Epithelzelllinie H8 wurden von Shanghai Bioleaf Biotech Co., Ltd. (Shanghai, China) gekauft. CC-Zellen SiHa, HeLa und HCC94 wurden in Dulbecco's Modified Eagle Medium (DMEM) mit hohem Glukosegehalt, das 10 % fötales Rinderserum (FBS) enthielt, kultiviert, während CaSki-, C33A- und H8-Zellen in Roswell Park Memorial Institute (RPMI)-1640-Medium mit 10 % FBS (37℃ und 5 % CO2 ). Die Zellen wurden alle 2 Tage abgelöst und subkultiviert.
CC-Stammzellensortierung (CD44 + HeLa-Zellen)
CC-Stammzellen wurden durch Zellsuspensionskultur von der CC-Zelllinie HeLa abgetrennt. CC-Zellen HeLa wurden mit serumfreiem Iscove’s Modified Dulbecco’s Medium (IMDM) in einer Ultra-Low-Adhäsions-Petrischale für 21 Tage kultiviert, wobei das Medium alle 3–5 Tage semiquantitativ ausgetauscht wurde. Einige Zellen wurden in Kugeln suspendiert und HeLa-Kugeln bildende Zellen (SFCs) wurden erhalten. Die Eigenschaft kugelförmiger Zellen wurde identifiziert und analysiert. Die Zellen wurden durch Trypsin abgelöst und auf 1 × 10 6 . eingestellt Zellen/ml. Zellen wurden mit CD44-Antikörper versetzt und durch Durchflusszytometrie sortiert. HeLa-Zellen mit positivem CD44 waren HeLa-Tumorstammzellen, während mit negativem CD44 HeLa-Nicht-Stammzellen waren. CC-Stammzellen wurden in DMEM/F12 kultiviert und mit 20 ng/ml basischem Fibroblasten-Wachstumsfaktor (bFGF), 20 ng/ml epidermaler Wachstumsfaktor (EGF) und B27 angehängt. Das Medium enthielt 1 % Penicillin und Streptomycin [20].
Zellbehandlung
CD44 + HeLa-Zellen wurden transfiziert mit sh-SOX2, sh-SOX2 Negativkontrolle (NC), sh-CCAT1, sh-CCAT1 NC, miR-185-3p Mimic, Mimic NC, sh-CCAT1 und miR-185-3p Inhibitor sowie sh-CCAT1 und Inhibitor NC. Alle Oligonukleotidsequenzen wurden von GenePharma (Shanghai, China) geliefert. Abgelöst durch Trypsin wurden die Zellen in einer 6-Well-Platte mit 3 × 10 6 . ausgesät Zellen/Well. Bei Erreichen einer Konfluenz von 60 % wurden die Zellen auf serumfreies Medium umgestellt und 1 h inkubiert. Die Transfektion wurde durch das Transfektionsreagenz Lipofectamine 2000 (Invitrogen, Carlsbad, Kalifornien, USA) erleichtert.
Quantitative Reverse Transkription der Polymerase-Kettenreaktion (RT-qPCR)
RNA in Geweben und Zellen wurde mit Trizol (Invitrogen) extrahiert. RNA (1 μg) wurde mit dem RTase-Kit für das Moloney-Maus-Leukämievirus (Invitrogen) in cDNA umgewandelt. cDNA wurde dem Echtzeit-PCR-System zugesetzt. Primer wurden von Shanghai Sangon Biotechnology Co., Ltd. (Shanghai, China) entwickelt (Tabelle 1). U6 war die Ladekontrolle von miR-185-3p, während Glycerinaldehydphosphatdehydrogenase (GAPDH) von SOX2, CCAT1 und FOXP3 war. Die Ergebnisse wurden nach 2 −ΔΔCt . analysiert Methode.
Western Blot Assay
Das Gesamtprotein in Zellen und Geweben wurde extrahiert. Die Proteinkonzentration wurde mit Bicinchoninsäure-Kits (AmyJet Scientific, Wuhan, Hubei, China) bestimmt. Das Protein wurde mit Ladepuffer gemischt und 5 Minuten gekocht, gefolgt von einem Eisbad und einer Zentrifugation. Das Protein wurde mit 10 % Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese behandelt und auf eine Membran übertragen. Die Membran wurde mit 5% Magermilch für 1 h blockiert, mit Primärantikörpern SOX2 (1:1000, Jiangsu Rui sitan Co., Ltd., Jiangsu, China), FOXP3 (1:1000, Abcam Inc., Cambridge, MA) sondiert , USA), GAPDH (1:1000, Cell Signaling Technology, Beverly, MA, USA) und erneut mit Meerrettichperoxidase markiertem sekundärem Antikörper sondiert, mit einem Konservierungsfilm bedeckt und exponiert. Bio-rad Gel Doc EZ Imager (Bio-rad, Kalifornien, USA) wurde für die Entwicklung übernommen. Die Proteinbilder wurden mit der Software ImageJ2x analysiert.
Cell Counting Kit (CCK)-8 Assay
Der CCK-8-Assay wurde mit den Kits (Beyotime, Shanghai, China) implementiert. Zellen (1 × 10 4 ) wurden auf eine 96-Well-Platte ausgesät und inkubiert. Für 0, 24, 48 und 72 h kultiviert, wurden die Zellen mit 10 μL/Well CCK-8-Lösung versetzt und 1 h lang ausgebrütet. Der Wert der optischen Dichte wurde mit einem Multiskan Spectrum-Mikroplattenlesegerät mit voller Wellenlänge bei 450 nm bestimmt. Sechs Vertiefungen wurden genommen, um den Durchschnittswert zu zählen. Die Zellwachstumskurve wurde als Ordinate mit der Zeit und als Ordinate die relative Lebensfähigkeit der Zellen aufgetragen. Der Wert der optischen Dichte repräsentiert die Zellproliferation.
Durchflusszytometrie
Zellen (1 × 10 6 ) wurden bei 1500 U/min zentrifugiert, mit 200 µL Bindungspuffer suspendiert, mit 5 µL Propidiumiodid (PI) und 5 µL Annexin V-Fluoresceinisothiocyanat (FITC) wiederum inkubiert und mit 400 µL Puffer angehängt. Die Zellapoptoserate wurde mit einem Durchflusszytometer (BD Biosciences, NJ, USA) überprüft.
Kratztest
Die transfizierten Zellen wurden abgelöst und zu einer Zellsuspension präpariert. Zellsuspensionen (1 × 10 6 Zellen/ml) wurden in einer 6-Well-Platte ausgesät und auf 80–90% Konfluenz kultiviert. Quer- und Längskratzer auf der 6-Well-Platte wurden gleichmäßig mit einer aseptischen 10-μl-Pipettenspitze entlang des Lineals gemacht. Das Originalmedium wurde durch Vollmedium ersetzt und die Zellen wurden kontinuierlich kultiviert. Die Zellwanderung wurde um 0 h und 72 h unter einem Mikroskop beobachtet und an derselben Stelle abgebildet.
Transwell-Assay
Die transfizierten Zellen wurden trypsiniert, bei 1000 g zentrifugiert und mit DMEM gespült. Eine vollständig hydratisierte Transwell-Kammer wurde in eine 24-Well-Platte gestellt, die 10 % FBS-DMEM (600 ml/Well) enthielt. Zellsuspension (5 × 10 5 Zellen/ml, 200 μl) wurde in die obere Transwell-Kammer (beschichtet mit 1:8 Matrigel 80 μl) gegeben, während 500 μl 20 % FBS-DMEM in die untere Kammer gegeben wurden. Die Zellen wurden 24 h kontinuierlich kultiviert, mit 500 μL 4% Paraformaldehyd (PFA) fixiert und mit 0,1% Kristallviolett-Färbelösung gefärbt. Dann wurden die Zellen auf der Oberfläche und am Rand der oberen Kammer mit einem Wattestäbchen abgewischt. Fünf Felder wurden zufällig ausgewählt und die Zellen wurden unter einem Nikon Eclipse TE2000-S Mikroskop (Nikon, Japan) gezählt.
Sphärenbildungsassay
Die Zellen wurden in eine 6-Well-Platte mit geringer Adsorption ausgesät, die serumfreies Suspensionsmedium mit 200 Zellen/Well enthielt. Nach 2 W wurde die Kugelbildungsrate der Zellen unter einem Nikon Eclipse TE2000-S Mikroskop (Nikon) beobachtet und die Kugelbildungsrate wurde als durchschnittliche Anzahl von Kugeln/Anzahl der ausgesäten Zellen berechnet × 100%.
Chromatin-Immunpräzipitation (ChIP)-Assay
Der ChIP-Assay wurde mit dem ChIP-Kit (Upstate, NY, USA) durchgeführt. SOX2 (1:1000, Restem Biotech, Jiangsu, China) und normale Kaninchen-IgG (12–370, Millipore, USA)-Antikörper wurden verwendet, um den vernetzten Protein-DNA-Komplex zu immunpräzipitieren. CD44 + HeLa-Zellen wurden mit 1% PFA fixiert und inkubiert, um eine DNA-Protein-Vernetzung zu erzeugen. Dann wurde die DNA durch Ultraschall in ein 200–300 bp großes Chromatinfragment geschnitten. Die ausgefällte Chromatin-DNA wurde gewonnen und durch RT-qPCR analysiert.
RNA-Pull-Down-Assay
Die biotinmarkierten miR-185-3p-Wildtyp-(WT)- und Mutanten-Typ-(MUT)-Plasmide (50 nM) wurden in CD44 + . transfiziert HeLa-Zellen bzw. Die Zellen wurden nach 48 h Transfektion mit spezifischem Zelllysat (Ambion, Austin, Texas, USA) ausgebrütet. Das Zelllysat (50 ml) wurde unterverpackt. Das restliche Lysat wurde mit M-280-Streptavidin-Kügelchen (Sigma, St. Louis, MO, USA), die mit RNase-freier und Hefe-tRNA (Sigma) vorbeschichtet waren, ausgebrütet. Anschließend wurden die Zellen zweimal mit kaltem Lysat, dreimal mit Niedrigsalzpuffer und einmal mit Hochsalzpuffer gereinigt. Als NC wurde eine antagonistische miR-185-3p-Sonde eingesetzt. Die Gesamt-RNA wurde mit Trizol extrahiert und der CCAT1-Spiegel wurde durch RT-qPCR getestet.
Dualer Luciferase-Reporter-Gen-Assay
Potenzielle Bindungsstellen E1 und E2 von SOX2 auf der CCAT1-Promotorregion wurden von https://jaspar.genereg.net/ vorhergesagt. Die CCAT1-Promotorsequenz, die SOX2 und die CCAT1-Bindungsstelle E1 enthält, wurde synthetisiert, und die Vektoren CCAT1 3'UTR WT (E1-WT) und CCAT1 3'UTR MUT (E1-MUT) wurden gebildet. Die Vektoren wurden in pmirGLO (Beyotime) kloniert. Danach wurde CCAT1-WT/pmirGLO oder CCAT1-MUT/pmirGLO mit sh-SOX2 oder sh-SOX2 NC zu CD44 cotransfiziert + HeLa-Zellen jeweils für 2 d und dann lysiert. Die Luciferase-Aktivität wurde durch ein Luciferase-Nachweissystem (Takara, Dalian, China) getestet.
Die Bioinformatik-Website wurde verwendet, um die Bindungsstellen von CCAT1 und miR-185-3p vorherzusagen und zu analysieren. Die Bindungsstellen von CCAT1 und miR-185-3p wurden durch einen dualen Luciferase-Reportergen-Assay verifiziert. CCAT1 3′UTR, das die miR-185-3p-Bindungsstelle enthält, wurde zusammengestellt. CCAT1 3′UTR WT und CCAT1 3′UTR MUT wurden konstruiert und mit Mimetika NC und miR-185-3p Mimetika zu CD44 + . cotransfiziert HeLa-Zellen für 2 d. Dann wurden die Zellen lysiert und die Luciferase-Aktivität wurde durch das Luciferase-Nachweissystem (Takara) getestet. Die gleiche Methode wurde angewendet, um die Targeting-Beziehung zwischen miR-185-3p und FOXP3 zu überprüfen.
Statistische Analyse
Alle Daten wurden mit der Software SPSS 21.0 (IBM Corp. Armonk, NY, USA) ausgewertet. Die Messdaten wurden als Mittelwert ± Standardabweichung angegeben. Der t-Test wurde für die Diskrepanz zwischen zwei Gruppen und die Einweg-Varianzanalyse (ANOVA) angewendet, gefolgt von Tukeys Mehrfachvergleichstest für die Diskrepanz zwischen den Gruppen. Die Klassifizierungsvariable wurde durch den exakten Test nach Fisher bewertet. Ein p Wert von weniger als 0,05 wurde als signifikant angesehen.
Ergebnisse
miR-185-3p-Expression nimmt ab, während die SOX2-, CCAT1- und FOXP3-Expression in CC-Geweben zunehmen und die SOX2- und CCAT1-Expression mit der Tumorgröße, Lymphknoten Metastasen (LNM) und FIGO-Stadium
Beim Nachweis der Rolle der SOX2/CCAT1/miR-185-3p/FOXP3-Achse auf die Proliferation und Selbsterneuerungsfähigkeit von CC-Stammzellen wurden miR-185-3p-, SOX2-, CCAT1- und FOXP3-Expressionen in CC-Geweben und angrenzenden normalen Geweben getestet durch RT-qPCR und Western-Blot-Assay. Es zeigte sich, dass (Abb. 1a–c) die Expression von miR-185-3p reduziert war, während die Expression von SOX2, CCAT1 und FOXP3 in CC-Geweben erhöht war.
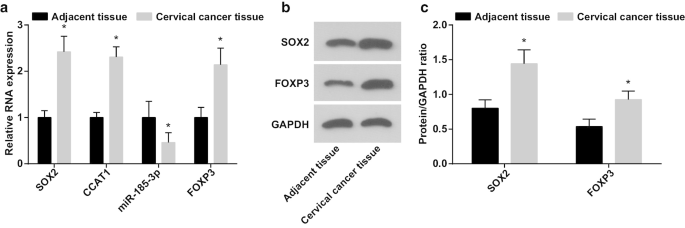
Die miR-185-3p-Expression nimmt ab, während die SOX2-, CCAT1- und FOXP3-Expression in CC-Geweben zunehmen. a Vergleich der SOX2-mRNA-, CCAT1-, miR-185-3p- und FOXP3-mRNA-Expression in CC und angrenzenden normalen Geweben. b Proteinbanden der SOX2- und FOXP3-Proteinexpression in CC und angrenzenden normalen Geweben. c Vergleich der SOX2- und FOXP3-Proteinexpression in CC und angrenzenden normalen Geweben. *p < 0,05 gegenüber angrenzendem Normalgewebe. Die Messdaten wurden als Mittelwert ± Standardabweichung dargestellt und Vergleiche zwischen zwei Gruppen wurden durch den t-Test bewertet
Die Beziehung zwischen SOX2/CCAT1-Expression und klinisch-pathologischen Merkmalen von CC wurde analysiert (Tabelle 2). Überexprimiertes SOX2 und CCAT1 in CC waren mit Tumorgröße, LNM und FIGO-Stadium verbunden, was darauf hindeutet, dass die SOX2- und CCAT1-Expression bei Patienten mit größerer Tumorgröße, LNM und fortgeschrittenem FIGO-Stadium von CC-Patienten höher war.
miR-185-3p-Ausdruck verringert sich, während SOX2-, CCAT1- und FOXP3-Ausdrücke bei CD44 zunehmen + HeLa-Zellen
Als nächstes wurden miR-185-3p, SOX2, CCAT1 und FOXP3 Expressionen in humanen zervikalen Epithel immortalisierten H8- und CC-Zelllinien SiHa, CasKi, HeLa, HCC94 und C33A getestet. Es wurde vermutet, dass (Abb. 2a–c) die miR-185-3p-Expression abgebaut war, während SOX2, CCAT1 und FOXP3 in CC-Zelllinien erhöht waren. Unter diesen wiesen SOX2/CCAT1/miR-185-3p/FOXP3 in HeLa-Zellen den größten Expressionsunterschied zu H8-Zellen auf; somit wurden HeLa-Zellen ausgesiebt und sortiert; HeLa-SFCs wurden durch Zellsuspensionskulturen erhalten, und die CD44-Expression in der Zelloberfläche vor und nach dem Sortieren wurde durch Durchflusszytometrie identifiziert. Die Ergebnisse betonten, dass die positive CD44-Rate in HeLa-SFCs deutlich höher war als vor der Sortierung, was auf eine erfolgreiche Sortierung von CC-Stammzellen schließen lässt (Abb. 2d). Die sortierten Stammzellen wurden als CD44 + . bezeichnet HeLa-Zellen. Dann wurden miR-185-3p-, SOX2-, CCAT1- und FOXP3-Expressionen in HeLa und CD44 getestet + HeLa-Zellen (Abb. 2e–g). Es zeigte sich, dass die Expression von SOX2, CCAT1 und FOXP3 hochreguliert und miR-185-3p herunterreguliert war bei CD44 + HeLa-Zellen.
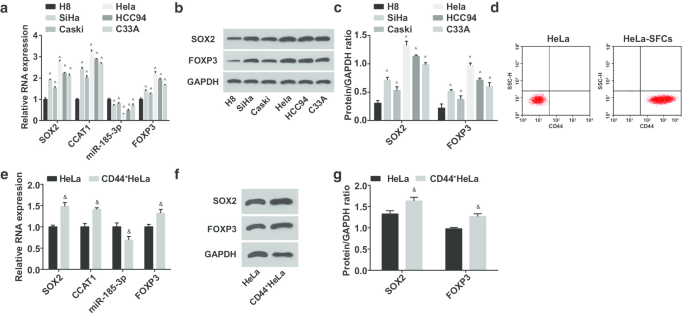
miR-185-3p-Expression reduziert sich, während SOX2-, CCAT1- und FOXP3-Expression bei CD44 ansteigt + HeLa-Zellen. a Vergleich der SOX2-mRNA-, CCAT1-, miR-185-3p- und FOXP3-mRNA-Expression in H8- und CC-Zelllinien. b Proteinbanden der SOX2- und FOXP3-Proteinexpression in H8- und CC-Zelllinien. c Vergleich der SOX2- und FOXP3-Proteinexpression in H8- und CC-Zelllinien. d Nachweis der CD44-Expressionsrate in HeLa-Zellen vor und nach der Sortierung durch Durchflusszytometrie. e Vergleich der SOX2-mRNA-, CCAT1- und miR-185-3p-Expression zwischen HeLa-Zellen und CD44 + HeLa-Zellen. f Proteinbande der SOX2-Proteinexpression in HeLa-Zellen und CD44 + HeLa-Zellen. g Vergleich der SOX2-Proteinexpression zwischen HeLa-Zellen und CD44 + HeLa-Zellen. ^p < 0,05 vs. H8-Zellen. & p <0,05 vs. HeLa-Zellen. Die Messdaten wurden als Mittelwert ± ± Standardabweichung dargestellt, Vergleiche zwischen zwei Gruppen wurden durch t-Test bewertet und Vergleiche zwischen mehreren Gruppen wurden durch Einweg-ANOVA bewertet, gefolgt von Tukeys Post-hoc-Test
Herunterregulieren von SOX2 und Herunterregulieren von CCAT1 verringern Proliferation, Migration, Invasion und Sphärenbildungsrate und erhöhen die Apoptose von CD44 + HeLa-Zellen
Anschließend wurden SOX2 und CCAT1 in CD44 + . zum Schweigen gebracht HeLa-Zellen, um ihre Auswirkungen auf die Proliferation und Selbsterneuerung von CC-Stammzellen zu untersuchen. Durch CCK-8-Assay, Durchflusszytometrie, Scratch-Test, Transwell-Assay und Sphärenbildungsexperiment nachgewiesen, wurden die Proliferation, Migration, Invasion und Sphärenbildungsrate unterdrückt und die Apoptose von CD44 + HeLa-Zellen wurden durch Hemmung von SOX2 und CCAT1 gefördert (Abb. 3a–i). Es war ein Hinweis darauf, dass das Abschalten von SOX2 oder CCAT1 die Proliferation und Selbsterneuerung von CC-Stammzellen hemmte.
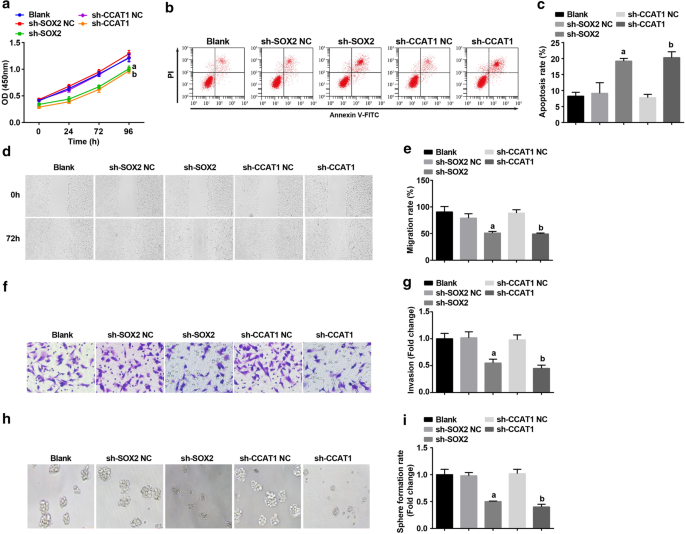
Das Herunterregulieren von SOX2 und das Herunterregulieren von CCAT1 verringern die Proliferation, Migration, Invasion und Sphärenbildungsrate und erhöhen die Apoptose von CD44 + HeLa-Zellen. a Der CCK-8-Assay testete die Zellwachstumskurve in Zellen, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden. b Die Durchflusszytometrie wies die Zellapoptose in Zellen nach, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden. c Vergleich der Zellapoptoserate in Zellen, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden. d Zellmigration in Zellen, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden, getestet durch Scratch-Test. e Vergleich der Zellmigration in Zellen, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden. f Nachweis der Invasionsfähigkeit von Zellen in Zellen, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden, durch Transwell-Assay. g Vergleich der Invasionsfähigkeit von Zellen, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden. h Das Kugelbildungsexperiment testete die Selbsterneuerungskapazität in Zellen, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden. ich Vergleich der Kugelbildungsrate in Zellen, die mit sh-CCAT1 oder sh-SOX2 behandelt wurden. a p < 0,05 gegenüber der sh-SOX2-NC-Gruppe. b p < 0,05 gegenüber der sh-CCAT1-NC-Gruppe. Die Messdaten wurden als Mittelwert ± ± Standardabweichung dargestellt, und Vergleiche zwischen mehreren Gruppen wurden durch eine einfache ANOVA, gefolgt von Tukeys Post-hoc-Test bewertet
MiR-185-3p aufgebraucht kehrt die Rolle der Herunterregulierung von CCAT1 in CD44 um + HeLa-Zellen
Dann untersuchten wir, ob miR-185-3p am Prozess von CCAT1 beteiligt ist, der die Proliferation und Selbsterneuerung von CC-Stammzellen reguliert. CD44 + HeLa wurde mit miR-185-3p-Mimic transfiziert oder mit sh-CCAT1 und miR-185-3p-Inhibitor co-transfiziert. Die Ergebnisse zeigten, dass die Hochregulierung von miR-185-3p die Proliferation, Migration, Invasion und Sphärenbildungsrate deutlich reduzierte, während die Apoptoserate von CD44 + . erhöht wurde HeLa-Zellen. Mit miR-185-3p-Inhibitor behandelte Zellen könnten die Rolle des herunterregulierten CCAT1 bei der Proliferation, Migration, Invasion, Apoptose und Zellsphärenbildung von CD44 umkehren + HeLa-Zellen (Abb. 4a–i).
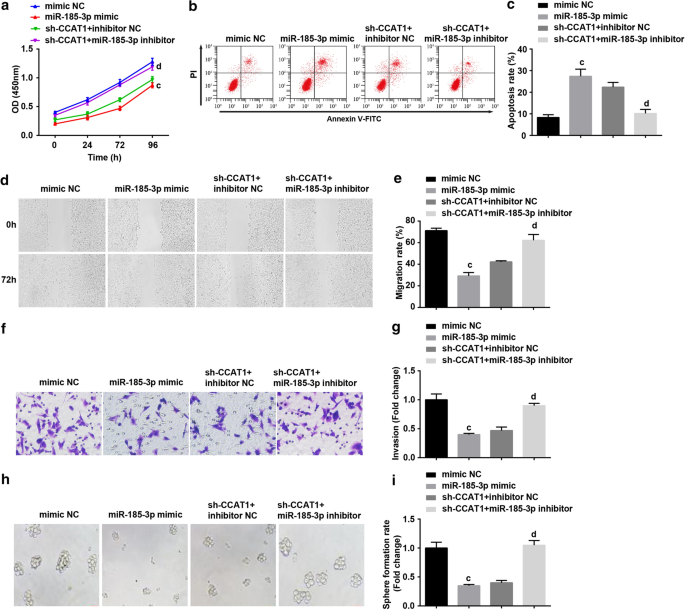
Überexprimiertes miR-185-3p unterdrückt Proliferation, Migration, Invasion und Sphärenbildungsrate und erhöht die Apoptose von CD44 + HeLa-Zellen. a Der CCK-8-Assay testete die Zellwachstumskurve in Zellen, die mit miR-185-3p-Mimic behandelt wurden. b Die Durchflusszytometrie wies die Zellapoptose in Zellen nach, die mit miR-185-3p-Mimic behandelt wurden. c Vergleich der Zellapoptoserate in Zellen, die mit miR-185-3p-Mimic behandelt wurden. d Zellmigration in Zellen, die mit miR-185-3p-Mimic behandelt wurden, getestet durch Scratch-Test. e Vergleich der Zellmigration in Zellen, die mit miR-185-3p-Mimic behandelt wurden. f Nachweis der Invasionsfähigkeit von Zellen, die mit miR-185-3p-Mimic behandelt wurden, durch Transwell-Assay. g Vergleich der Invasionsfähigkeit von Zellen, die mit miR-185-3p-Mimic behandelt wurden. h Das kugelbildende Experiment testete die Selbsterneuerungskapazität in Zellen, die mit miR-185-3p-Mimic behandelt wurden. ich Vergleich der Kugelbildungsrate in Zellen, die mit miR-185-3p-Mimic behandelt wurden. ein p < 0,05 gegenüber der sh-SOX2-NC-Gruppe. b p < 0,05 gegenüber der sh-CCAT1-NC-Gruppe. c p < 0,05 gegenüber der mimischen NC-Gruppe. d p < 0,05 gegenüber der sh-CCAT1 + -Inhibitor-NC-Gruppe. Die Messdaten wurden als Mittelwert ± ± Standardabweichung dargestellt, und Vergleiche zwischen mehreren Gruppen wurden durch eine einfache ANOVA, gefolgt von Tukeys Post-hoc-Test bewertet
Niedrig exprimiertes SOX2 und niedrig exprimiertes CCAT1 verringern die FOXP3-Expression und erhöhen die miR-185-3p-Expression in CD44 + HeLa-Zellen
Anschließend untersuchten wir die SOX2/CCAT1/miR-185-3p/FOXP3-Expression in CD44 + HeLa-Zellen nach Transfektion von sh-SOX2, sh-CCAT1, miR-185-3p-Mimik und Co-Transfektion von sh-CCAT1 und miR-185-3p-Inhibitor. Die Expression von SOX2, CCAT1 und FOXP3 war reduziert, während die Expression von miR-185-3p in Zellen, die mit sh-SOX2 behandelt wurden, erhöht war. Die Expressionen von CCAT1 und FOXP3 wurden reduziert und die Expression von miR-185-3p wurde in Zellen verstärkt, die mit sh-CCAT1 behandelt wurden. Die miR-185-3p-Expression war erhöht und die FOXP3-Expression war in Zellen verringert, die mit miR-185-3p-Mimic eingeführt wurden. In Zellen, die nacheinander mit sh-CCAT1 und miR-185-3p-Inhibitor transfiziert wurden, war die FOXP3-Expression erhöht und die miR-185-3p-Expression reduziert (Abb. 5a–d).
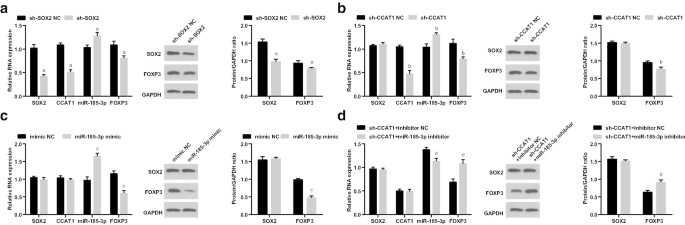
Niedrig exprimiertes SOX2 und schwach exprimiertes CCAT1 verringern die FOXP3-Expression und erhöhen die miR-185-3p-Expression bei CD44 + HeLa-Zellen. a SOX2/CCAT1/miR-185-3p/FOXP3-Expression in den Gruppen sh-SOX2 NC und sh-SOX2. b SOX2/CCAT1/miR-185-3p/FOXP3-Expression in den Gruppen sh-CCAT1 NC und sh-CCAT1. c SOX2/CCAT1/miR-185-3p/FOXP3-Expression in den mimischen NC- und miR-185-3p-mimetischen Gruppen. d SOX2/CCAT1/miR-185-3p/FOXP3-Expression in den sh-CCAT1 + -Inhibitor-NC- und sh-CCAT1 + miR-185-3p-Inhibitor-Gruppen. ein p < 0,05 gegenüber der sh-SOX2-NC-Gruppe. b p < 0,05 gegenüber der sh-CCAT1-NC-Gruppe. c p < 0,05 gegenüber der mimischen NC-Gruppe. d p < 0,05 gegenüber der sh-CCAT1 + -Inhibitor-NC-Gruppe. Die Messdaten wurden als Mittelwert ± ± Standardabweichung dargestellt, und Vergleiche zwischen mehreren Gruppen wurden durch eine einfache ANOVA, gefolgt von Tukeys Post-hoc-Test bewertet
SOX2 bindet an CCAT1, was die Expression von miR-185-3p beeinflusst, und FOXP3 ist ein Zielgen von miR-185-3p
Die potenziellen Bindungsstellen des Transkriptionsfaktors der CCAT1-Promotorregion wurden von der Website https://jaspar.genereg.net/ vorhergesagt und analysiert und es zeigte sich, dass SOX2 und CCAT1 potenzielle Bindungsstellen in der CCAT1-Promotorregion aufwiesen (Abb. 6a). ChIP-qPCR berichtete, dass (Abb. 6b):Im Gegensatz zur IgG-Gruppe waren mehr CCAT1-Promotorfragmente in der SOX2-Gruppe an der E1-Bindungsstelle angereichert, was bewies, dass SOX2 an den CCAT1-Promotor an der E1-Stelle gebunden war und SOX2 an der Regulierung von CCAT1 beteiligt. Ein dualer Luciferase-Reportergen-Assay zeigte, dass (Abb. 6c):Die Luciferase-Aktivität wurde in Zellen, die mit sh-SOX2 und E1-WT cotransfiziert waren, unterdrückt, was darauf hindeutet, dass SOX2 an CCAT1 binden könnte.
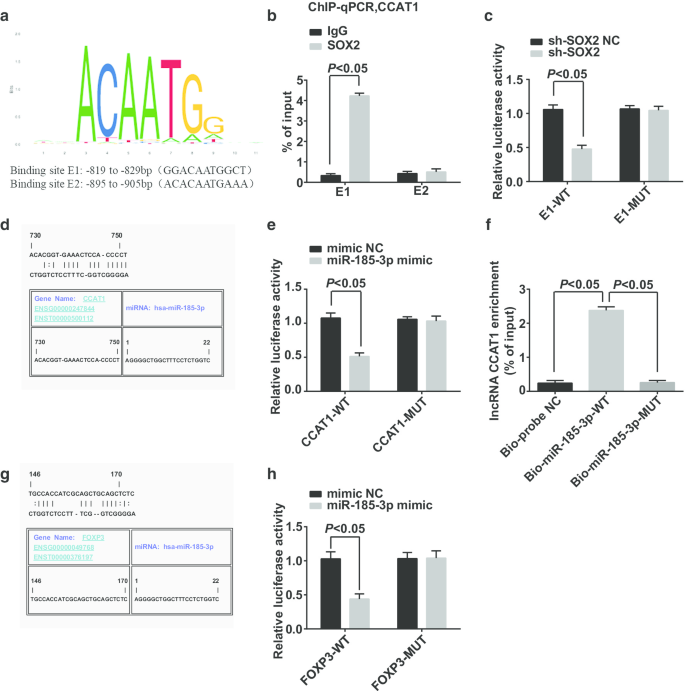
SOX2 kann an CCAT1 binden, was die Expression von miR-185-3p beeinflusst, und FOXP3 ist ein Zielgen von miR-185-3p. a Vorhersage von Bindungsstellen in SOX2- und CCAT1-Promotorregionen durch Bioinformatik-Sites. b Das ChIP-qPCR-Experiment bestätigte die Bindungsbeziehung zwischen SOX2 und CCAT1. c Die Bindungsstelle von SOX2 und CCAT1 wurde durch einen dualen Luciferase-Reportergen-Assay bestätigt. d Vorhersage von Bindungsstellen in CCAT1 und miR-185-3p durch Bioinformatik-Sites. e Überprüfung der Bindung von CCAT1 und miR-185-3p durch dualen Luciferase-Reportergen-Assay. f Die Bindungsbeziehung zwischen CCAT1 und miR-185-3p in Zellen, verifiziert durch RNA-Pulldown-Assay. g Vorhersage der Targeting-Beziehung zwischen miR-185-3p und FOXP3 durch die Bioinformatik-Website. h Identifizierung der Targeting-Beziehung zwischen miR-185-3p und FOXP3 durch dualen Luciferase-Reportergen-Assay. Die Messdaten wurden als Mittelwert ± Standardabweichung dargestellt und Vergleiche zwischen zwei Gruppen wurden durch den t-Test bewertet
Die Jefferson-Website sagte voraus, dass CCAT1 an miR-185-3p binden könnte (Abb. 6d). Ein dualer Luciferase-Reportergen-Assay berichtete, dass (Abb. 6e) die Luciferase-Aktivität in Zellen, die mit miR-185-3p-Mimic und CCAT1-WT eingeführt wurden, verringert war, was darauf hindeutet, dass miR-185-3p an CCAT1 binden könnte. Ein RNA-Pull-Down-Assay wurde verwendet, um zu überprüfen, ob CCAT1 an miR-185-3p binden kann. Die Ergebnisse zeigten, dass (Abb. 6f) der Anreicherungsgrad von CCAT1 in mit Bio-miR-185-3p-WT behandelten Zellen deutlich anstieg, während der Anreicherungsgrad von CCAT1 in mit Bio-miR-185-3p-MUT behandelten Zellen zeigte kein signifikanter Unterschied. Dieses Ergebnis zeigte, dass CCAT1 miR-185-3p adsorbieren kann, wodurch die Expression von miR-185-3p beeinflusst wird.
Die Zielbeziehung zwischen miR-185-3p und FOXP3 wurde von der Jefferson-Website vorhergesagt (Abb. 6g). Dualer Luciferase-Reportergen-Assay bestätigte, dass (Abb. 6h) die relative Luciferase-Aktivität von Zellen drastisch reduziert wurde, nachdem FOXP3-WT und miR-185-3p-Mimetik auf CD44 cotransfiziert wurden + HeLa-Zellen, während FOXP3-MUT mit miR-185-3p-Mimic co-transfiziert war, beeinflusste die relative Luciferase-Aktivität der Zellen nicht, was darauf hindeutet, dass miR-185-3p auf FOXP3 abzielte.
Diskussion
CC ist die vierthäufigste bösartige Erkrankung bei Frauen weltweit, gefolgt von Brust-, Dickdarm- und Lungenkrebs [3]. Es wurde berichtet, dass CC-Zellen mit positiver SOX2-Expression die Eigenschaften von Krebsstammzellen aufweisen [21]. Eine Studie hat berichtet, dass CCAT1 eine essentielle onkogene lncRNA ist, die mit CC verwandt ist und eine unterstützende Rolle beim Wachstum und der Invasion von CC-Zellen spielt [11]. Eine andere Studie zeigt, dass miR-185-3p die Strahlenempfindlichkeit von Nasopharynxkarzinomen vorhersagen und das Wachstum und die Apoptose von Krebszellen regulieren könnte [22]. Es wurde berichtet, dass der zirkulierende Autoantikörper gegen FOXP3 das kontinuierliche Fortschreiten der zervikalen Läsion widerspiegelt und ein potenzieller Biomarker für die Frühprognose von CC sein könnte [23]. Die aktuelle Studie wurde entwickelt, um zu untersuchen, wie die SOX2/CCAT1/miR-185-3p/FOXP3-Achse die Proliferation und die Selbsterneuerungsfähigkeit von CC-Stammzellen beeinflusst.
Basierend auf unseren Ergebnissen waren die SOX2- und CCAT1-Expressionen in CC-Geweben und -Zellen erhöht, die mit Tumorgröße, LNM und fortgeschrittenem FIGO in Verbindung standen. Functionally proved, down-regulating SOX2 and CCAT1 declined proliferation, migration, invasion and sphere cells number and increased apoptosis of CC stem cells. Similar to our study, SOX2 expression trends to increase in CC [24, 25]. Moreover, SOX2 expression is also up-regulated in CC cells derived from cancer stem cells [26]. Overexpressed SOX2 was suggested to link with clinicopathological characteristics of patients with several types of cancer, not limited to CC. For example, it was suggested that up-regulated SOX2 shows in cervical squamous cell carcinoma patients staged in FIGO I-II [27]. Moreover, SOX2 expression is linked to LNM in oral squamous cell carcinoma [28]. When it comes to the molecular function of SOX2 for cancer progression, there is an observational work presenting that down-regulated SOX-2 suppresses cell migration and invasion of cervical squamous cell carcinoma [29]. Meanwhile, another research has offered a proof that up-regulated SOX2 enhances CC cell clonogenicity, proliferation and tumorigenicity in vitro and in vivo than control cells [30].
Concerning to the regulatory relation between SOX2 and CCAT1, an existed study has presented that silencing SOX2 markedly reduces CCAT1 mRNA level [31]. As to the role of CCAT1 in cancers, a study has showed that CCAT1 expression is markedly elevated in CC tissues versus in the adjacent normal tissues [11, 12]. Of note, CCAT1 overexpression in CC is positively related to the tumor size [12]. In terms of the role of CCAT1 in cancer cell activity, there is a research highlighting that overexpressed CCAT1 accelerates CC cell proliferation, colony formation and invasion [11]. Interestingly, a previous research has demonstrated that the cell viability, invasive and migratory abilities are declined via knocking down CCAT1 [12]. Anyway, the functional effect of SOX2 and CCAT1 in other cancers was similar to that in CC.
Afterward, our research revealed that CCAT1 could bind to miR-185-3p, the down-regulated CCAT1 in CC and overexpressing miR-185-3p suppressed the proliferation and self-renewal abilities of CC stem cells. It is reported that CCAT1 and miR-185-3p are negatively correlated [13]. Furthermore, a result reported that a reduction is seen in miR-185-3p expression in radioresistant nasopharyngeal carcinoma cases [22]. Regarding to the suppressive function of miR-185-3p in cancer cell aggressiveness, a study has revealed that up-regulation of miR-185-3p suppresses the invasive and metastatic properties of nasopharyngeal carcinoma cells [32]. Furthermore, Zou et al. have suggested that restored miR-185 represses breast cancer cell growth and invasion [33]. There is a article finding that up-regulation of miR-185 declines the proliferation, invasion and colony formation capacities of non-small cell lung cancer cells in vitro [34]. It is presented that in vitro cell proliferation, invasion and migration as well as in vivo tumor growth are suppressed via miR-185-overexpressing in non-small cell lung cancer cells [35]. From those studies, the anti-tumor role of miR-185-3p in the present study was consistent with previous researches.
To proceed, we unveiled that miR-185-3p targeted FOXP3, the overexpressed gene in CC to regulate CC stem cell activities. In fact, FOXP3, the regulator of SOX2 cancer stem-like cell marker in colon cancer [36], has been investigated in CC, showing an up-regulation in CC cells [19] [37]. It was evidenced that elevating FOXP3 promotes the formation of tumor spheres and stimulates the stemness of non-small cell lung cancer cells [38].
Conclusion
Collectively, we explored for the first time that SOX2 transcription could activate CCAT1, thereby inhibiting miR-185-3p and regulating FOXP3 to promote the proliferation and self-renewal of CC stem cells, which is a potential avenue to treat CC. Additionally, however, limitations in this present study still exist in the relatively small trial size in the designed experiment. Thus, clinical researches might be further carried out to detect the efficacy for the treatment of CC.
Verfügbarkeit von Daten und Materialien
Nicht zutreffend.
Nanomaterialien
- Demonstration eines flexiblen Graphen-basierten Biosensors für den empfindlichen und schnellen Nachweis von Eierstockkrebszellen
- Nanopartikel für die Krebstherapie:Aktuelle Fortschritte und Herausforderungen
- Zellbasierte Arzneimittelabgabe für Krebsanwendungen
- Nanotechniken inaktivieren Krebsstammzellen
- Auswirkung der Verteilung von Goldnanopartikeln in TiO2 auf die optischen und elektrischen Eigenschaften von farbstoffsensibilisierten Solarzellen
- Graphenoxid-hybridisierte nHAC/PLGA-Gerüste erleichtern die Proliferation von MC3T3-E1-Zellen
- TEM- und STEM-Studien zu den Querschnittsmorphologien von Dual-/Tri-Layer-Breitband-SiO2-Antireflexfolien
- Herstellung und Charakterisierung von ZnO-Nano-Clips durch das Polyol-vermittelte Verfahren
- Frequenzmodulierte Wellendielektrophorese von Vesikel und Zellen:Periodische Kehrtwendungen bei der Übergangsfrequenz
- Faktoren, die den Erfolg und die Sicherheit des Stanzprozesses beeinflussen



