Studien zur Etablierung und Anwendung eines präzisen Lysosom-pH-Indikators basierend auf selbstzersetzbaren Nanopartikeln
Zusammenfassung
Der saure pH-Wert von Lysosomen steht in engem Zusammenhang mit der Autophagie; Daher geben pH-Änderungen, die von den genauen Lysosomen bekannt sind, mehr Informationen über den Autophagieprozess und -status. Bisher konnten jedoch nur pH-Änderungen in einem relativ breiten Bereich angezeigt werden, der genaue pH-Nachweis der Lysosomen ist nie angekommen. In unserer Studie haben wir einen Endo-/Lysosomen-pH-Indikator basierend auf dem selbstzersetzbaren SiO2 . etabliert Nanopartikelsystem mit spezifischen Syntheseparametern. Das zentrale konzentrierte Methylenblau (MB) in den zentral-hohlen strukturellen Nanopartikeln zeigte eine empfindliche Freisetzung in Abhängigkeit von pH-Werten von pH 4.0–4.8, was genau dem pH-Bereich von Lysosomen entspricht. Die lineare Korrelation der optischen Dichte (OD)-Werte und der pH-Werte wurde aufgebaut, die zum Nachweis des pH-Wertes von Lysosomen in 6 verschiedenen Zelllinien verwendet wurde. Darüber hinaus ist es uns mit diesem System gelungen, die durchschnittlichen pH-Änderungen von Lysosomen vor und nach der NP-Endocytose des schwarzen mesoporösen Siliziums (BPSi) präzise zu detektieren, wodurch der Mechanismus der Autophagie-Termination nach der BPSi-Endocytose aufgeklärt wurde. Der selbstzersetzbare luminale pH-Indikator auf Nanopartikelbasis kann also eine neue Methodik und Strategie bieten, um den pH-Wert von Lysosomen besser zu kennen und dann mehr Details zum Autophagieprozess oder anderen wichtigen Signalen über den Stoffwechsel anzuzeigen.
Einführung
Lysosomen dienen als Endziel für Makromoleküle, wo diese Makromoleküle durch hydrolytische Enzyme abgebaut werden, die durch einen niedrigen pH-Wert aktiviert werden [1]. Der saure pH-Wert der Lysosomen, der durch die vakuoläre H + −ATPase (v-ATPase) [2] aufrechterhalten wird, die Protonen aus dem Zytoplasma in das lysosomale Lumen pumpt, sollte die Aktivität von ~ 60 Arten hydrolytischer Enzyme aufrechterhalten [3]. Darüber hinaus haben neuere Literaturberichte gezeigt, dass der saure pH-Wert von Lysosomen eng mit der Autophagie zusammenhängt [4], so dass die genauen pH-Änderungen der Lysosomen mehr Informationen über den Autophagieprozess und -status geben werden. Basierend auf unseren Studien und Literaturrecherchen wird die Endozytose mit Amin-positiv geladenen Nanopartikeln wahrscheinlich die pH-Änderung in Endo/Lysosomen erhöhen, wie z /P>
Der durch die Endozytose von Amin-Nanopartikeln induzierte Anstieg des pH-Werts wird die nukleäre Lokalisation des Transkriptionsfaktors EB (TFEB) dramatisch erhöhen [7], führt nicht nur zu einer transkriptionellen Hochregulierung des Signalwegs, sondern verursacht auch eine lysosomale Dysfunktion, die letztendlich zu einer Blockierung des autophagischen Flusses führt [ 7,8,9]. Da TFEB die Autophagie reguliert und folglich seine Überexpression zu einer signifikanten Zunahme der Autophagosomenproduktion in kultivierten Zellen führt.
Um den Autophagieprozess und die Details der Autophagie vorherzusagen, sind daher der genaue pH-Wert des Lysosoms und seine Änderungsmessung sehr wichtig. Bis jetzt konnten von den Endo/Lysosomen-pH-Wert anzeigenden Literaturübersichten [10] und den kommerziellen Produkten zum Nachweis der Endo/Lysosomen-pH-Werte nur pH-Änderungen in einem relativ breiten Bereich angezeigt werden, und die genaue Lysosomen-pH-Detektion ist nie angekommen . Um die Einblicke in die Autophagie zu kennen, ist die Etablierung einer präzisen Methode zum Nachweis der luminalen pH-Änderung ein wichtiger Ansatz.
Basierend auf unseren bisherigen Erfahrungen mit selbstzersetzbarem SiO2 Nanopartikel haben wir in dieser Studie einen präzisen pH-Indikator entwickelt, der die Erkennung von luminalen pH-Änderungen ermöglichen könnte. SiO2 Nanopartikel haben gute Vorteile in der einstellbaren Größe und Biokompatibilität [11]. Durch die Einstellung spezifischer Syntheseparameter wird das etablierte selbstzersetzbare SiO2 Der pH-Indikator könnte die Nutzlast Methylenblau (MB) bei pH 4.0–4.8 empfindlich freisetzen, was genau dem pH-Bereich von Lysosomen entspricht. Darüber hinaus zeigte die MB-Freisetzung eine lineare Korrelation mit den pH-Wert-Änderungen (Schema 1). Dann testeten wir die Machbarkeit des pH-Indikators auf Zellebene, indem wir 6 verschiedene Zelllinien einführten, es gelang uns, die durchschnittlichen pH-Änderungen von Lysosomen vor und nach der Endozytose von schwarzen mesoporösen Silizium (BPSi) NPs zu bestimmen und den Mechanismus der Autophagietermination nach BPSi . zu klären Endozytose. Der selbstzersetzbare luminale pH-Indikator auf Nanopartikelbasis kann also eine neue Methodik und Strategie bieten, um den pH-Wert von Lysosomen besser zu kennen und dann mehr Details zum Autophagieprozess oder anderen wichtigen Signalen über den Stoffwechsel anzuzeigen.
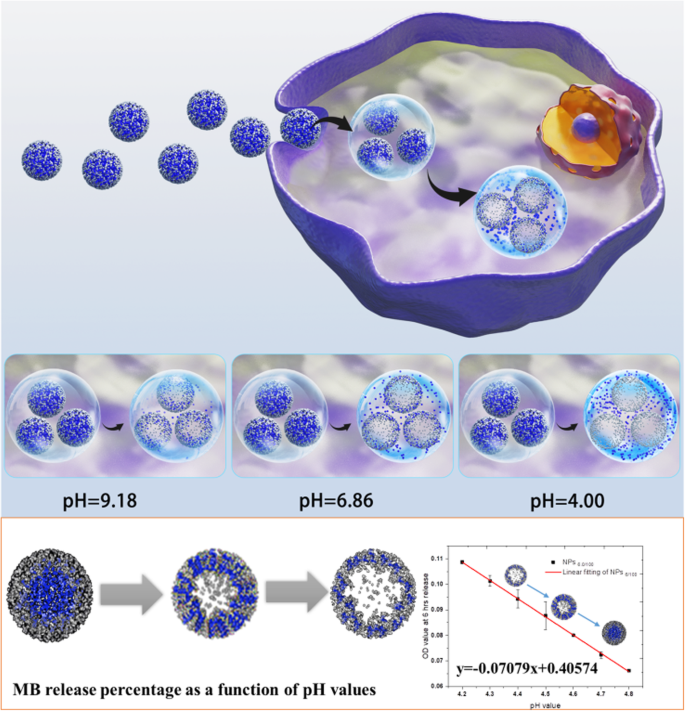
Schematische Darstellung von MB@SiO2 Messung des pH-Wertes von Lysosomen in lebenden Zellen
Materialien und Methoden
Materialabschnitt
Natriumsilicid (NaSi) und Si-Wafer (Durchmesser 20 cm, p + (100), 0,01–0,02 Ω cm) wurden von SiGNa Chemistry Inc. bzw. Ocmetic Inc. bereitgestellt. Ammoniumbromid (NH4 .) Br, 99%), Natriumbromid (NaBr, 99%), Toluol (wasserfrei, 99,8%), Salzsäure (HCl, 37%), MB und Tetraethylorthosilikat (TEOS) wurden von Sigma-Aldrich gekauft. 0,5 kDa Methoxy-PEG-Silan und 2 kDa Methoxy-PEG-Silan wurden getrennt von Fluorochem Ltd. und Laysan Bio Inc. gekauft. Das Medium RPMI 1640 wurde von Life Technologies bereitgestellt. Fötales Rinderserum (FBS) wurde von der biologischen Technologie von TianHang gekauft. Natriumbicarbonat, Streptomycinsulfat, Penicillin G, HEPES, Lysozym-Lösung, CellLight Early-Endosomen-GFP, LysoTracker™ Red DND-99, Pierce® BCA Protein Assay Kit, verstärkte Chemilumineszenz, pHrodo™ Red Transferrin-Konjugat, Live Cell Imaging Solution und Trizol-Reagenz wurde von Thermo Fisher Scientific gekauft. Ethanol und Ammoniak-Wasser wurden von Sinopharm bereitgestellt. BioRT Master HiSensi cDNA First-Strand-Synthese-Kit wurde von Hangzhou Bioer Technology Co., Ltd. gekauft. RIPA-Lysat wurde von Heart Biological Technology Co., Ltd. gekauft. P62, TFEB und β-Aktin-Antikörper wurden von der Proteintech Group gekauft , Inc. LC 3B-Antikörper wurde von Abcam bereitgestellt. 2-(4-Pyridyl)-5-((4-(2-dimethylaminoethyl-laminocarbamoyl)methoxy)phenyl)oxazol (PDMPO) wurde von Yeasen Biotech Co., Ltd. bereitgestellt.
Ziel, Design und Setting der Studie
Das Ziel dieser Studie ist es, (1) den Einfluss von BPSi-Nanopartikeln auf die Autophagie in HepG2-Zellen zu untersuchen, (2) den zugrunde liegenden Mechanismus der pH-Wert-Änderung von Lysosomen, die die Autophagie beeinflussen, herauszufinden, (3) einen präzisen pH-Indikator für Lysosomen zu etablieren, der messen könnte den pH-Wert von Lysosomen genau und schließlich (4) zeigen den Einfluss der pH-Schwankung auf die Autophagie. Um den oben genannten Studienzweck zu verwirklichen, verwendeten wir das Transkriptom-Sequenzierungsexperiment, um die Transkriptom-Genveränderungen in HepG2-Zellen nach der Fütterung von BPSi-Nanopartikeln zu untersuchen und durch RT-qPCR und Western-Experimente zu verifizieren. Fluoreszenzfarbstoffe wie PDMPO wurden verwendet, um die Änderung des lysosomalen pHs in HepG2-Zellen nach der Fütterung von BPSi zu messen. Um den pH-Wert von Lysosomen präzise zu messen, haben wir MB@SiO2 . entwickelt Nanopartikel mit 10 Parametern und testete die Charakterisierungen dieser 10 Arten von Nanopartikeln durch Experimente wie DLS und HR-TEM. Die MB-Beladungseffizienz- und Freisetzungskinetik-Studie von selbstzersetzenden Nanopartikelsystemen der Serie 10 wurde in verschiedenen pH-Lösungen und HepG2-Zellen getestet. Um die intrazelluläre Position der Nanopartikel nach dem Eintritt in die Zelle zu überprüfen, führten wir ein Zell-TEM-Experiment und eine konfokale Mikroskopie mit lebenden Zellen durch. Schließlich haben wir die lysosomalen pH-Änderungen in 6 Arten von Zellen nach der Fütterung von BPSi gemessen, um die Universalität von MB@SiO2 . zu überprüfen Nanopartikel zur Messung der lysosomalen pH-Änderungen.
BPSi-Nanopartikelsynthese
Die BPSi-Nanopartikel wurden nach unserer vorherigen Methode [12] hergestellt und von unserem Mitarbeiter (Wujun Xu, Department of Applied Physics, University of Eastern Finland) bereitgestellt. Die Herstellung von BPSi, NaSi, Ammoniumsalz und NaBr (NaSi:NH4 Br:NaBr von 1:4:4, w/w/w) wurde in einer Handschuhbox mit einer Ar-Atmosphäre gemahlen. Sie wurden in einem Röhrenofen unter einem N2 . reagieren gelassen Atmosphäre bei 240 °C für 5 h (Gl. 1). Nach dem Abkühlen auf Umgebungstemperatur wurden die erhaltenen Mikropartikel durch separates Spülen mit 0,5 µM HCl- und 1,0 µM HF-Lösungen gereinigt. Die Mikropartikel wurden in Ethanol bei 1000 U/min für 15 Minuten kugelgemahlen, und die BPSi-Nanopartikel mit dem gewünschten Durchmesser wurden durch Einstellen der Zentrifugationsgeschwindigkeit gesammelt.
$$ \mathrm{NaSi}+{\mathrm{NH}}_4\mathrm{Br}\to \mathrm{NaBr}+{\mathrm{NH}}_3+\mathrm{Si}/\mathrm{H}+{ \mathrm{H}}_2 $$ (1)Durch das Experiment zur dynamischen Lichtstreuung wurden die Durchmesserverteilung und die Oberflächenladung des Nanopartikels untersucht. Alle NPs wurden nach der Sterilisation durch leichte Ultraschallbehandlung (5 Sekunden, um sie gleichmäßig in der Lösung zu verteilen, Ultraschallreiniger SB-5200DT, Ningbo Scientz Biotechnology Co., Ltd.) im Medium dispergiert, kurz bevor sie in die Zellen eingebracht wurden.
Einrichtung eines selbstzersetzenden Nanopartikelsystems der 10er-Reihe
Die selbstzersetzenden Nanopartikel der 10er-Reihe wurden mit den zuvor beschriebenen Methoden [13,14,15,16] mit modifizierten Parametern synthetisiert. In einem typischen Verfahren wurde zunächst eine bestimmte Menge MB zu einer Mischung aus Ethanol (75 ml) mit einer Ammoniak-Wasser-Lösung (25 %, 3.4 ml) gegeben, dann wurde eine bestimmte Menge TEOS zugegeben. Die Serie selbstzersetzlicher MB@SiO2 Die NPs wurden nach 24-stündigem Rühren erhalten und dreimal gewaschen, bevor sie getrocknet wurden. Die in den Protokollen hinzugefügten MB- und TEOS-Mengen waren wie in Tabelle 1 beschrieben. Die Bedeutung von 1,0/100 in den NPs1,0/100 stellten den Bestand an MB und TEOS dar, als wir die Nanopartikel synthetisierten, mit 1,0 mg MB und 100 &mgr;l TEOS. Und die Bedeutung von 1,5/100 in den NPs1,5/100 und andere stimmen mit 1.0/100 überein.
Zellkultur
Um die Effizienz und Universalität des pH-Indikators auf Basis selbstzersetzbarer Nanopartikel zu testen, haben wir versucht, ihn an bestimmten Tumorzelllinien zu testen. Als Forschungsobjekte haben wir daher Leberkrebs, Lungenkrebs, Dickdarmkrebs und Melanozytom-Zelllinien ausgewählt. Die Zelllinien menschlicher Dickdarmkrebszellen HCT116, HCT8 und HCT15; menschliche Leberkrebszellen HepG-2; menschliche Lungenkrebszellen A549; und Maus-Melanomzellen B16 wurden in RPMI 1640-Medium (Life Technologies), ergänzt mit 10 % hitzeinaktiviertem FBS, 2,0 µg/l Natriumbicarbonat, 0,1 µg/l Streptomycinsulfat, 0,06 µg/l Penicillin G und 5,958 µg/l&supmin; gehalten HEPES. Die Zellen wurden in einem Standard-Zellkulturinkubator bei 37 °C in einer befeuchteten Atmosphäre mit 5 % CO2 . gehalten .
Charakterisierungen von selbstzersetzenden Nanopartikelsystemen der 10er-Reihe
Die Morphologie aller Nanopartikel der Serie wurde durch HR-TEM mit STEM-Modus charakterisiert, und die Si-Abbildung wurde durch EDS-Element-Mapping untersucht. Die Analyse der Nanopartikelgrößenverteilung wurde mit der ImageJ-Software durchgeführt, indem die Nanopartikeldurchmesser in zufällig ausgewählten STEM-Bildern berechnet wurden. Das Zetapotential der Nanopartikel und der Polydispersitätsindex (PDI) wurden durch die dynamische Lichtstreuung (DLS) in Reihenpuffern mit spezifischen pH-Werten gemessen. Die Daten wurden mit SPSS15.0 analysiert und die statistischen Ergebnisse wurden als Mittelwert ± S.D.
. dargestelltDie MB-Studie zur Beladungseffizienz und Freisetzungskinetik von selbstzersetzenden Nanopartikelsystemen der Serie 10
Um die MB-Beladungseffizienz und Freisetzungskinetik zu untersuchen, wurde zuerst die Standardkurve von MB in Serienkonzentrationen erstellt. Die Absorption von MB wurde durch ein UV-Vis-Spektrum mit einer Absorption bei 660 nm, dem λ ., durchgeführt max des Monomers MB. Die MB-Ladeeffizienz wurde durch die folgende Gleichung berechnet, MB-Ladeeffizienz (%) = die Menge an verkapseltem MB/(Gesamtmenge der MB-Eingabe).
Die Freisetzung von MB aus Nanopartikeln der 10er-Reihe wurde in reinem Wasser und pH-Puffer mit unterschiedlichen pH-Werten (pH 4,0, pH 6,86 und pH 9,18) und Lysozym-Lösung (Thermo Scientific™ # .) untersucht 90082). Darüber hinaus wurde auch die MB-Freisetzungskinetik nach einer bestimmten Dauer in verschiedenen pH-Puffern untersucht. Die OD-Werte bei 660 nm und die MB-Freisetzungsprozentsätze als Funktion der Zeit wurden dann untersucht.
Genauer gesagt wurden die MB-Freisetzungsstudien nach den folgenden Protokollen durchgeführt; gelöste Nanopartikel der 10er-Reihe in 15 ml Standardpuffer von pH 4,0, 6,86 bzw. 9,18 mit Lysosomenlösung; und führte die MB-Freisetzung in einem Hula-Mischer bei 37 °C durch. Während der folgenden 15 Tage wurde 1 ml jeder Probe gesammelt und dann 10 Minuten bei 12000 U/min zentrifugiert. Der Überstand und der Niederschlag wurden auf ihre Absorptionsspektren bei 200–800 nm gemessen.
Darüber hinaus wurde die MB-Freisetzung in präzisen pH-Puffern mit Lysozymlösung im pH-Bereich von 4,1 bis 5,5 ebenfalls mit den gleichen Protokollen oben untersucht. Als Beobachtungszeitpunkte wurden bestimmte Zeitdauern (6 h, 12 h und 24 h) gesetzt. Die Absorptionen bei 660 nm wurden in jeder Probe aufgezeichnet. Die lineare Beziehung der Absorption jeder pH-Lösung und die Summe der Quadrate der Reste wurden jeweils zu jedem Zeitpunkt gezählt.
Um die MB-Freisetzungsprofile in Zellen nachzuweisen, wurden HepG-2-Zellen in einem 75 cm 2 . kultiviert Kulturflasche und mit NPs (300 µg/ml) gefüttert, wenn die Zellen auf 70 % der Kulturflasche proliferiert waren. Alle 30 min später wurden die Zellen gesammelt. Die Zellen wurden wiederholt eingefroren und aufgetaut, um das MB vollständig in den Zellen freizusetzen. Die Zelllysate wurden 10 min bei 12000 U/min zentrifugiert. Der Überstand wurde erhalten und seine Absorption bei 660 nm gemessen, um die Gesamtmenge an freigesetztem MB zu berechnen. In dieser Studie wurden HepG2-Zellen aufgrund ihrer schnellen Zellproliferation als Forschungsobjekte ausgewählt, die die Varianz zwischen allen 10 getesteten Gruppen minimieren konnte.
Zelluläre Kolokalisation der Nanopartikel der 10er-Reihe und die Freisetzungsleistung in 6 verschiedenen Zelllinien
Zell-TEM wurde verwendet, um die Kolokalisation der Nanopartikel in den Endo/Lysosomen nach Standard-Zell-TEM-Protokollen zu untersuchen. Die Zellen wurden in einer Intensität von 1 × 10 6 . ausgesät Zellen/Kolben und inkubiert für 24 Stunden, um die Zellanhaftung zu ermöglichen. Zehn Serien von Nanopartikeln in Medium mit der gleichen Konzentration (100 µg/ml) wurden mit den Zellen für weitere 12 bzw. 24 Stunden inkubiert. Die Zellen wurden dann dreimal mit PBS gewaschen, um die überschüssigen Nanopartikel zu entfernen, dann in 2,5% Glutaraldehydlösung länger als 1 Tag fixiert. Die fixierten Zellen wurden dann gewaschen und mit Osmiumtetroxid, 1% in entionisiertem Wasser für 1 Stunde gefärbt, gefolgt von dreimaligem Waschen mit PBS und 2 Mal mit DI-Wasser. Als nächstes wurde das klassische Zell-TEM-Protokoll [17, 18] durchgeführt, und Schnitte mit einer Dicke von 90 nm wurden für die TEM-Beobachtung gesammelt. Die MB-Freisetzung als Funktion des pH-Werts wurde in 6 Zelllinien mit beiden NPs6/100 . untersucht und NPs7.5/80 . Außerdem wurden die OD-Werte aus der Freisetzung von MB und die MB-Freisetzungsprozentsätze für die Datenanalyse aufgezeichnet.
Untersuchung der intrazellulären Aufnahme von MB@SiO2 Nanopartikel
Die konfokale Lebendzellmikroskopie wurde verwendet, um die zelluläre Aufnahme und das intrazelluläre Schicksal von MB@SiO2 . zu beurteilen Nanopartikel. Frühe HepG-2-Zell-Endosomen wurden 16 h lang gefärbt (CellLight Early-Endosomen-GFP, BacMam 2.0 ThermoFisher Scientific C10586, mit Erregung/Emission ~~488/510 nm). Und dann wurden die Zellen mit NPs6/100 . inkubiert (MB-Anregung/Emission:640/650–700 nm) bei einer Nanopartikelkonzentration von 100 μg/ml in bestimmten Zeitintervallen (2 h, 2.5 h, 3 h, 5 h und 6 h). Vor der Aufnahme der Bilder wurde der Lysotracker mit dem LysoTracker™ Red DND-99 (Thermo Fisher Scientific L7528, Anregung/Emission:577/590 nm) 40 min lang gefärbt. Danach die Färbelösung entfernen und die Zellen 2–3 Mal in PBS waschen. Die Bilder wurden mit einem konfokalen Mikroskop Nikon A1R aufgenommen.
Transkriptom-Sequenzierung zur Bewertung der Genexpressionsänderung nach BPSi-Fütterung
Die Gesamt-RNA-Extraktion der Kontrollgruppe und der BPSi-behandelten Gruppe wurde unter Verwendung des Trizol-Reagens nach den Standardverfahren durchgeführt. Die Qualität der anfänglichen Gesamt-RNA-Probe für das Sequenzierungsexperiment wurde mit einem NanoDrop ND-2000-Spektrophotometer nachgewiesen. Gesamt-RNA, die die Qualitätskontrolle bestanden hat, wurde in nachfolgenden Sequenzierungsexperimenten verwendet. Ein Vergleich der Genexpression wurde durch Next-Generation-Sequencing durchgeführt. Alle Sequenzierungsprogramme wurden von der BGI-Shenzhen Corporation (Shenzhen, China) unter Verwendung der BGISEQ-500-Plattform durchgeführt. Durch Sequenzierung erhaltene Rohdaten werden einer Qualitätskontrolle unterzogen, um zu bestimmen, ob die Sequenzierungsdaten für eine nachfolgende Analyse geeignet sind. Führen Sie bei Bestehen eine quantitative Analyse der Gene auf der Grundlage der Genexpressionsniveaus durch und führen Sie eine signifikante Anreicherungsanalyse der Genontologie (GO)-Funktionen der unterschiedlich exprimierten Gene zwischen den ausgewählten Proben durch.
Reverse Transcription Quantitative Polymerase Chain Reaction (RT-qPCR)-Assay zur Bestätigung der Aktivierung des TFEB-CLEAR-Gennetzwerks
Gesamt-RNA wurde aus den kultivierten HepG-2-Zellen der Kontroll- und BPSi-behandelten Gruppen unter Verwendung des Trizol-Reagenzes extrahiert und unter Verwendung des BioRT Master HiSensi cDNA First-Strand-Synthese-Kits (Hangzhou Bioer Technology Co., Ltd.) in cDNA revers transkribiert .) mit Zufallsprimern. cDNA wurde verwendet, um das TFEB-CLEAR-Gennetzwerk durch quantitative PCR mit dem Applied Biosystems™ 7500 Echtzeit-PCR-System (Applied Biosystems, Life Technologies, Carlsbad, CA) mit Aktin als Referenzkontrolle zu amplifizieren. Die für die quantitative RT-PCR verwendeten Primer sind in Tabelle S4 aufgeführt.
Western Blot Assay zur Bestätigung, dass die Autophagie nach der BPSi-Fütterung aktiviert wird
Zelluläre Proteine der Kontrollgruppe und mit BPSi behandelten Gruppen unterschiedlicher Konzentration wurden durch RIPA-Lysat (Heart Biological Technology Co., Ltd.) extrahiert. Der Protease-Inhibitor wurde dem RIPA-Lysat zugesetzt und auf Eis vorgekühlt. Die Zellen wurden dreimal mit vorgekühltem PBS gewaschen. Entleeren Sie die Flüssigkeit vollständig und stellen Sie die Schüssel für 2 min in Eis. 400 Mikroliter RIPA-Lysat wurden auf die Oberfläche der gesamten Schale gegeben, mehrmals mit einer Pipette pipettiert und 30 min auf Eis inkubiert, wobei die Schale mehrmals geschüttelt wurde, um die Zellen vollständig zu lysieren. Die lysierte Zellflüssigkeit wurde in 1 &mgr;ml Eppendorf-Röhrchen überführt und bei 13.000 U/min für 10 min, 4 °C zentrifugiert. Der erhaltene Überstand wurde 10 min in Wasser gekocht und zur späteren Verwendung bei –20°C aufbewahrt. Die Proteinkonzentration wurde mit dem Pierce® BCA Protein Assay Kit (Thermo Scientific) quantifiziert.
Zellextrakte mit 25 µg Gesamtprotein wurden direkt einer SDS-PAGE unterzogen und transferiert. Die Membranen wurden mit 5 % Magermilch blockiert und mit primären Antikörpern sondiert, die P62 erkennen (Proteintech # 18420-1-AP), TFEB (Proteintech # 13372-1-AP), LC 3B (Abcam # ab192890) und β-Aktin (Proteintech # 20536-1-AP). Sekundärantikörper wurden entsprechend der Herkunftsspezies der Primärantikörper ausgewählt und durch verstärkte Chemilumineszenz (Pierce) oder unter Verwendung des Bio-Rad ChemiDoc XRS + Gel Imaging Systems (Bio-Rad, USA) nachgewiesen. Die normalisierte Bandenintensität von P62, TFEB und LC 3B relativ zu β-Aktin wurde durch Densitometrie unter Verwendung der ImageJ-Software in den BPSi-Gruppen quantifiziert, und die Daten sind der Mittelwert ± S.D. aus drei unabhängigen Experimenten.
Messen des Zell-pH-Werts durch PDMPO und pHrodo™ Red Transferrin-Konjugat durch konfokale Mikroskopie
PDMPO-Studie
1 × 10 5 HepG-2-Zellen wurden über Nacht auf sterilen konfokalen Platten kultiviert und die BPSi-Nanopartikel mit einer Konzentration von 100 µg/ml gefüttert. Am nächsten Tag wurden die Objektträger vor der Immunfluoreszenzfärbung dreimal mit 0,01 µM phosphatgepufferter Kochsalzlösung (PBS), pH 7,4 gewaschen, dann wurde 1 µM PDMPO-Farbstoff (Ex/Em = 329/440) zugegeben. Nach dreimaligem Waschen mit PBS wurden die Zellen mit frischem RPMI-1640-Kulturmedium inkubiert und unter einem Fluoreszenzmikroskop (Nikon A1R, Japan) mit einer CCD-Kamera beobachtet und innerhalb von 5 Minuten Bilder aufgenommen, und das Verhältnis von blauer und grüner Fluoreszenzintensität in den Lysosomen wurde dann nach dem Verfahren von Chen et al. [19].
pHrodo™ Red Transferrin-Konjugatstudie
HepG-2-Zellen wurden in konfokalen Platten auf die gleiche Weise für die Zellanhaftung 24 Stunden lang ausplattiert, dann wurden die Platten 10 Minuten lang auf Eis gehalten. Gewaschene Zellen mit kalter Live Cell Imaging Solution, die 20 mM Glucose und 1 % BSA enthält. pHrodo™ Red Transferrin-Konjugat (Ex/Em = 560/585 nm) mit 25 μg/ml in Live Cell Imaging Solution hinzugefügt und bei 37 °C für 20 min inkubiert, dann Zellen in Live Cell Imaging Solution gewaschen. Die Beobachtung erfolgte ebenfalls durch konfokale Mikroskopie. Die quantitative Analyse der Intensität der Mikroskopiebilder wurde mit der Software ImageJ durchgeführt.
Nachweis des lysosomalen pH-Werts von Zellen
Die Zellen A549, HepG-2, HCT8, HCT15, HCT116 und B16 wurden in einem 75 cm2 . kultiviert Kulturflasche und gefüttert mit NPs, wenn die Zellen auf 70 % der Kulturflasche proliferiert waren, und 6 h später wurden die Zellen gesammelt. Die Zellen wurden wiederholt eingefroren und aufgetaut, um das MB vollständig in den Zellen freizusetzen. Die Zelllysate wurden 10 min bei 12000 U/min zentrifugiert. Der Überstand wurde erhalten und seine Absorption bei 660 nm gemessen, um die Gesamtmenge an freigesetztem MB zu berechnen. Die Absorption der NPs6.0/100 bei einem Standard-pH-Wert wurde verglichen, um den pH-Wert jeder Zelle zu erhalten.
Statistische Analyse
Die statistische Analyse wurde mit der Software SPSS15.0 unter Verwendung einer Zwei-Wege-Varianzanalyse (ANOVA) für unabhängige Gruppen und unter Verwendung der Tukey-HSD-Methode für den Mehrfachvergleichstest durchgeführt. Die statistische Signifikanz basierte auf einem Wert von P < 0.05.
Ergebnisse und Diskussion
Wir haben zuerst eine differenzielle Genexpression nachgewiesen, wenn Zellen mit den dualen PEG-funktionellen schwarzen porösen Siliziumdioxid-Nanopartikeln (BPSi-NPs) gefüttert wurden (von unserem Kooperationslabor angeboten [12]). Die Änderung des Zetapotentials von − 18.5 auf + 2.8 mV zeigte ebenfalls, dass die Oberflächen-Doppel-PEGylierung erfolgreich war (Abb. S1a). Der mittlere Durchmesser der BPSi-Nanopartikel betrug 156 nm (Abb. S1b). Basierend auf der Cluster-Heatmap der differentiellen Genexpression (Abb. S2) haben wir mehr als 2-fach differentielle Genexpressionen für die weitere Untersuchung ausgewählt. Go und KEGG wurden eingeführt, um die differentiellen Gene zu analysieren. Aus den Go-Anreicherungsblasenkarten (Abb. 1a) wurden die metabolischen und Lysosomen-assoziierten Gene, einschließlich Phagolysosom-Zusammenbau, Phagozytose und xenobiotischer Stoffwechselprozess, für die weitere Analyse ausgewählt. Bemerkenswerterweise waren die TFEB-CLEAR [17]-assoziierten Genexpressionen signifikant erhöht. Die RT-PCR-Ergebnisse (Abb. 1b) bestätigten auch die Ergebnisse der Gensequenzierung, die Gene auf dem TFEB-dem koordinierten lysosomalen Expressions- und Regulationsweg (CLEAR) nahmen signifikant zu, wie CTSD, CTSF, TFEB, MFN1, LAMP2 und TPP1 . Diese Gene wurden auf dem Lysosomenweg markiert, wie in Abb. S3 gezeigt. Ihre Expression ist höher als die der Kontrollgruppe und hat statistische Signifikanz (P BPSi VS-Steuerung <0,05). Und TFEB reguliert positiv die Expression lysosomaler Gene, kontrolliert die Lysosomenpopulation und fördert den zellulären Abbau lysosomaler Substrate.
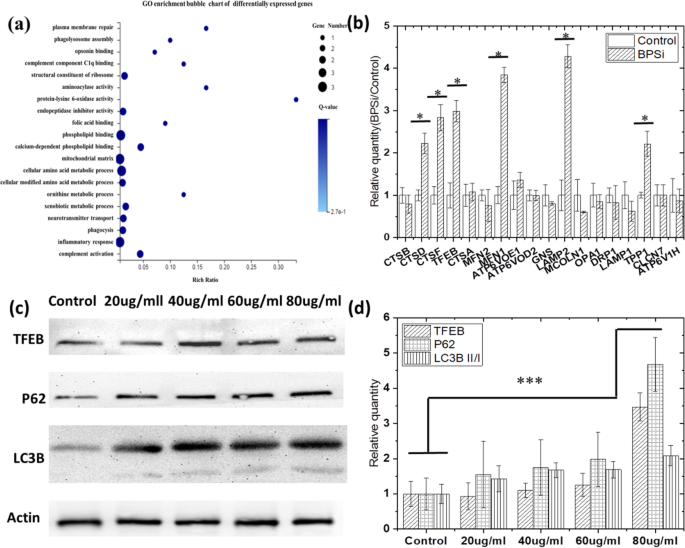
Autophagie wird in HepG-2-Zellen nach der Fütterung von BPSi aktiviert. a GO-Anreicherungsblasenkarte der unterschiedlich exprimierten Gene, die durch Transkriptom-Sequenzierung gefunden wurden. b Überprüfung von Genveränderungen im TFEB-CLEAR-Gennetzwerk nach BPSi-Behandlung durch RT-qPCT-Experimente. c P62-, TFEB-, LC3B II/I-Proteinexpression nach BPSi-Behandlung. d Normalisierte graue Bandenintensität von P62, TFEB, LC3B II/I-Protein in BPSi-behandelten Gruppen gemäß der Kontrollgruppe. Die Daten wurden als Mittelwert ± SD dargestellt.
Darüber hinaus reguliert TFEB die Autophagie, und seine Überexpression führt zu einer signifikanten Zunahme der Autophagosomenproduktion in kultivierten Zellen, da die Hauptfunktion des TFEB-Gens darin besteht, die Biosynthese von Lysosomen zu induzieren und das Auftreten von Autophagie zu fördern [20]. Eine Western-Blot-Analyse wurde auch verwendet, um zu bestätigen, ob die Autophagie auftrat, wenn Zellen mit BPSi-NPs gefüttert wurden. Der Zweck des Western-Tests bestand darin, weiter zu bestätigen, dass die Expression von TFEB und das Auftreten von Zellautophagie nach BPSi-Fütterung erhöht war. Die Proteine LC3B und P62 sind beide Autophagie-Marker. Wenn Autophagie auftritt, nimmt die Expression des Mikrotubuli-assoziierten Proteins 1A/1B-Leichtkette 3B (LC3B) II/I zu. p62 ist ein Rezeptor für Vesikel, die durch Autophagie abgebaut werden, und ist auch ein Rezeptor für zu beseitigende ubiquitinierte Proteinaggregate, und seine Expression nimmt ab, wenn Autophagie auftritt. Also messen wir die Expression dieser Proteine im Western-Experiment.
Aus den Western-Blot-Ergebnissen in Abb. 1c, d, TFEB (P 80 μg/ml VS-Kontrolle = 0,000008), LC3B II/I (P 80 μg/ml VS-Kontrolle = 0,000297) und p62 (P 80 μg/ml VS-Kontrolle = 0,000016) Proteine alle signifikant hochreguliert. Da die Hochregulation von TFEB- und LC3B II/I-Proteinen die Aktivierung der Autophagie anzeigt [18], vermuteten wir, dass die BPSi-Endozytose das Auftreten von Autophagie fördert. Das p62-Protein soll jedoch während des Autophagie-Prozesses aufgrund der Trägerprotein-Natur herunterreguliert werden, die die Endosomen zu Lysosomen gebracht und schließlich abgebaut hat. In unserer Studie zeigte die signifikante Hochregulation von p62 die Beendigung des Abbaus während des Endo-Lysosom-Fusionsprozesses [21], der wahrscheinlich durch einen pH-Anstieg in Endo-Lysosom-Vesikeln verursacht wurde. Somit kann die BPSi-Endozytose zuerst das Auftreten von Autophagie induzieren und dann den Autophagieprozess durch Erhöhung der Endo-/Lysosomen-pH-Werte aufgrund ihrer Amidalkalität hemmen.
Um die pH-Erhöhungseigenschaften in Endo/Lysosomen durch BPSi-Endocytose zu beweisen, wurden in unserer Studie zwei kommerzielle pH-Fluoreszenzsonden verwendet, pHrodo™ Red Transferrin Conjugate (Thermo Fisher # P35376) und RatioWorks™ PDMPO.
pHrodo™ Red als kommerzieller intrazellulärer pH-Indikator zeigt normalerweise bei neutralem pH-Wert schwach fluoreszierend, jedoch zunehmend fluoreszierend, wenn der pH-Wert sinkt. Es sollte den zellulären zytosolischen pH-Wert im Bereich von 9–4 mit einem pKa von ~ 6,5 bei einer Anregung/Emission von 560/585 nm quantifizieren. Wir konnten aus 6 Zelllinienbestimmungen eine qualitative Analyse ziehen, dass die Endozytose von BPSi-NPs die Fähigkeit besitzt, den pH-Wert in Endo/Lysosomen aufgrund der nachlassenden roten Fluoreszenzsignale zu erhöhen (Abb. S4 und S5). Nachdem wir die Experimente jedoch mehrmals nach den Produktbetriebsprotokollen wiederholt hatten, analysierten wir den genauen pH-Wert, der zwischen verschiedenen Zelllinien vor oder nach der Fütterung mit BPSi-NPs abnahm, kaum quantitativ, da keine Korrelation zwischen Intensität und den festgestellten pH-Werten bestand.
PDMPO wurde dann als bessere Lösung zur Anzeige der pH-Wert-Änderungen nach BPSi-Endocytose verwendet, die Ratio-Imaging-Techniken in die quantitative pH-Messung einführt. PDMPO [2-(4-Pyridyl)-5-((4-(2-dimethylaminoethyl-laminocarbamoyl)methoxy)phenyl)oxazol] wird als azidotrope pH-Sonde mit dualer Anregung und dualer Emission charakterisiert. Es emittiert eine intensive grüne Fluoreszenz bei niedrigeren pH-Werten und ergibt eine intensive blaue Fluoreszenz bei höheren pH-Werten. Diese einzigartige pH-abhängige Fluoreszenz macht PDMPO zu einer idealen pH-Sonde für saure Organellen mit pKa = 4,47. PDMPO markiert selektiv saure Organellen (wie Lysosomen) von lebenden Zellen und die beiden unterschiedlichen Emissionspeaks können verwendet werden, um die pH-Schwankungen lebender Zellen in Verhältnismessungen zu überwachen. Es gelang uns jedoch immer noch nicht, die pH-Werte in 6 Zelllinien vor und nach der BPSi-Fütterung zu messen. Wie die in S6 gezeigten Ergebnisse zeigen, wurden bei allen 6 Zelllinien vor und nach der BPSi-Fütterung keine signifikanten Unterschiede beobachtet. Obwohl eine Korrelation zwischen dem Blau/Grün-Verhältnis und den pH-Werten festgestellt wurde (Abb. S7), führt die nichtlineare Korrelation von pH 4–5 dazu, dass die PDMPO-Methode bei der quantitativen Analyse von Endo/Lysosomen vor und nach BPSi-Fütterungen versagt.
Aus den Daten von zwei kommerziellen pH-Indikatoren oben zeigten wir zunächst unseren Verdacht, dass PEG-dekorierte Nanopartikel mit Amid an der PEG-Kette den Endo-/Lysosomen-pH-Wert aufgrund der alkalischen Natur des Amids erhöhen könnten. Ohne die quantitative Analyse präziser pH-Änderungen (0,1 pH-Bereich) können wir jedoch immer noch keine Korrelationen zwischen dem Autophagiestatus und den Endo-/Lysosomen-pH-Werten feststellen und versagten daher bei der Autophagie-Vorhersage.
Basierend auf unserer vorherigen Studie zu selbstzersetzenden Nanopartikeln [13, 14] [15, 22] behielten wir die gleiche Konzentration von Ammoniumhydroxid in 75 % Ethanol bei, passten jedoch die MB- und TEOS-Konzentrationen an. Es wurden zwei Reihen von TEOS-Mengen auf 100 µl und 80 µl eingestellt, um unterschiedliche Schalendicken und Porengrößen zu erhalten. Es wurden zehn Serien von MB-Mengen eingestellt, um unterschiedliche Größen der Mittelhohlstruktur und MB-Ladewirkungsgrade zu erhalten.
Die in den Protokollen hinzugefügten MB- und TEOS-Mengen waren wie in Tabelle 1 unten beschrieben.
Wie in Abb. 2a, b gezeigt, nahm die Nanopartikelgröße mit der Zunahme der MB-Menge in beiden TEOS-Konzentrationen (100 µl und 80 µl) zu. Bei der gleichen MB-Konzentration nahm die Partikelgröße mit der Zunahme der TEOS-Menge zu. Moreover, with the increase of the TEOS amount, the shell thickness grew up, which has been proved by the element mapping (shown as Fig. 2c). The polydispersity index (PDI) and surface charge of the nanoparticles are shown in Fig. S8 and Table S1. The morphology studies predicted that with the increase of MB amount, the loading efficiency will grow up, leading to the faster release profile, while with the increase of TEOS amount, the release will slow down. And we need to find out the appropriate MB and TEOS concentration, with which we could obtain the optimized nanoparticle systems, that we may be able to make the MB release profile linear correlated with the pH changes.

Morphology characterization of 10 different self-decomposable nanoparticles with specific MB or TEOS amount. a STEM figures. b Nanoparticle size distribution analysis. c Si mapping of 10 self-decomposable nanoparticles. Scale bars in all figures are 100 nm. The size distribution analysis was performed by randomly chosen 100 nanoparticles from STEM figures and measured by ImageJ software. Data was presented as mean ± S.D.
The MB loading efficiency was determined by UV-Vis spectrum. The standard curve (Fig. S9) of MB was firstly drawn using series concentrations of MB solution (from 6.25 to 46.88 μg/mL), with the equation as y = 67.63x + 0.10919, R 2 = 0.9987. As calculated with the equation above, we obtain MB loading efficiency of 10 self-decomposable nanoparticles with specific parameters, detailed data shown in Fig. S10.
Before the study of the MB release profiles in different pH solutions, the release profiles in pure water have been studied. As shown in Fig. S11 and Fig. S12, all the nanoparticles with TEOS amount of 80 μL presented increased MB release along with the duration increase, which was reflected by the UV–Vis absorption. Moreover, with the MB encapsulated amount increase, the growth trend of MB release becomes more significant. Also, the release velocity grows faster. However, as the TEOS amount increase to 100 μL, the particle surface became more densed and the release becomes slower when the MB amount below 3.0 mg; almost no increase trend could be observed in the MB release in water during 14 days of release. As long as the MB amount increases to above 4.0 mg, an obvious increase trend of MB release could be observed. One thing to be noticed is that the nanoparticle parameter of both NPs7.5/80 and NPs6.0/100 presented solid growth as the time prolongs, almost showed a linear increase trend during the first 7 days, and then reached the platform.
Then, we focused on the MB release behavior in different pH buffers to figure out whether self-decomposable nanoparticles with specific parameters could have the linear pH-dependent MB release.
Firstly, we carried out the MB release experiments at pH 4.0 buffer solution. From Fig. S13, we could easily reach the conclusion that with the same TEOS amount of 100 μL, the MB release velocity presented a similar trend was observed in the 5 nanoparticle systems of TEOS at 80 μL (Fig. S14), the center positive correlation with the MB encapsulated amount.
Concentrated MB diffuses into the surrounding solution via diffusion due to concentration difference. The bigger concentration gradient makes the faster MB release. Compared with the MB release in pure water, we found that the acidic environment speeded up the release of MB (Fig. S13 and S14 compared with Fig. S11 and S12), indicating that the MB release is not only driven by diffusion; however, in acidic solutions, electrostatic repulsion is also an important driven force due to the positive charge nature of MB. We then calculated the release percentage of each nanoparticle parameter according to the MB loading efficiency, MB standard curve, and the dilution ratio at measurements. The release percentage reflected the release speed of MB in pH 4.0 acidic solution, and the results (Fig. 3) showed that only the release percentage of NPs7.5/80 presented linear release in pH 4.0 solution. Other nanoparticle systems with specific MB and TEOS parameters showed similar release trends, and the release percentage did not have a linear growth. One exception is NPs6/100 , and the MB release reached the platform in only 72 h; thus, it was hard to tell whether the MB release could grow linear before that duration at this stage.
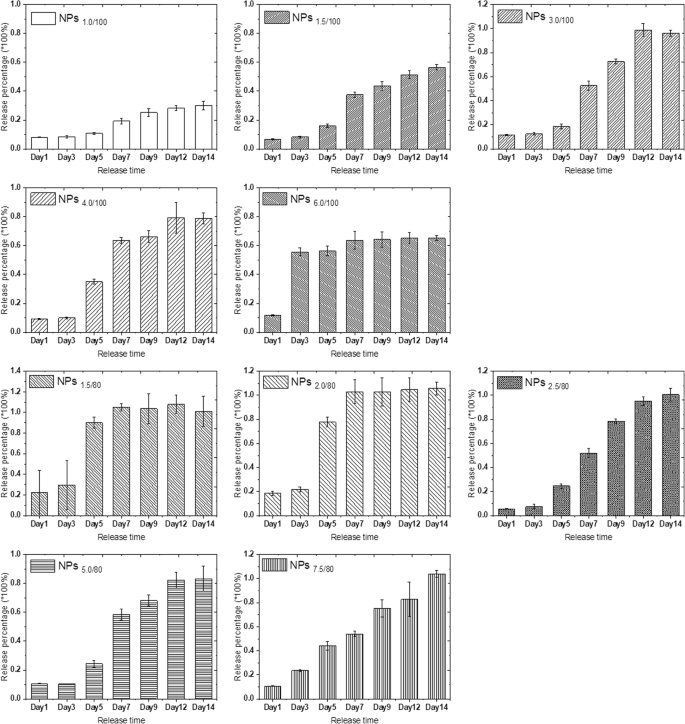
MB release percentage of 10 series self-decomposable nanoparticles after a specific duration in pH 4.0 buffer. All experiments were triple repeated, and the data were shown as mean ± S.D.
Meanwhile, we tested the MB release profiles in near-neutral and alkali buffers (pH 6.86 and pH 9.18). The results in both Fig. S15, S16, Fig. S17, and S18 demonstrated that the MB release slowed down with the solution pH increase to 6.86; moreover, with the central MB concentration increased, the MB release percentage decreased. At pH 9.18, all nanoparticles with 10 specific parameters presented a very slow MB release (Fig. S19 and S20); no matter in UV–Vis absorption or the release percentage, the trend was similar with the one in pH 6.86 buffer, but with even lower release percentage. So, it was clear that the self-decomposable nanoparticles only presented MB release linear growth in acidic solutions. We thought back to the endo/lysosomes pH, from 4 to 5, which is exactly the pH range of MB linear growth as a function of time in a specific MB/TEOS parameter. Thus, we get more confident that the self-decomposable nanoparticle system may be an accurate measuring tool for quantitative determining the endo/lysosome average pH, then provide evidence on the exact pH value of autophagy status.
The precondition of using the specific self-decomposable nanoparticles as an endo/lysosome pH indicator is that the nanoparticles stay stable in the endo/lysosome during the whole measurement process. Secondly, the MB release in endo/lysosome should occur smoothly when the measurement carried out.
The colocalization of the nanoparticle in the endo/lysosomes by cell TEM study and the MB release in 6 different cell lines were studied. From the cell TEM results, all of the 10 series nanoparticles stayed in the endo/lysosomes without escaping, after 24-h incubation with the HepG-2 cells (Fig. 4). Since the diameter of HepG2 cells used in Fig. 4 is about 10–20 μm and the diameter of MB@SiO2 nanoparticles is between 75 and 200 nm, it will be very difficult to clarify the nanoparticle morphologies using the images with low magnification (as shown in Fig. S21). We also investigated the intracellular location of the nanoparticles in the other 5 cell lines, and 4 nanoparticles were randomly selected to demonstrate the nanoparticles were trapped in the endo/lysosomes (Fig. S22). The nanoparticles with all parameters showed a central hollow structure in all other 5 cell lines after 24-h incubation, indicating the MB release. Moreover, under more precise observation, we noticed the MB release may be different due to different hollow sizes, pointing to the fact that (1) the endo/lysosome pH in different cells is different and (2) the MB release from the nanoparticles is very sensitive to the endo/lysosomes pH, especially for the NPs6/100 and NPs7.5/80 . From Fig. S23, we can find that nanoparticles have already realized the endocytosis and stayed in the vesicles 2 h after nanoparticle feeding, then nanoparticles gradually accumulated in lysosomes.
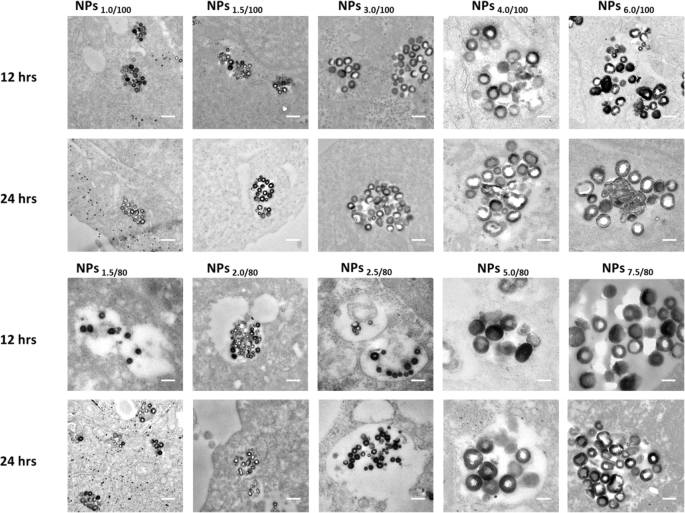
The colocalization of the nanoparticle in the endo/lysosomes by cell TEM study after 12 and 24 h incubation with the 10 series nanoparticles. The scale bar is 200 nm in the TEM images
We then evaluated the correlation between the pH values and the OD values in pH 4.0–4.8. From the results in Fig. 5 and Fig. S24, for NPs6/100 and NPs7.5/80 nanoparticle systems, the MB release presented a linear decrease as a function of pH in the pH range from 4.0 to 4.8.
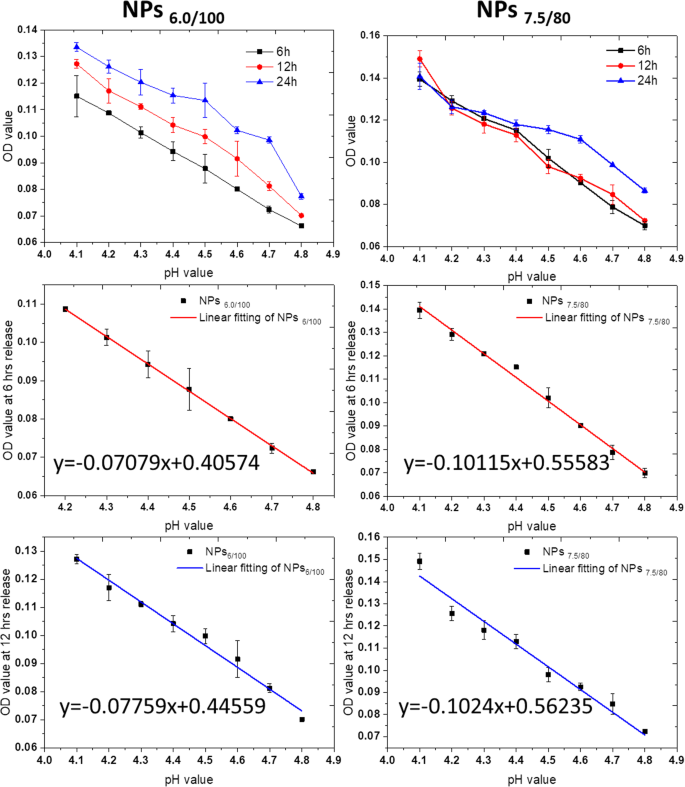
MB release as a function of pH values in NPs6/100 and NPs7.5/80 after specific incubation duration, 6 h, 12 h, and 24 h. The linear correlation equations were also calculated for 6 h and 12 h for MB release from both NPs6/100 and NPs 7.5/80 as a function of pH values. All experiments were carried out triplicated, and the data were shown as mean ± S.D.
We then converted the OD value to the MB release percentage according to the MB loading efficiency and feeding amount. As shown in Fig. S25, in the first 6 h, the MB release percentage in NPs6/100 and NPs7.5/80 nanoparticle systems also presented as a function of pH values. We then calculated the residual sum of squares and Pearson’s related coefficient at 6 h and 12 h release duration, respectively, as the residual sum of squares present a negative correlation with closeness of linear fitting, while the closer the absolute value of Pearson’s related coefficient to 1, the more linear it is. As shown in Table S2 and S3, the highest degree of linearity is the fitting of NPs6.0/100 nanoparticle systems, followed by the one of NPs7.5/80 at 6 h release.
Till then, we were so excited by the results that the method for precisely monitoring the pH values has been established, especially with the accuracy less than or equal to 0.1 pH value interval. That means, we have great possibilities to figure out the correlation between endo/lysosome pH values and the autophagy status, which is of great significance for better studying the autophagy mechanism and predicting the autophagy process. As we can see in Fig. S26, the MB release in HepG-2 cells have already reached the plateau after incubation for 4 h. Thus, we chose 6 h as the observation time point.
We then carefully investigated the MB release of NPs6.0/100 in 6 cell lines in the nanoparticle cell interaction duration of 6 h, including liver cancer HepG-2 cell line, colon cancer HCT8, HCT 15, and HCT 116 cell lines, lung cancer A549 cell line, and myomelanocytic cancer B16 cell line.
As shown in Table 2, we clearly differentiate the endo/lysosomes in 6 cancer cell lines, with the accuracy at 0.01 pH values, which is impossible to be done with the commercial intraocular pH indicator kits.
Moreover, we re-evaluated pH values in endo/lysosomes of the HepG2 cells before and after cultured with BPSi nanoparticles. We reached the conclusion that BPSi uptake significantly increases the endo/lysosome pH values, from 4.70 ± 0.09 to 5.59 ± 0.05, perfectly illustrating the reason for BPsi uptaken induced the autophagy initially then terminated the autophagy flux. The intracellular uptake of BPSi makes the quantities of the endo/lysosomes increased, which was consistent with the results of gene sequencing, that autophagy-related genes (TFEB-CLEAR) were activated. Meanwhile, the autophagy termination by the increased pH values in endo/lysosomes also coincide with the results of p62 proteins upregulation in Western blot study.
Diskussion
Nanoparticles can generally cause autophagy in cells [23], and studies have shown that the autophagic response to nanoparticles presenting a neutral or anionic surface involves enhanced clearance of autophagic cargo. Cell exposure to nanoparticles presenting a cationic surface, on the other hand, results in transcriptional upregulation of the TFEB pathway, but also causes lysosomal dysfunction, ultimately resulting in blockage of autophagic flux [7]. And our results are in consistent with these previous conclusions. In our study, we found that the expression of autophagy-related genes and proteins in HepG2 cells has been increased after feeding of BPSi nanoparticles through transcriptome sequencing, RT-qPCR, and Western experiments. However, the expression level of autophagy-related P62 protein does not decrease as the autophagy is activated. We suspect that the PEG-amine on the surface of BPSi nanoparticles raises the pH value of the lysosome, resulting in inhibition of P62 degradation. Existing lysosomal pH indicators cannot verify our guess. To accurately measure the lysosomal pH of living cells, we established a new method for endo/lysosomes pH qualitative determination based on self-decomposable nanoparticle systems. Ten nanoparticle systems with specific MB/TEOS parameters were employed for obtaining optimized pH sensitively responsive measurement method. The radial MB concentration gradient from inner out served as a major driving force for MB release. The drug release proceeded with simultaneously carrier decomposition, which was driven by a diffusion-controlled mechanism. Moreover, as the pH value decreases, the hydrogen ion concentration increases, and the enhanced electrostatic interaction promotes inner MB to release faster than in neutral solution [24]. The optimized central hollow nanoparticle system could release the central concentrated MB as a linear function of precise pH values in the range of pH 4.0–4.8, which is exactly the pH of lysosomes. Finally, by this qualitative pH indicator based on self-decomposable nanoparticles, we have succeeded in the detection of the average pH values of lysosomes in 6 cell lines. Moreover, by this system, we can qualitatively differentiate the pH changes of lysosomes before and after BPSi nanoparticle endocytosis by HepG-2 cells, clarifying the mechanism of the autophagy occurrence and then termination after BPSi endocytosis. The self-decomposable nanoparticle systems pave a brand new way for studying the luminal pH values, providing new tools to know better of the cell signaling and metabolism, and then providing new ways and methods for the treatment of cancer [25, 26].
Schlussfolgerung
In this study, we found that BPSi can promote cell autophagy through transcriptome sequencing, but the amino groups on the surface of the nanoparticles can increase the pH of the lysosome and inhibit the degradation of autophagic flow. Thus, the lysosome pH significantly influences the autophagy stages. And precisely acquiring the information of lysosome pH will promote the perceiving of autophagy. However, the existing fluorescent lysosomal pH indicators could only determine a wide range of lysosomal pH; thus, we established a precise lysosomal pH indicator based on the self-dissociation system. By adjusting the synthesis parameters of MB@SiO2 , the release of MB loaded on the nanoparticles was linearly and negatively correlated with pH. And the nanoparticles mainly stay in the lysosome after entering the cell. By measuring the amount of MB released in the cells, the pH value of the lysosome can be calculated exactly according to the linear function. The established precise pH indicator provided a brand new tool and methodology to precisely study the lysosome pH values and further acquire more information on autophagy.
Verfügbarkeit von Daten und Materialien
Alle während dieser Studie generierten oder analysierten Daten sind in diesem veröffentlichten Artikel und seinen ergänzenden Informationsdateien enthalten.
Abkürzungen
- BPSi:
-
Black mesoporous silicon
- FBS:
-
Fötales Rinderserum
- GO:
-
Gene ontology
- HCl:
-
Salzsäure
- KEGG:
-
Kyoto Encyclopedia of Genes and Genomes
- MB:
-
Methylene blue
- NaBr:
-
Sodium bromide
- NaSi:
-
Natriumsilicid
- NH4Br:
-
Ammonium bromide
- NPs:
-
Nanopartikel
- OD:
-
Optische Dichte
- PBS:
-
Phosphatgepufferte Kochsalzlösung
- PDMPO:
-
2-(4-Pyridyl)-5-((4-(2-dimethylaminoethy- laminocarbamoyl) methoxy) phenyl) oxazole
- RT-qPCR:
-
Reverse transcription quantitative polymerase chain reaction
- TEOS:
-
Tetraethylorthosilikat
Nanomaterialien
- SaaS-Anwendungsüberwachung:Den Wald und die Bäume sehen
- Die Entwicklung und Anwendung von Glasfasergewebe
- Herstellung von ICA-beladenen mPEG-ICA-Nanopartikeln und ihre Anwendung bei der Behandlung von LPS-induzierter H9c2-Zellschädigung
- Ultraschmalbandiger perfekter Absorber und seine Anwendung als plasmonischer Sensor im sichtbaren Bereich
- Die potenzielle Leber-, Gehirn- und Embryotoxizität von Titandioxid-Nanopartikeln bei Mäusen
- Bildung und Lumineszenzeigenschaften von Al2O3:SiOC-Nanokompositen auf der Basis von durch Phenyltrimethoxysilan modifizierten Aluminiumoxid-Nanopartikeln
- Die Herstellung einer Au@TiO2-Dotter-Schale-Nanostruktur und ihre Anwendungen für den Abbau und den Nachweis von Methylenblau
- Grüne Kompetenz bei der Synthese und Stabilisierung von Kupfernanopartikeln:katalytische, antibakterielle, zytotoxische und antioxidative Aktivitäten
- TEM- und STEM-Studien zu den Querschnittsmorphologien von Dual-/Tri-Layer-Breitband-SiO2-Antireflexfolien
- Kondensatorfolie:Eigenschaften, Aufbau und Anwendung



