Ein hochempfindlicher elektrochemischer DNA-Biosensor aus Acryl-Gold-Nanokomposit zur Bestimmung des Geschlechts von Arowana-Fischen
Zusammenfassung
Die vorliegende Forschung beschreibt eine einfache Methode zur Identifizierung des Geschlechts von Arowana-Fischen (Scleropages formosus ). Der DNA-Biosensor war in der Lage, spezifische DNA-Sequenzen auf extrem niedrigem Niveau bis hinunter zu Atto-M-Regimen nachzuweisen. Ein elektrochemischer DNA-Biosensor basierend auf einem Hybridkomposit aus Acryl-Mikrosphären-Gold-Nanopartikel (AcMP-AuNP) wurde hergestellt. Hydrophobe Poly(n-butylacrylat-N-acryloxysuccinimid)-Mikrokügelchen wurden mit einem einfachen und gut etablierten einstufigen Photopolymerisationsverfahren synthetisiert und physikalisch an den AuNPs an der Oberfläche einer Kohlenstoff-Siebdruckelektrode (SPE) adsorbiert. Der DNA-Biosensor wurde einfach durch Aufpfropfen einer aminierten DNA-Sonde auf die Succinimid-funktionalisierten AcMPs über eine starke kovalente Bindung konstruiert. Die DNA-Hybridisierungsreaktion wurde durch Differential-Pulsvoltammetrie-(DPV)-Technik unter Verwendung einer Anthrachinonmonosulfonsäure-Redoxsonde als elektroaktiver Oligonukleotid-Marker (Tabelle 1) bestimmt. Eine niedrige Nachweisgrenze bei 1,0 × 10 −18 M mit einem breiten linearen Kalibrierungsbereich von 1,0 × 10 –18 bis 1,0 × 10 –8 M (R 2 = 0.99) kann durch den vorgeschlagenen DNA-Biosensor unter optimalen Bedingungen erreicht werden. Der elektrochemische Nachweis von Arowana-DNA kann innerhalb von 1 Stunde abgeschlossen werden. Aufgrund seiner geringen Größe und seines geringen Gewichts ist der entwickelte DNA-Biosensor vielversprechend für die Entwicklung eines funktionellen Kits für die Verwendung in Fischkulturen.
Hintergrund
Asiatische Arowana (Scleropages formoss ), ein Süßwasserfisch, [1] ist in der Landschaft Südostasiens wie Malaysia, Singapur, Thailand, Indonesien, Kambodscha, Vietnam, Laos, Myanmar und auf den Philippinen weit verbreitet. Darüber hinaus kommt der Arowana-Fisch auch in Australien und Neuguinea vor [1,2,3,4]. Es ist im Volksmund als Drachenfisch, Asia-Knochenzunge, Kelisa oder Baju-Rantai bekannt [5, 6]. Er überlebt noch als primitive Fischart aus der Jurazeit [7, 8]. Die Chinesen und Asiaten betrachteten sie wie viele andere Kulturen als Symbol für Glück und Glück [6]. Im Allgemeinen ist die Arowana im ausgewachsenen Alter etwa 7 kg schwer und 1 m lang [9]. Dieser Zierfisch besitzt attraktive Farben und Morphologie und ist an seinen charakteristischen körperlichen Merkmalen zu erkennen, wie z. Es gibt drei Hauptfarbvarianten, d. h. golden, rot und grün von eng verwandten Süßwasserfischen innerhalb der asiatischen Arowana-Arten. Es gibt auch mehrere andere unterschiedliche Arten, die aus verschiedenen Teilen Südostasiens stammen und für viele Flusssysteme regional sind [8].
Aufgrund ihrer hohen Popularität und großen Nachfrage in Zierzwecken wurde die asiatische Arowana heftig nach Gewinn gejagt [6] und führt zu einem schnellen Rückgang ihrer Population. Angesichts ihres hohen Bedarfs in der Zierpflanzenindustrie, der Übernutzung natürlicher Populationen und der Seltenheit natürlicher Lebensräume aufgrund von Veränderungen in der Lebensumgebung wird die Asiatische Arowana seit 1980 durch das Übereinkommen über den internationalen Handel als vom Aussterben bedrohte Art eingestuft in Endangered Species of Wild Fauna and Flora (CITES) und wurde kürzlich auf der Roten Liste der IUCN von 2006 als gefährdet eingestuft [1, 3, 8, 10, 11]. Der kommerzielle Handel mit dieser vom Aussterben bedrohten Art ist jedoch gemäß CITES verboten, außer in bestimmten Ländern, z. B. Indonesien, Singapur und Malaysia. [2, 3, 12]. Es gibt eine Reihe von CITES-registrierten Züchtern in Asien, die aktiv die Zucht und den Handel mit Arowana-Fischen betreiben [2, 12]. Dieser asiatische Süßwasserfisch besteht aus geografisch isolierten Stämmen und ist das einzige Mitglied der Art mit unterschiedlichen Farbvarianten, die auf unterschiedlichen geografischen Verteilungen in den Flüssen Südostasiens basieren. Die Verbreitung der Arten ist heute weitaus weiter verbreitet und erstreckt sich auf den Nil in Afrika, den Amazonas in Südamerika, Australien und Neuguinea [1, 4, 8].
Unter den verschiedenen Farben der asiatischen Arowana sind rote und goldene Arowana-Fische im Vergleich zu schwarzen, grünen, silbernen und anderen Farbvarietäten die teuersten und beliebtesten Ziertiere in der Brutindustrie [1, 5, 10, 13]. Das Phänomen des Eierdiebstahls der Asiatischen Arowana ist im Vergleich zu anderen Fischarten untypisch. Im Allgemeinen werden Arowana-Fische im Alter von 3–4 Jahren ausgewachsen und legen nur wenige Eier (30–100) [14, 15] von extragroßer Größe (ca. 1 cm Durchmesser) [16]. Interessanterweise werden die befruchteten Eier und Larven dann geschützt und im Maul männlicher Arowana-Fische aufgezogen, und sie zeigen eine hohe elterliche Fürsorge. Die Identifizierung des Geschlechts anhand der visuellen Beobachtung des Arowana-Babys ist schwierig, da es kein ausgeprägtes phänotypisches Organ des Sexualdimorphismus gibt [14, 15]. Nur ein Elternteil (vermutlich das Männchen) kann identifiziert werden, da die Nachkommen aus seinem Maul geerntet werden. Der andere Elternteil kann nicht unter einer Reihe potenzieller Elternteile identifiziert werden [16].
Üblicherweise halten die Bastler die Arowana-Jungfische sowohl zu Zierzwecken im Aquarium als auch zur Zucht in der Fischzucht. Alle Arten von jungen Arowana-Fischen werden jedoch zum gleichen Preis verkauft, da es an unterstützender Technologie zur Unterscheidung von Geschlecht und Farbvielfalt fehlt. Bis heute gibt es keine etablierte Methode, um das Geschlecht und die Farbe von Arowana-Fischen im Jugendstadium zu identifizieren. Stattdessen wurden Hunderte von Studien mit DNA-Analysen durchgeführt, die auf der genetischen Struktur und Biographie von Arowana-Fischen basieren, um das Geschlecht und die Farbe in ihrem frühen Alter zu identifizieren. Eine traditionelle Methode, die auf der Schätzung der Körpergröße und der Mundhöhle basiert, kann nur im Alter von etwa 3 Monaten des Arowana-Babys zur Identifizierung von Geschlecht und Farbe durchgeführt werden [17]. Diese herkömmliche visuelle Untersuchungsmethode ist jedoch zeitaufwendig und liefert oft ungenaue Ergebnisse. Andererseits sind die weit verbreiteten Standardmethoden auf Basis der DNA-Sequenzierung, d. h. Polymerase-Kettenreaktion (PCR) und Gelelektrophorese, arbeits-, zeit- und ressourcenintensiv. Ein alternativer Algorithmus der Methode der erfinderischen Problemlösung (ARIZ) wurde zuvor für die Erkennung der Geschlechtserkennung von Arowana verwendet [18]. ARIZ ist ein alternatives Tool zur Geschlechtserkennung, das neun verschiedene Teile und insgesamt 40 komplexe Schritte enthält. Es erfordert sehr viel Zeit zum Erlernen und Üben und erfordert sehr erfahrenes Personal für die Bedienung. Zum Beispiel wurde die Anwendung von ARIZ in verschiedenen Engineering-Systemen eingesetzt, aber die meisten Fälle deckten nicht alle Anforderungen und Prozesse von ARIZ ab.
In dieser Forschung wurden Acrylpolymer-Mikrokügelchen, die mit funktionellen Succinimid-Gruppen über N-Acryloxysuccinimid (NAS)-Einheiten modifiziert waren, als Matrix für die Immobilisierung von DNA-Sonden verwendet. Wie bereits von Chen und Chiu 2000 und Chaix et al. 2003 [19, 20] kann die funktionelle Succinimidgruppe mit funktionellen Amingruppen unter Bildung einer kovalenten Bindung reagieren. Der Einbau der NAS-Funktionalität in Acrylmikrokügelchen für DNA-Mikrobiosensoranwendungen bietet die Vorteile eines einfachen Herstellungsverfahrens, bei dem die Kügelchen über ein einstufiges Verfahren unter Verwendung von Photopolymerisation in kurzer Zeit (mehrere Minuten) synthetisiert und funktionalisiert werden können. Darüber hinaus haben die Mikrokügelchen den Vorteil einer geringen Größe und bieten eine große Oberfläche für die Immobilisierung der DNA-Sonde, wodurch die Diffusionsbarriere für Reaktanten und Produkte verringert wird. Dies ermöglicht die Verbesserung der Biosensorleistung in Bezug auf kürzere Reaktionszeiten und einen breiteren linearen Reaktionsbereich, was in der hier berichteten Arbeit demonstriert wird.
In dieser Studie wird ein elektrochemisches DNA-Biosensor-Verfahren vorgeschlagen, das hochempfindlich, einfach, leicht herzustellen und kostengünstig ist, um das Geschlecht juveniler Arowana-Fische mit hoher Genauigkeit zu bestimmen. Der DNA-Biosensor wurde aus einer mit kolloidalen Goldnanopartikeln (AuNPs) modifizierten Kohlenstoff-Siebdruckelektrode (SPE) und mit einer NAS-Funktionsgruppe funktionalisierten Polyacrylat-Mikrokügelchen aufgebaut. Die AuNPs wurden über elektrostatische Wechselwirkung auf der Kohlenstoff-SPE-Oberfläche immobilisiert und spielten eine wichtige Rolle bei der Verbesserung der Elektrodenleitfähigkeit und der Erleichterung des Elektronentransfers, während die Acrylmikrokugeln (AcMPs) durch physikalische Adsorption direkt auf die AuNP-modifizierten SPE abgeschieden wurden. Die aminierte DNA-Sonde von Arowana wurde dann kovalent an das immobilisierte AcMP-AuNP-Komposit an der exponierten Succinimidgruppe der AcMPs gebunden. Die Sonden-Target-Hybridisierung wurde mit Anthrachinon-Redox-Markierung mittels Differential-Puls-Voltammetrie (DPV) nachgewiesen. Der Einbau kleiner und einheitlicher AcMPs konnte eine große DNA-Beladungskapazität halten und die Empfindlichkeit und Nachweisgrenze des elektrochemischen arowana DNA-Biosensors erhöhen.
Methoden
Geräte und Elektroden
Alle elektrochemischen Messungen wurden mit DPV unter Verwendung von Autolab PGSTAT 12 Potentiostat/Galvanostat (Metrohm) bei 0,02 V Stufenpotential innerhalb des Potentialfensters von –1,0 V bis –0,1 V durchgeführt. Mit AcMPs und AuNPs modifiziertes SPE von Scrint Technology Co. Malaysia wurde als die Arbeitselektrode. Als Hilfs- bzw. Referenzelektroden wurden eine stabförmige Platin(Pt)-Elektrode und eine Ag/AgCl-Elektrode, gefüllt mit 3,0 M KCl-Innenlösung, verwendet. Das Ultraschallbad Elma S30H wurde verwendet, um homogene Lösungen herzustellen.
Chemikalien
2–2-Dimethoxy-2-phenylacetophenon (DMPP) wurde von Fluka bezogen. 1,6-Hexandioldiacrylat (HDDA), n-Butylacrylat (nBA) und Au(III)-chloridtrihydrat wurden von Sigma-Aldrich geliefert. Die kolloidalen AuNPs wurden nach der von Grabar et al. (1995). Natriumdodecylsulfat (SDS) und NaCl wurden von Systerm bezogen. NAS und Anthrachinon-2-sulfonsäure-Monohydrat-Natriumsalz (AQMS) wurden von Acros bezogen. Milli-Q-Wasser (18 ml) wurde verwendet, um alle chemischen und biologischen Lösungen herzustellen. Die Stammlösung der DNA-Sonde wurde mit 0,05 M K-Phosphat-Puffer (pH 7,0) verdünnt, während komplementäre DNA (cDNA) und nicht-komplementäre (ncDNA) Lösungen mit 0,05 M Na-Phosphat-Puffer bei pH 7,0 mit 1,0 mM . hergestellt wurden AQMS. Der K-Phosphat-Puffer erleichtert die maximale Immobilisierung der DNA-Sonde auf dem Succinimid-funktionalisierten Acrylmaterial, während der Na-Phosphat-Puffer optimale Bedingungen für die DNA-Hybridisierungsreaktion bietet [21, 22].
Synthese von Acryl-Mikrosphären
AcMPs wurden nach den zuvor beschriebenen Methoden mit leichten Modifikationen hergestellt [22]. Kurz gesagt wurde eine Mischung aus 450 μl HDDA, 0,01 g SDS, 0,1 g DMPP, 7 ml nBA-Monomer und 6 mg NAS in 15 ml Milli-Q-Wasser gelöst und bei Raumtemperatur (25 °C) beschallt ) für 10 Minuten. Danach wurde die Emulsionslösung mit UV-Licht 600 s lang unter einem kontinuierlichen Fluss von N2 . lichtgehärtet Gas. Die resultierenden Poly(nBA-NAS)-Mikrokügelchen wurden dann durch Zentrifugation bei 4000 UpM für 30 Minuten gesammelt, gefolgt von dreimaligem Waschen in K-Phosphatpuffer (0,05 M, pH 7,0) und bei Umgebungstemperatur trocknen gelassen.
Herstellung eines DNA-Biosensors unter Verwendung von Acryl-Mikrosphären
Vor der Oberflächenmodifizierung wurde das Kohlenstoff-SPE gründlich mit DI-Wasser gespült, mit den Acrylpolymer-Mikrokügelchen mit 3 mg/ml tropfenbeschichtet und bei Umgebungsbedingungen an der Luft trocknen gelassen, gefolgt von einem Tropfenguss mit 5 mg/ml kolloidale AuNPs. Die elektrochemischen Eigenschaften von Kohlenstoff-SPE vor und nach der Modifizierung mit AcMPs und AuNPs wurden mit der CV-Methode untersucht. Abbildung 1 zeigt die Methode, die aus der 3-stufigen Herstellung eines elektrochemischen DNA-Biosensors und der 1-stufigen arowana-cDNA-Detektion besteht. Etwa 10 μL kolloidaler AuNPs (1 mg/300 μL) wurden zunächst auf einem Kohlenstoff-SPE abgeschieden und bei 25 °C luftgetrocknet. Da die AcMPs (1 mg) leicht in Ethanol (100 &mgr;l) suspendiert wurden, um eine stabile Dispersion zu bilden, wurden 10 &mgr;l AcMP-Suspension auf das AuNP-modifizierte SPE tropfenbeschichtet. Das AcMP-AuNP-modifizierte Kohlenstoff-SPE wurde dann für 6 h in 300 μl 5 μM arowana DNA-Sondenlösung getaucht, damit der DNA-Immobilisierungsprozess stattfinden konnte, und vorsichtig mit K-Phosphatpuffer (0,05 M, pH 7,0) dreimal gewaschen, um Entfernen Sie die ungebundene Einfangsonde. Die immobilisierte DNA-Sonde wurde später in 300 μl Ziel-DNA-Lösung mit 2 M NaCl und 1 mM AQMS eingetaucht, um eine DNA-Hybridisierung und Interkalationsreaktionen innerhalb einer Stunde zu ermöglichen, gefolgt von einem sequentiellen Spülen mit Milli-Q-Wasser und Na-Phosphat Puffer (0,05 M, pH 7,0) zur Entfernung von nicht hybridisierten DNA-Fragmenten und einer spezifischen Bindung des elektrochemischen AQMS-Markers. Alle DPV-Messungen wurden in 4,5 ml 0,05 M K-Phosphat-Puffer bei pH 7,0 und Raumtemperatur durchgeführt.
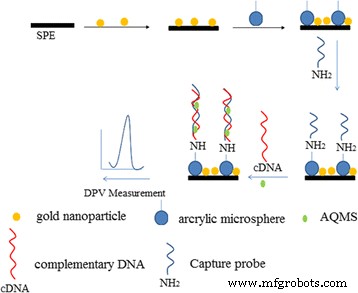
Das Herstellungsverfahren des elektrochemischen arowana DNA-Biosensors basierend auf einer AcMP-AuNP-modifizierten Elektrode
Optimierung des elektrochemischen Arowana-DNA-Biosensors
Die mit dem jeweiligen AcMP-, AuNP- und AcMP-AuNP-Komposit modifizierten DNA-Elektroden wurden für cDNA- (5 μM) und ncDNA-(5 μM)-Tests mit der elektroanalytischen DPV-Methode in Gegenwart von 1 mM AQMS und 2 M NaCl at . verwendet die Scanrate von 0,5 V/s gegenüber der Ag/AgCl-Referenzelektrode. Die Dauer der Immobilisierung der DNA-Sonde wurde durch separates Einweichen von neun Einheiten AcMP-AuNP-modifizierter SPEs in 300 μl 5 μM arowana DNA-Sondenlösung für 1, 2, 3, 5, 6, 7, 8, 12 und 18 Stunden bestimmt, bevor Reaktion mit 5 &mgr;M cDNA in DNA-Hybridisierungspuffer (0,05 M Na-Phosphatpuffer bei pH 7,0), der 1 mM Antrachinon-Redox-Interkalator und 2 M NaCl enthält. Die DNA-Hybridisierungszeit wurde durch Eintauchen der DNA-Elektrode in 300 μL einer 5 μM cDNA-Lösung in Gegenwart von 2 M NaCl und 1 mM AQMS für 10–100 min untersucht. Der Einfluss der Temperatur auf die DNA-Hybridisierungsdauer wurde durch Messung der arowana DNA-Biosensor-Antwort bei 4, 25, 40 und 50 °C für eine Versuchsdauer von 5–90 min im Messpuffer mittels DPV-Technik gemessen. Für die Untersuchung des pH-Effekts wurde der arowana-DNA-Biosensor in 5 μM cDNA-Lösung getaucht, die aus 0,05 M Na-Phosphat-Puffer hergestellt wurde, der mit 2 M NaCl und 1 mM AQMS zwischen pH 5,5 und pH 8,0 konditioniert war, gefolgt von einer DPV-Messung. Die Wirkung verschiedener positiv geladener Ionen (z. B. Ca 2+ , Na + , K + , und Fe 3+ Ionen) auf die elektrochemische Antwort des arowana-DNA-Biosensors wurde durch Zugabe von CaCl2 . durchgeführt , NaCl, KCl und FeCl3 in 0,05 M Na-Phosphatpuffer (pH 7,0) vor der DNA-Hybridisierungsreaktion und der DPV-Messung. Die Ionenstärke des Hybridisierungspuffers wurde durch Variieren der Na-Phosphat-Puffer- bzw. NaCl-Konzentrationen von 0,002–0,1000 M bis 1,52–5,50 M optimiert. Die lineare Kalibrierungskurve des arowana DNA-Biosensors wurde dann durch quantitative Messung einer Reihe von cDNA-Konzentrationen von 1,0 × 10 –18 . erstellt bis 2,0 × 10 −2 μM über das DPV-Verfahren. Alle Experimente wurden dreifach durchgeführt.
DNA-Extraktion und Arowana-DNA-Analyse
Insgesamt 15 Gewebeproben von Arowana-Fischen wurden freundlicherweise vom Fisheries Research Institute (FRI), Department of Fisheries Malaysia, zur Verfügung gestellt. Alle Fischgewebeproben wurden in 70 % Ethanol in einem Kühler bei 4 °C gelagert und an das Labor versandt. Die Fischgewebeproben wurden mit Milli-Q-Wasser gewaschen und in kleine Stücke geschnitten und bei Umgebungsbedingungen getrocknet, bevor sie bei -20 °C im Gefrierschrank aufbewahrt wurden. Arowana-DNA aus jeder Gewebeprobe (jeweils 35–40 mg) wurde dann separat mit dem QIAquick PCR Purification Kit (Manchester, UK) gemäß dem Protokoll des Herstellers extrahiert und bei –20 °C gelagert, wenn sie nicht verwendet wurde. Die PCR-Amplifikation des genomischen DNA-Fragments wurde dann unter Verwendung eines Bio-Rad-PCR-Thermocyclers (PTC-100, Hercules, USA) durchgeführt. Die DNA-Fragmente des PCR-Produkts wurden dann mit 1,5% Agarosegelelektrophorese aufgetrennt. Die arowana-DNA-Extrakte wurden auch mit dem elektrochemischen DNA-Biosensor analysiert, um das Geschlecht zu bestimmen. Die erhaltenen DPV-Antworten wurden mit dem Grundlinienstrom verglichen, der ohne das Vorhandensein von arowana-DNA erhalten wurde. A t Es wurde ein Test durchgeführt, um einen signifikanten Unterschied zwischen der DNA-Biosensor-Antwort und dem Grundlinienstrom bei 4 Freiheitsgraden und einem Konfidenzniveau von 95 % zu bestimmen. Die DNA-Biosensor-Antwort, die signifikant höher als der Grundlinienstrom erhalten wurde, zeigte an, dass ein männlicher Arowana-Fisch nachgewiesen wurde und umgekehrt.
Ergebnisse und Diskussion
Die so synthetisierten AcMPs wurden unter dem Rasterelektronenmikroskop (REM, LEO 1450VP) beobachtet (Fig. 2). Die Größenverteilung von Acryl-Fehlkristallen, die durch Photopolymerisation hergestellt wurden, ist in Abb. 3 dargestellt.

REM-Aufnahme von Acrylpolymer-Mikrokügelchen
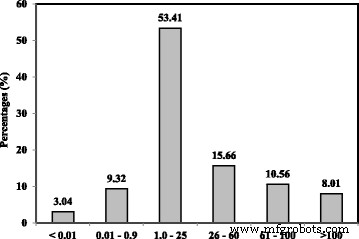
Größenverteilung von Acryl-Mikrosphären, die durch Photopolymerisation hergestellt wurden
Die Wirkung der unterschiedlichen Scan-Raten der Kohlenstoff-SPE mit AcMPs-AuNPs in Gegenwart von K3 Fe(CN)6 zeigten, dass die Oxidations- und Reduktionsspitzenströme mit zunehmender Abtastrate von 0,05 auf 0,30 V/s zunahmen (Abb. 4). Daher wird erwartet, dass der Elektronentransferprozess an der Elektrodenoberfläche reversibel ist [22,23,24,25].
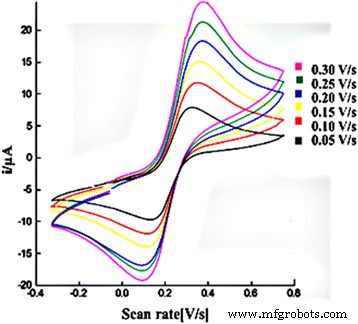
Zyklische Voltammogramme von 1,0 mM K3 Fe(CN)6 in 0,05 M Na-Phosphat-Puffer von pH 7,0 mit unterschiedlichen Scanraten (0,05, 0,10, 0,15, 0,20, 0,25 und 0,30 V/s) für ein modifiziertes Kohlenstoff-SPE mit AcMP-AuNP-Material an der Elektrodenoberfläche
Basierend auf der Randles-Sevcik-Gleichung
$$ \mathrm{ip}=0.4463\ \mathrm{nFAC}\ {\left(\mathrm{nFvD}/\mathrm{RT}\right)}^{1/2} $$ (1)es wurde eine gute Linearität zwischen dem Redox-Spitzenstrom und der Quadratwurzel der Abtastrate mit einem Korrelationskoeffizienten (R 2 ) von 0,996 im Bereich von 50–300 mV/s gemäß Gl. 2 und Abb. 5a.
$$ \mathrm{ip}=1.463{\mathrm{v}}^{1/2\hbox{--} }2.451 $$ (2)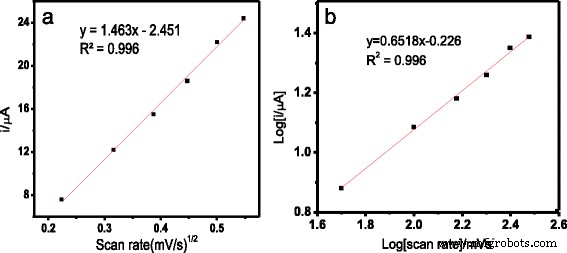
Auftragung der Oxidationsspitzenströme (ip/μA) gegen die Quadratwurzel der Abtastrate ((mV/s) 1/2 ) (a ) und Auftrag des Logarithmus der Oxidationsspitzenströme (ip/μA) gegen den Logarithmus der Scanraten (log (mV/s)) (b )
Dies weist darauf hin, dass die Reaktion an der Oberfläche der modifizierten Elektrode eine diffusionskontrollierte Reaktion war [22,23,24,25].
Außerdem wurde basierend auf Fig. 5b, wenn der logarithmische Wert des Oxidationsstroms gegen den logarithmischen Wert der Abtastrate aufgetragen wurde, eine lineare Linie mit einer Steigung von 0,65 erhalten, was nahe dem theoretischen Wert von 0,50 für diffusionskontrollierte Prozesse lag . Daher hat die Studie gezeigt, dass die Reaktion an der Oberfläche des modifizierten SPE größtenteils diffusionskontrolliert ist.
Für den Idealfall eines schnellen, reversiblen Ein-Elektronen-Transferprozesses beträgt ΔEp =0,059 V bei 298 K. Die mit der Abtastrate zunehmenden Peakpotentialverschiebungen zeigten jedoch größere Peakpotentialabstände von mehr als 0,059 V (Abb. 4 ). Dies impliziert, dass der Elektronentransferprozess an der Elektrodenoberfläche langsam ist [22, 25, 26], wahrscheinlich aufgrund des Widerstands, der durch das Vorhandensein von AcMP-Material erzeugt wird, das die Elektrodenoberfläche bedeckt.
Abbildung 6 zeigt die DPV-Reaktion des arowana-DNA-Biosensors basierend auf AcMP-, AuNP- und AcMP-AuNP-modifizierten Kohlenstoff-SPEs. Der zwischen Experiment (a) und (c) beobachtete signifikante DPV-Stromunterschied zeigt, dass die arowana-DNA-Sonden erfolgreich über starke kovalente Bindungen zwischen der funktionellen Succinimid-Gruppe von AcMP und der funktionellen Amin-Gruppe der aminierten DNA-Sonde auf die AcMPs gepfropft wurden und die immobilisierten Die arowana-DNA-Sonde war nur für ihre cDNA selektiv [19, 20]. Die AuNPs spielten eine Rolle bei der Unterstützung der Elektronenleitfähigkeit vom interkalierten AQMS zur hergestellten Elektrodenoberfläche. Ohne den Einschluss von AuNPs in das Kompositmaterial (f), nur Gold-Nanopartikel (e) und die Gold-Nanopartikel und AcMP-Komposit (d), kann nur eine sehr geringe Stromantwort beobachtet werden. Die niedrigen DPV-Ströme, die in Experiment (b) erworben wurden, waren darauf zurückzuführen, dass keine DNA-Hybridisierungsreaktion mit ncDNA auftrat, was auch auf keine spezifischen Absorptionen des AQMS-Redoxindikators auf der Elektrodenoberfläche hinweist [27, 28].
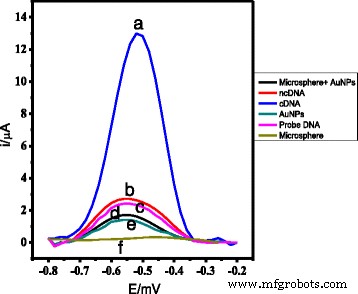
Das DPV-Signal einer AcMP-AuNP-basierten DNA-Elektrode nach Hybridisierung mit cDNA (a ) und nicht komplementäre DNA (b ), die DPV-Antwort der AcMPs (f ) und AuNP-modifizierte SPE (e ) und AcMP-AuNP-Komposit-modifizierter SPE sowie die Reaktion des DNA-Biosensors basierend auf AcMP-AuNP-Komposit-modifizierter Sonden-DNA-SPE (c ) vor der Reaktion mit cDNA in Gegenwart von 1 mM AQMS bei einer Scanrate von 0,5 V/s gegen Ag/AgCl-Referenzelektrode
Für die Dauer der DNA-Sonden-Immobilisierung zeigt Abb. 7a, dass die DNA-Biosensor-Antwort während der ersten 1–3 Stunden der DNA-Sonden-Immobilisierungsdauer langsam ansteigt, und der abrupte Anstieg der DNA-Biosensor-Antwort kann zwischen 3 und 6 Stunden der DNA-Sonden-Immobilisierungsdauer beobachtet werden . Dies lag daran, dass eine längere Immobilisierungszeit erforderlich war, um das Anbringen einer größeren Menge von DNA-Sonden an der AcMP-AuNP-modifizierten Elektrode zu fördern. Bei einer weiteren Verlängerung der DNA-Sonden-Immobilisierungszeit wurde keine merkliche Änderung der DNA-Biosensor-Antwort wahrgenommen, da die Bindungsstellen der immobilisierten AcMPs vollständig an DNA-Sonden gebunden haben. Die Antwort des arowana DNA-Biosensors hängt auch von der DNA-Hybridisierungszeit ab. Das in Fig. 7b dargestellte Biosensor-Antwortprofil zeigt einen zunehmenden aktuellen DPV-Antworttrend mit einer DNA-Hybridisierungsdauer von 10 bis 60 min, wonach die aktuelle Antwort fast ein Plateau erreicht. In diesem Stadium haben die immobilisierten DNA-Sonden auf der Elektrode vollständig mit cDNA hybridisiert [29].
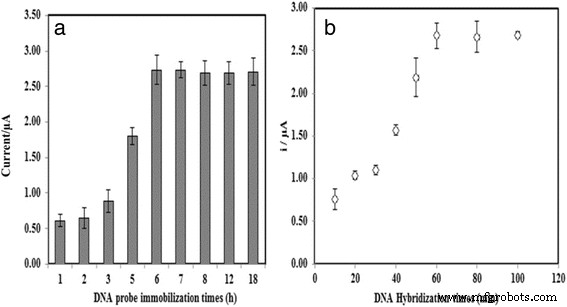
Auswirkungen der Immobilisierungszeit der DNA-Sonde (a ) und DNA-Hybridisierungszeit (b ) auf die Antwort des arowana DNA-Biosensors unter Verwendung von 5 μM DNA-Sonde und cDNA in Gegenwart von 1 mM AQMS bei 2 M Ionenstärke
Es ist auch zu bemerken, dass die DNA-Hybridisierungszeit des hergestellten arowana DNA-Biosensors temperaturabhängig war, und als großer Vorteil erhielten wir eine maximale Stromantwort bei Raumtemperatur innerhalb von 30 Minuten (Abb. 8). Bei niedriger Temperatur, d. h. 4 °C, war eine lange Zeit für eine vollständige DNA-Hybridisierungsreaktion erforderlich, da die Kälte die Reaktionsgeschwindigkeit der DNA-Hybridisierungsreaktion verlangsamte. Eine schnellere DNA-Hybridisierungszeit konnte bei einer Temperatur über 25 °C erreicht werden, was auf die höhere DNA-Hybridisierungsreaktionsrate zurückzuführen ist, die zwischen der immobilisierten DNA-Sonde und der cDNA auftrat, um die Duplex-DNA bei hohen Temperaturen zu bilden. Eine hohe Temperatur könnte jedoch die doppelhelikale Struktur der DNA dauerhaft verformen, und eine Regeneration des DNA-Moleküls ist auch nach der Neueinstellung der Temperatur auf den optimalen Wert nicht möglich [28, 30].
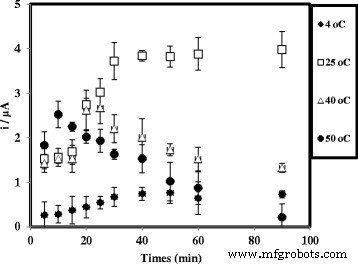
Einfluss der Temperatur auf die DNA-Hybridisierungszeit des arowana DNA-Biosensors. Die DPV-Reaktion wurde in 0,05 M K-Phosphat-Puffer (pH 7,0) bei 4, 25, 40 und 50 °C über einen Versuchszeitraum von 5–90 Minuten gemessen
Als Teil der Reaktionsoptimierung des arowana DNA-Biosensors wurde der Einfluss des pH-Werts der Lösung auf die DNA-Hybridisierungsreaktion untersucht. Der DNA-Biosensor zeigte aufgrund der Protonierung des Phosphodiester-Rückgrats der DNA eine vernachlässigbare Stromänderung zwischen pH 5,5 und pH 6,5, was die Löslichkeit von DNA-Molekülen in wässriger Umgebung verringerte (Abb. 9). Bei weiterem Anstieg des pH-Werts des DNA-Hybridisierungsmediums stieg die Antwort des arowana DNA-Biosensors bei pH 7,0 abrupt an, wonach ein starker Abfall des DPV-Stroms erkennbar war, da sich die pH-Umgebung aufgrund der irreversiblen Denaturierung der DNA bei höheren pH-Werten in einen basischen Zustand änderte Bereich [23, 24, 31,32,33]. Da die maximale DPV-Reaktion bei einem neutralen pH-Wert erreicht wurde, wurde die nächste elektrochemische Bewertung der arowana-DNA-Biosensor-Reaktion bei pH 7,0 unter Verwendung von 0,05 M Na-Phosphat-Puffer gehalten.
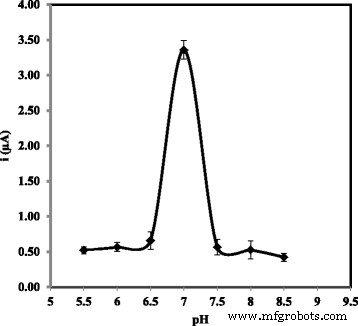
Die DPV-Reaktion des arowana DNA-Biosensors basierend auf AcMP-AuNP-kompositmodifiziertem Kohlenstoff-SPE zwischen pH 5,5 und pH 8,0. Die DPV-Messung wurde in 0,05 M K-Phosphat-Puffer (pH 7,0) bei 25 °C und einer Scanrate von 0,5 V/s gegenüber der Ag/AgCl-Referenzelektrode durchgeführt
Die Wirkung der Wertigkeit von Kationen auf die DNA-Hybridisierungsreaktion wurde unter Verwendung verschiedener Salzkationen, z. Ca 2+ , Na + , K + , und Fe 3+ Ionen im DNA-Hybridisierungspuffer. Die positiv geladenen Ionen könnten elektrostatisch mit der negativ geladenen Phosphodiesterkette des DNA-Moleküls wechselwirken, um die sterische Hinderung und elektrostatische Abstoßung zwischen der immobilisierten DNA-Sonde und der Ziel-DNA zu überwinden, wodurch der DNA-Hybridisierungsprozess erleichtert wird [34]. Abbildung 10 zeigt, dass die DNA-Hybridisierungsreaktion in Gegenwart von Kationen in der Reihenfolge Na + . günstig war> K + > Fe 3+ > Ca 2+ . Das Vorhandensein von Ca 2+ und Fe 3+ Es wurde festgestellt, dass Ionen eine bemerkenswerte Abnahme der Stromantwort des arowana-DNA-Biosensors im Vergleich zu Na + . verursachen und K + Ionen. Diese Phänomene wurden auf die Bildung schwerlöslicher Calciumphosphat- und Ferrum(III)-Phosphatsalze im DNA-Hybridisierungspuffer zurückgeführt [22], die den Ionengehalt der Lösung reduzierten und eine starke elektrostatische Abstoßung zwischen den DNA-Molekülen verursachten. Als Ergebnis wurde die DNA-Hybridisierungsrate verringert und führte zu einer schlechten Biosensorleistung. Die höchste DNA-Biosensor-Antwort wurde erhalten, wenn Na + Ionen wurden dem DNA-Hybridisierungs-Phosphatpuffer wegen ihrer geringen Größe und ihrer starken Affinität zur DNA-Phosphodieter-Bindung zugesetzt.
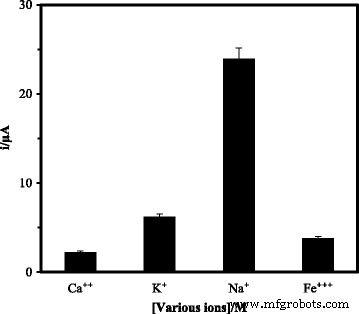
Die Wirkung von Ca 2+ , Na + , K + , und Fe 3+ Ionen im DNA-Hybridisierungspuffer (0,05 M Na-Phosphat-Puffer bei pH 7,0) auf die DPV-Antwort des arowana DNA-Biosensors
Die Konzentration von NaCl und Na-Phosphatpuffer (pH 7,0) muss ebenfalls optimiert werden, um eine optimale Ionenstärke für den Hybridisierungspuffer bereitzustellen. Abbildung 11b zeigt, dass eine Ionenstärke von unter und über 2 M die hohe elektrostatische Abstoßung zwischen DNA-Strängen nicht überwinden konnte. Es wurde gefunden, dass etwa 0,05 M Na-Phosphat-Puffer ( 11a ) und 2 M NaCl die optimale Ionenstärke für den Assay von Arowana-Ziel-DNA mit maximaler Biosensorleistung bereitstellen. Optimale Hybridisierungspufferbedingungen in Bezug auf pH, Pufferkapazität und Ionenstärke würden es ermöglichen, dass die DNA-Hybridisierungsreaktion bei minimalster sterischer Hinderung abläuft [30].
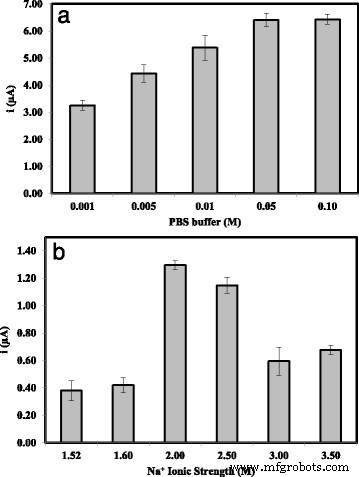
Die Antworttrends des arowana-DNA-Biosensors als a Na-Phosphat-Pufferkonzentration und b Ionenstärke des Hybridisierungspuffers variierte von 0,002–0,100 M bzw. 1,52–5,50 M
Der optimierte DNA-Biosensor wurde dann zum Nachweis einer Reihe von arowana-cDNA-Konzentrationen zwischen 1,0 × 10 –12 . verwendet und 1,0 × 10 –2 μM. Der DNA-Biosensor zeigte einen breiten linearen Ansprechbereich von 1,0 × 10 –18 bis 1,0 × 10 –8 M (R 2 =0,99). Die Nachweisgrenze (LOD) liegt bei 1,0 × 10 –18 M wurde auf der Grundlage der dreifachen Standardabweichung der Biosensor-Antwort bei der Ansprechkurve berechnet, die sich der LOD annähert, dividiert durch die lineare Kalibrierungssteigung. Die homogene AcMP-Partikelgröße im Mikrometerbereich zeigte einen signifikanten Einfluss auf die Empfindlichkeit und Reproduzierbarkeit des DNA-Biosensors (RSD =5,6 %). The large binding surface area of the immobilised NAS-functionalised AcMPs permitted a large number of DNA molecules to bind covalently to the electrode surface, thereby increasing the DNA biosensor analytical performance with respect to dynamic linear range and detection limit of the arowana DNA biosensor (Fig. 12).
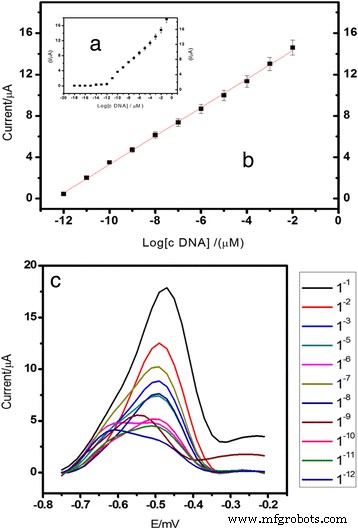
The arowana DNA biosensor response curve (a ) and linear calibration range (b ) and the DPV voltammogram (c ) obtained using 1.0 × 10 −18 to 1.0 × 10 −2 μM cDNA at pH 7.0
Determination of Arowana Fish Gender with DNA Biosensor
The developed electrochemical DNA biosensor has been validated with the standard PCR-based method to determine the gender of Asian arowana fish. With the results tabulated in Table 2, both methods provided the same result for the gender determination of arowana fish. This indicates that the proposed DNA biosensor can be used for accurate determination of arowana gender in a simple and fast way.
Conclusions
The electrochemical DNA biosensor developed in this study demonstrated good sensitivity, wide linear response ranges, and low detection limit in the determination of arowana target DNA. In addition, the DNA biosensor showed a good response towards arowana cDNA, which implies that the electrochemical DNA biosensor could be used to successfully detect the arowana DNA segments. The developed arowana DNA biosensor can be further redesigned into a point-of-use device prototype that offers a great potential for the application in the fish culture for early identification of arowana gender and colour, which is economically advantageous in fishery and aquaculture sectors.
Nanomaterialien
- 6 Tipps zur optimalen Nutzung Ihres PM-Programms
- IBM &Warwick stellen erstmals hochreaktives dreieckiges Molekül dar
- Demonstration eines flexiblen Graphen-basierten Biosensors für den empfindlichen und schnellen Nachweis von Eierstockkrebszellen
- N,N-Dimethylformamid, das die Fluoreszenz von MXen-Quantenpunkten für die empfindliche Bestimmung von Fe3+ reguliert
- Ultrasensitiver Biosensor zum Nachweis von Vibrio cholerae-DNA mit Polystyrol-Co-Acrylsäure-Komposit-Nanosphären
- Funktionelles poröses Polymer auf Triphenylphosphin-Basis als effizienter heterogener Katalysator für die Synthese cyclischer Carbonate aus CO2
- Entwicklung der Kontaktfläche bei Normallast für raue Oberflächen:von atomaren zu makroskopischen Skalen
- Auswirkung verschiedener Bindemittel auf die elektrochemische Leistung einer Metalloxidanode für Lithium-Ionen-Batterien
- Lektionen vom Militär zur Verbesserung des Kundenerlebnisses im Einzelhandel
- Tipps zum Schutz von Baumaschinen vor Sommerhitze



