Silica-Nanopartikel für die intrazelluläre Proteinzufuhr:ein neuartiger Syntheseansatz unter Verwendung von grün fluoreszierendem Protein
Zusammenfassung
In dieser Studie wird ein neuartiger Ansatz zur Herstellung von mit grünem Fluoreszenzprotein (GFP) dotierten Siliziumdioxid-Nanopartikeln mit einer engen Größenverteilung vorgestellt. GFP wurde aufgrund seiner Autofluoreszenz als Modellprotein ausgewählt. Proteindotierte Nanopartikel haben ein hohes Anwendungspotenzial im Bereich der intrazellulären Proteinabgabe. Darüber hinaus können fluoreszenzmarkierte Partikel für das Bioimaging verwendet werden. Die Größe dieser proteindotierten Nanopartikel wurde unter Verwendung eines mehrstufigen Syntheseprozesses von 15 bis 35 nm eingestellt, der die Partikelkernsynthese gefolgt von Schritten zum Nachwachsen der Schale umfasste. GFP wurde durch eine Eintopfreaktion selektiv in die Silicamatrix des Kerns oder der Hülle oder in beide eingebaut. Die erhaltenen Nanopartikel wurden durch Bestimmung von Partikelgröße, hydrodynamischem Durchmesser, ζ-Potential, Fluoreszenz und Quantenausbeute charakterisiert. Die Messungen zeigten, dass die Fluoreszenz von GFP während der Partikelsynthese erhalten blieb. Zelluläre Aufnahmeexperimente zeigten, dass die GFP-dotierten Nanopartikel als stabile und effektive Fluoreszenzsonden verwendet werden können. Die Studie zeigt das Potenzial des gewählten Ansatzes für den Einbau funktioneller biologischer Makromoleküle in Siliziumdioxid-Nanopartikel, was neue Anwendungsfelder wie die intrazelluläre Proteinzufuhr eröffnet.
Hintergrund
In den letzten Jahren hat die Verkapselung von Proteinen in Mikro- und Nanopartikel aufgrund des breiten Anwendungspotenzials von Materialien wie Biosensoren [1] oder Bioreaktoren [2] sowie auf dem Gebiet der kontrollierten Proteinabgabe [3] große Aufmerksamkeit erlangt. intrazellulärer Proteintransport [4] und Tissue Engineering [5]. Bei vielen dieser Anwendungen ist die katalytische Aktivität verkapselter Enzyme eine Grundfunktion solcher Materialien. Dagegen üben pharmazeutisch relevante Proteine, Peptidhormone oder Antikörper als potenzielle Fracht solcher Nanomaterialien ihre Funktion durch die spezifische Bindung von Targets innerhalb von Geweben oder Zellen aus. Eine Voraussetzung all dieser Anwendungen ist daher die Aufrechterhaltung einer intakten Konformation und Funktionalität der Cargo-Proteine. Nanostrukturierte Systeme haben sich aufgrund ihrer geringen Größe, großen spezifischen Oberfläche und anderer einzigartiger Eigenschaften zu einem der sich am schnellsten entwickelnden Gebiete in der biomedizinischen Forschung entwickelt [6]. Daher ist die Entwicklung neuer Partikelträger zur Verbesserung der Funktionalität und Stabilität der entworfenen Systeme ein wichtiges Thema auf diesem Gebiet [7]. Die Matrix nanopartikulärer Träger kann auf biomakromolekularen oder organischen Komponenten wie Kohlenhydraten, Lipiden oder Polymeren basieren und Systeme wie Fest-Lipid-Nanopartikel, Liposomen oder Dendrimere bilden. Darüber hinaus können nanostrukturierte Systeme auch auf anorganischen Materialien wie Metallen oder Oxiden basieren [8]. Alle diese Materialsysteme müssen verschiedene gängige sowie spezifische Anforderungen erfüllen. Zunächst müssen die Matrixmaterialien biokompatibel sein, um sichere Anwendungen zu ermöglichen [9]. Zum anderen müssen sie stabil genug sein, um ihre Funktion als Trägermaterial über den Lebenszyklus der Systeme zu erfüllen. Darüber hinaus müssen sie die Kapazität für eine signifikante Proteinaufnahme und -retention sowie für eine kontrollierte Proteinfreisetzung bereitstellen [10].
Neben der Anlagerung von Proteinen an die Oberfläche von Nanoobjekten durch Adsorption oder kovalente Bindung [11] können Proteine in Nanostrukturen eingeschlossen werden, wodurch deren Stabilität und enzymatische Aktivität erhöht wird [2]. Ein Nanoeinschluss kann durch Hydrolyse und Kondensation eines Silica-Vorläufers über Sol-Gel-Prozesse [12] oder über Wasser-in-Öl-Mikroemulsionsansätze erreicht werden, was eine Polymerisation des die Hülle umgebenden Enzyms an der Wasser-Öl-Grenzfläche verursacht [13]. Bei diesen Methoden kann das Einschließen von Proteinen durch zwei verschiedene chemische Ansätze erfolgen, wobei kovalente oder nicht-kovalente Bindungsprozesse verwendet werden [14]. Insbesondere amorphes Siliziumdioxid ist aufgrund seiner hohen Biokompatibilität, Inertheit und mechanischen Stabilität ein vielversprechendes Trägermaterial für Proteine [15]. Es wurden verschiedene Wege, insbesondere biomimetische Ansätze zur Enzymverkapselung in Siliziumdioxid verfolgt [2, 16], wobei das Freisetzungsprofil der Enzyme durch chemische Reaktionen des Linkers oder den Abbau der Siliziumdioxidmatrix gesteuert wird. Mesoporöse Materialien wurden auch als Matrix verwendet, um Enzyme in Poren von 2–50 nm zu immobilisieren [13, 17]. Die Ladungsfreisetzung aus mesoporösen Nanopartikeln kann durch Anwendung der „Gatekeeper“-Strategie oder durch Modifizieren der inneren Oberfläche der Poren angepasst werden, um die Bindungsaffinität mit Wirkstoffen zu kontrollieren [10b]. Dennoch kann die Porengröße die Beladung der mesoporösen Silicagerüste mit Enzymen begrenzen [18], weshalb neuerdings neue Strategien für den Proteintransport untersucht werden.
Da Silica-Nanopartikel weit verbreitet für die Biobildgebung verwendet werden [19], stellt der Einbau fluoreszierender Proteine eine Option zur Herstellung biokompatibler Fluoreszenzsonden dar. In der Literatur wurde beispielsweise der Einbau von grün fluoreszierendem Protein (GFP) in Silica-Nanopartikel über reverse Emulsionstechniken beschrieben [20]. Diese Studien zeigen, dass der Einbau von GFP in die Silica-Partikelmatrix nicht nur die Fluoreszenzintensität des Proteins erhöht, sondern auch seine thermische Stabilität, Stabilität gegenüber chemischer Denaturierung und Protease-Behandlung. Für die Synthese wohldefinierter Silica-Nanopartikel im unteren nanoskaligen Bereich mit enger Größenverteilung ist die Methode jedoch weniger geeignet. Darüber hinaus umfassen die Synthesebedingungen den Kontakt mit Tensiden, Alkoholen oder stark alkalischen Basen sowie hohe Temperaturen, die alle möglicherweise nicht mit dem Einbau anfälliger Proteine vereinbar sind [20, 21].
Daher berichten wir über einen neuen Ansatz zur Herstellung von proteindotierten Silica-Nanopartikeln unter Verwendung von GFP als Modellprotein. Zu diesem Zweck verwendeten wir eine Eintopfsynthese unter milden Synthesebedingungen (Raumtemperatur, niedriger Salzgehalt) gefolgt von Dialyseschritten zur Reinigung. Der Ansatz zeichnet sich durch sein Potenzial zur Herstellung von in Proteinen eingeschlossenen Siliziumdioxid-Nanopartikeln aus, die eine enge Größenverteilung im Größenbereich unter 50 nm aufweisen.
Methoden
Materialien
Alle Chemikalien wurden wie von Sigma-Aldrich (Taufkirchen, Deutschland) bezogen und ohne weitere Reinigung verwendet. Für alle Synthese- und Reinigungsschritte wurde Reinstwasser (18,2 MΩ, Milli-Q-Wasserreinigungssystem Typ ELIX 20, Millipore Corp., USA) verwendet.
Vorbereitung des GFP
GFP wurde durch Proteinexpression und anschließende Reinigung wie an anderer Stelle beschrieben erhalten [22]. Kurz gesagt wurde GFP mit einem N-terminalen His6-Tag unter Verwendung eines hochgradigen bakteriellen Expressionsvektors basierend auf dem pQE-Vektorsystem (Qiagen, Hilden, Deutschland) in E. coli XL1-Blue und gereinigt durch Ni-geladene Affinitätschromatographie (Qiagen, Hilden, Deutschland). Anschließend wurde das Protein zum Pufferaustausch in eine Konzentratorvorrichtung (3 kDa Molekulargewichts-Cut-off (MWCO)-Membran, Pall, Dreieich, Deutschland) überführt. GFP wurde dreimal durch Zugabe von 15 ml -Arginin- bzw. Natriumbicarbonatlösung gewaschen und anschließend in 3 ml der -Arginin/Natriumbicarbonat-Lösung gewonnen. Danach wurde die GFP-Suspension durch sterile 0,22 µm Celluloseacetatfilter (Carl Roth, Karlsruhe, Deutschland) in sterile Röhrchen filtriert. Vor der Verwendung wurde die Proteinkonzentration auf 1 mg ml –1 . eingestellt entweder in 7,2 mmol L −1 ʟ-Arginin (pH = 10,3) oder 10,0 mmol L −1 NaHCO3 (pH = 9.2) Lösung.
Synthesen und Reinigung von Nanopartikeln
Silica-Nanopartikel wurden gemäß einem zuvor beschriebenen modifizierten Protokoll hergestellt [23]. Kurz gesagt wurde Tetraethoxysilan (TEOS), das als unpolare Vorstufe verwendet wurde, in einem zweiphasigen Wasser/Cyclohexan-System, vermittelt durch ʟ-Arginin-Katalyse, hydrolysiert.
Vorbereitung der Kernpartikel
In einem Dreihalsrundkolben wurden 91 mg (0,52 mmol) ʟ-Arginin in 69 ml Wasser gelöst, bevor 4,5 ml Cyclohexan als Deckschicht zugegeben wurden. Die Reaktionsmischung wurde unter Rühren auf 40 °C erhitzt. Nach Zugabe von 5,5 ml (24,63 mmol) TEOS wurde die Mischung weitere 20 h unter diesen Bedingungen gehalten.
Silica Shell Layers
Für nachfolgende Schalenwachstumsschritte wurden entweder die Kernpartikel oder die aus dem ersten Schalenwachstumsschritt resultierenden Partikel verwendet. Für das Schalenwachstum wurden 14 mg (0,08 mmol) ʟ-Arginin in 36 ml Wasser gelöst und 10 ml zuvor hergestellter Partikeldispersionen zugegeben. Nach Zugabe von 5 ml Cyclohexan wurde die Mischung auf 40 °C erhitzt. Nach Zugabe von 3,52 ml (15,8 mmol) TEOS wurde die Mischung weitere 20 h gerührt.
Herstellung von GFP-dotierten Nanopartikeln. Zur Herstellung von GFP-dotierten Nanopartikeln wurden 30 Minuten nach der Zugabe von TEOS 200 μg (6,9 nmol) GFP zugegeben.
Partikelreinigung
Die Nanopartikel wurden durch anschließende Dialyse gegen Wasser (4 L, Wasseraustausch nach 30, 90 und 180 min) für 4 h unter Verwendung einer Cellulosehydratmembran (Nadir-Dialyseschlauch, MWCO 10 kDa, Carl Roth, Karlsruhe, Deutschland) gereinigt. Schließlich wurden die Nanopartikel mit sterilen 0,22 µm Celluloseacetat-Membranfiltern (Carl Roth, Karlsruhe, Deutschland) in sterile Flaschen filtriert.
Transmissionselektronenmikroskopie (TEM)
Morphologie und mittlerer Teilchendurchmesser wurden unter Verwendung eines JEM-2100F-Mikroskops (JEOL, Freising, Deutschland) bestimmt. Die Partikelgrößenverteilung wurde an einer Stichprobe von 50 Nanopartikeln mit der Software X-ImageJ (Version:1.45 s, winPenPack X-ImageJ Launcher des National Institute of Health (http://rsb.info.nih.gov/ij .) bestimmt /).
Hydrodynamischer Durchmesser
Der hydrodynamische Durchmesser der Nanopartikel wurde mit einem Zetasizer Nano ZSP (Malvern Instruments, Herrenberg, Deutschland) aufgezeichnet. Vor den Messungen wurden Partikeldispersionen 1:10 in Wasser verdünnt. Die Messungen wurden bei 25 °C durchgeführt. Jede Probe wurde 3 × 15 mal gemessen. Der Durchmesser wurde durch Berechnung der Volumenverteilung bestimmt. Dies wurde aus der Intensitätsgrößenverteilung unter Verwendung der Mie-Theorie umgerechnet.
ζ-Potenzial
Das ζ-Potential wurde unter Verwendung desselben Instruments unter den oben beschriebenen Bedingungen gemessen, außer dass die Proben in 0,01 M KCl (9:1) verdünnt wurden.
Analytische Ultrazentrifugation (AUC)
Zur Messung der Sedimentationsgeschwindigkeit wurde ein modifizierter Beckman-Coulter XL-80 K mit einem AnTi60-Rotor verwendet. Für die Experimente wurde die Temperatur auf 20 °C eingestellt, die Geschwindigkeit auf 10.000 U/min eingestellt und es wurden 21 Scans durchgeführt. Die Wellenlängen wurden auf 261 nm für Siliciumdioxid und 488 nm für die GFP-Erkennung eingestellt.
Fluoreszenzspektroskopie
Fluoreszenzspektren von Nanopartikeln, reinem GFP und Filtraten aus Auslaugungsexperimenten wurden mit dem Fluoromax-3 Spektrofluorometer (Spex, Horiba Scientific, Oberursel, Deutschland) aufgenommen. Für Messungen wurden reines GFP, Partikeldispersionen und Filtrat 1:10 in Wasser verdünnt. Die Anregungswellenlänge wurde auf 488 nm eingestellt und das Spektrum wurde in einem Spektralbereich von 498 bis 800 nm aufgenommen.
Fluoreszenz-Quantenausbeuten
Die Quantenausbeuten der erhaltenen Nanopartikel und des reinen GFP wurden mit der relativen Methode von Williamson et al. [24]. Als Referenz für GFP wurden Rhodamin 6G und Atto488 verwendet. Vergleichsmessungen wurden mit undotierten Nanopartikeln durchgeführt, die mit dem Referenzfarbstoff vermischt wurden. Fluoreszenzspektren wurden mit einer Anregungswellenlänge von 450 nm aufgenommen. Zusätzliche UV/vis-Messungen wurden mit einem Varian Cary 300 Scan UV (Agilent Technologies, Darmstadt, Deutschland) durchgeführt.
Zur Berechnung der Quantenausbeute gilt Gl. 2 wurde verwendet.
$$ {\varPhi}_P={\varPhi}_S\bullet \frac{{\mathrm{Steigung}}_S}{{\mathrm{Steigung}}_P}\bullet {\left(\frac{n_P}{n_S }\right)}^2 $$ (2)Hier, φ P ist die Quantenausbeute des Produkts, φ S die Quantenausbeute der Referenz. Die Begriffe SteigungS und SteigungP stellen die Steigungen dar, die aus den Kurven der integrierten Fluoreszenzintensität gegenüber der Absorption von Referenz bzw. Produkt abgeleitet wurden. n P und n S entsprechen dem Brechungsindex des verwendeten Lösungsmittels [25].
Proteinleckage
Für Auslaugversuche wurden unverdünnte Partikeldispersionen durch modifizierte Polyethersulfonmembranen (MWCO = 100 kDa oder 300 kDa, Pall, Dreieich, Deutschland) durch Zentrifugation (16.000 g, 5 min) ultrafiltriert.
Thermische Stabilität
Zur Analyse der thermischen Stabilität wurden Nanopartikel und reines GFP 0 und 24 h bei 20 oder 60 °C aufbewahrt. Nanopartikel und reines GFP wurden wie oben beschrieben verdünnt.
Photobleaching
Um die Stabilität von GFP-dotierten Nanopartikeln und reinem GFP gegen Photobleichen zu untersuchen, wurden die Lösungen über einen Zeitraum von bis zu 20 Minuten dem Licht von sieben grünen LEDs ausgesetzt. Die Fluoreszenzintensität von Proben, die bei t . genommen wurden = 0, 2 und 20 Minuten wurden gemessen.
Stabilität gegen Proteinabbau
Um die Stabilität von GFP gegen Proteinase K zu bestimmen, wurden reine GFP, unmarkierte Silica-Nanopartikel (CU S1U S2U ) mit zusätzlichem GFP und dreifach markierten Silica-Nanopartikeln (CF S1F S2F ) wurden in den gleichen GFP-Konzentrationen und der gleichen Partikelmenge verwendet. Alle Proben wurden 1:100 verdünnt. Für die Menge von 10 GFP-Molekülen wurde ein Proteinase-K-Molekül gewählt. Vor der Zugabe des Enzyms wurde eine Probe unter den obigen Bedingungen gemessen. Nach der Zugabe wurden die Messungen nach t . durchgeführt = 0, 15, 30, 45, 60 und 90 Minuten
Zelluläre Aufnahmeexperimente
Um die Internalisierung von Nanopartikeln und GFP durch Zellen zu bestimmen, wurden zelluläre Aufnahmeexperimente mit der Lungenkarzinomzelllinie A549 (ACC-107) durchgeführt.
Kultivierung von Zellen
A549-Zellen (DSMZ, Braunschweig, Deutschland) wurden in T75-Kolben (Greiner bio-one, Frickenhausen, Deutschland) unter Verwendung von Dulbecco's Modified Eagle Medium (DMEM, Thermo-Fisher-Scientific, Waltham, MA, USA) mit 10 % Fetal kultiviert Kalbsserum (FCS). 2 × 10 4 cm −2 A549-Zellen wurden auf Deckgläsern in 12-Well-Platten ausgesät und für 24 h kultiviert. Die Zellen wurden dann 24 h lang mit GFP-dotierten Nanopartikeln und GFP-Lösung in 1 ml Medium behandelt. Das SiO2 Konzentration der Nanopartikel betrug 37 μg mL −1 während die GFP-Konzentration 5 μg ml –1 . betrug sowohl für Nanopartikel als auch für reines GFP. Nach der Behandlung wurden die Zellen zweimal mit phosphatgepufferter Kochsalzlösung (PBS) gewaschen.
Probenvorbereitung und konfokale Bildgebung
Die Zellen wurden mit 4% Paraformaldehyd in PBS für 20 Minuten bei Raumtemperatur fixiert. Zur Färbung der Zellmembran Tetramethylrhodamin-konjugiertes WGA (Weizenkeim-Agglutinin (2 μg mL −1 (in PBS), W849, Thermo-Fisher-Scientific (Invitrogen), Waltham, MA, USA) wurde zugegeben und 10 min bei Raumtemperatur inkubiert. Nach drei Waschschritten mit PBS wurden die Zellen dreimal mit PBS gewaschen und mit Mowiol/DABCO (Carl Roth, Karlsruhe, Deutschland) auf Glasobjektträger montiert.
Konfokale Bilder wurden auf einem TCS SP5-System (Leica, Wetzlar, Deutschland) aufgenommen. Zur Bildgebung ein 63× Öl-Immersions-Objektiv (n =1,518) verwendet wurde. Sequenzielle Scans wurden mit der Argon-Ionen-Laserlinie bei λ . aufgenommen = 488 nm (25 %) für die Anregung von GFP und ein diodengepumpter Festkörperlaser bei λ = 561 nm (25 %) für die Anregung von Tetramethylrhodamin.
Ergebnisse und Diskussion
Diese Studie zielt darauf ab, Silica-Nanopartikel mit GFP unter geeigneten Bedingungen zu funktionalisieren, die die biochemischen Eigenschaften und die Funktionalität des Proteins erhalten. In früheren Arbeiten synthetisierten wir Nah-IR-farbstoffdotierte monodisperse fluoreszierende Siliziumdioxid-Nanopartikel im Größenbereich zwischen 15 und 80 nm unter Verwendung einer ʟ-Arginin-kontrollierten Hydrolyse von Tetraethoxysilan (TEOS) in einem zweiphasigen Cyclohexan/Wasser-System [26]. Hier haben wir dieses Syntheseverfahren übernommen, um GFP als Modellprotein in die Kieselsäurematrix einzubetten. In Schema 1 ist die Vorgehensweise zur Partikelsynthese schematisch dargestellt. GFP-dotierte und nicht-dotierte Strukturen (Kern/Schalen) sind grün bzw. grau hervorgehoben. In einem ersten Schritt werden GFP-dotierte Silica-Kernpartikel (CF ) erhalten. Nachfolgende Nachwachsschritte (CF S1 und CF S1 S2 ) ermöglichte die Synthese größerer Partikelgrößen. Während des ersten Nachwachsschritts wurde die Schale entweder mit (CF S1F ) oder ohne (CF S1U ) Einbau von Protein. Ebenso wird im zweiten Nachwachsschritt entweder ein markiertes (CF S1F S2F , CF S1U S2F ) oder ein unbeschriftetes (CF S1F S2U , CF S1U S2U ) Schale wurde hinzugefügt. Diese Variationen ermöglichen eine hervorragende Kontrolle über die Menge des eingebetteten Proteins und seine maßgeschneiderte Anordnung in bestimmten Schalen oder dem Partikelkern. Darüber hinaus sind reine Siliziumdioxid-Nanopartikel ohne eingebettetes GFP (CU , CU S1U , und CU S1U S2U ) wurden synthetisiert, um einen möglichen Einfluss der Proteineinbettung auf die Partikeleigenschaften zu untersuchen. Außerdem wurde das GFP für all diese Schritte in zwei verschiedenen Puffersystemen (ʟ-Arginin und NaHCO3 .) gelöst ) verschiedener pH-Werte, um den Einfluss des Proteinlösungsmittels auf die Partikelsynthese, die Morphologie, die Fluoreszenzintensität, die Emissionswellenlänge und das ζ-Potential zu bestimmen.
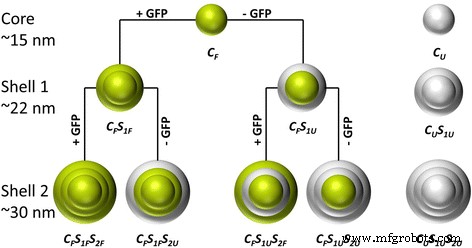
Übersicht über die synthetisierten Partikel und deren Partikelstruktur. Grüne Farbe zeigt die Einbettung von GFP in entweder den Kern bzw. die Hüllen an. Die graue Farbe stellt die Schalen ohne GFP dar (CF = Kern fluoreszierend, CU = Kern ohne Label, SF = Muschel fluoreszierend, SU = Schale ohne Label, S1 = erste Schalenschicht, S2 = zweite Hüllenschicht)
Nanopartikel-Charakterisierung
Bestimmung der physikalischen Partikelattribute
Um die Partikelgröße und Morphologie nach dem Einbau von GFP zu beschreiben und den Einfluss der beiden unterschiedlichen Puffersysteme auf diese Eigenschaften zu bestimmen, wurden TEM-Bilder aufgenommen (Abb. 1). Weitere TEM-Bilder von GFP(NaHCO3 .) ) modifizierte, GFP(ʟ-Arginin) modifizierte und unmarkierte Nanopartikel sind in der SI dargestellt (Zusatzdatei 1:Abbildung S1, Zusatzdatei 2:Abbildung S2, Zusatzdatei 3:Abbildung S3, Zusatzdatei 4:Abbildung S4). Nach dem Syntheseverfahren mit zwei Nachwachsschritten wurden drei verschiedene Partikelgrößen erhalten. Die Kernpartikel hatten eine Größe von ca. 15 nm, die Partikel nach dem ersten Nachwachsschritt ca. 22 nm und die Partikel nach dem zweiten Schritt ca. 32 nm. Zusammenfassend waren alle Nanopartikel annähernd kugelförmig und zeigten eine enge Größenverteilung (p < 10%). Die drei Generationen vollständig gefärbter GFP(ʟ-Arginin)-Nanopartikel (CF , CF S1F , und CF S1F S2F ) und das GFP(NaHCO3 ) (CF ) Kern-Nanopartikel wurden als Modell gewählt.

TEM-Bilder von drei Generationen von GFP-ʟ-Arginin-modifizierten Nanopartikeln und Kernpartikeln von GFP(NaHCO3 ) modifizierte Nanopartikel. In a , c und d , sind die drei Generationen von GFP(ʟ-Arginin) dargestellt:CF Kernpartikel (a , dTEM = 15,5 ± 1,1 nm); CF S1F Nanopartikel nach dem ersten Nachwachsschritt (Kern + Hülle 1) (c , dTEM = 23,5 ± 2,0 nm) und CF S1F S2F nach dem zweiten Nachwachsschritt (Kern + Hülle 1 + Hülle 2) (d , dTEM = 35,3 ± 2,0 nm). In b , das GFP(NaHCO3 )-markierte Kern-Nanopartikel (dTEM = 15,2 ± 1,2 nm) werden angezeigt
Beim Vergleich der Größen der verschiedenen GFP-dotierten und unmarkierten Nanopartikel (Tabelle 1) ist bemerkenswert, dass die gleiche Anzahl von Nachwachsschritten zu der gleichen mittleren Partikelgröße führte, unabhängig von der Anwesenheit von Protein oder der verwendeten Pufferlösung. Die unmarkierten Partikel hatten auch ähnliche Größen (CU :dTEM = 13,4 ± 0,4 nm, dDLS = 10 ± 3 nm; CU S1U :dTEM = 20,9 ± 1,3 nm, dDLS = 20 ± 6 nm; CU S1U S2U :dTEM = 33,2 ± 1,0 nm, dDLS = 38 ± 10 nm).
Zusammenfassend konnte gezeigt werden, dass der Einbau von Protein in die Silica-Matrix und die Pufferlösung, in der das Protein bereitgestellt wurde, keinen signifikanten Einfluss auf die resultierende Partikelgröße und Morphologie hatte.
Nach unserem besten Wissen sind in der Literatur keine anderen in GFP eingebetteten Silica-Nanopartikel beschrieben, die ähnlich kleine Größen sowie ebenso enge Größenverteilungen (< 10 %) aufweisen [20, 27]. Solche kleinen Nanopartikel haben ein vielversprechendes Anwendungspotenzial im Bereich der intrazellulären Proteinzufuhr sowie in der Krebsdiagnose und -therapie [28].
ζ-Potenzial
Das ζ-Potential aller Nanopartikel wurde durch Berechnungen unter Verwendung ihrer elektrophoretischen Mobilität bestimmt. Alle Arten von dotierten Nanopartikeln zeigten ein negatives ζ-Potential mit Absolutwerten im Bereich von − 28 bis − 36 mV (Abb. 2). Im Vergleich dazu zeigt das ζ-Potential von unmarkierten Partikeln sehr ähnliche Werte mit − 35,5 ± 2,0 mV für die Kernpartikel, − 34,0 ± 3,7 mV nach dem ersten Nachwachsen-Schritt und – 34,5 ± 1,2 mV nach dem zweiten Nachwachsen-Schritt. Diese stark negativen ζ-Potentialwerte (< − 28 mV) weisen auf eine hohe Stabilität der Nanopartikel gegen Agglomeration aufgrund elektrostatischer Abstoßung hin. Verglichen mit dem ζ-Potential unmarkierter Nanopartikel zeigen die Daten, dass weder die resultierende Partikelgröße noch der Einbau von GFP in die Partikelmatrix des Partikelkerns oder der Partikelhülle einen signifikanten Einfluss auf die Partikelladung hatte.
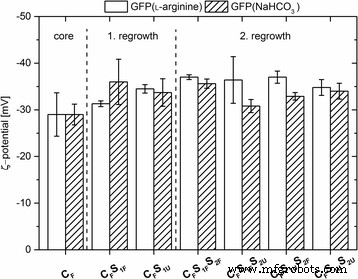
ζ-Potential [mV] markierter Nanopartikel. Die Nanopartikel wurden ausgehend von GFP hergestellt, das entweder in 7,2 mM ʟ-Arginin oder 10 mM NaHCO3 . gelöst war . Fehlerbalken geben die aus drei Messungen abgeleitete Standardabweichung an
Spektroskopiestudien
Fluoreszenzspektroskopie
Alle GFP-dotierten Silica-Nanopartikel zeigten ein ähnliches Emissionsmaximum (λ em = 508 nm), was auch mit dem Emissionsmaximum von freiem GFP vergleichbar war (SI, Additional file 5:Figure S5). Um die Fluoreszenzintensität der verschiedenen markierten Nanopartikel zu vergleichen, wurde die Nanopartikelkonzentration normalisiert (Berechnungen in SI 5.). Wie erwartet führte die schrittweise Zugabe von markierten Schalen zu einer Zunahme der Fluoreszenz der Nanopartikel (Abb. 3).
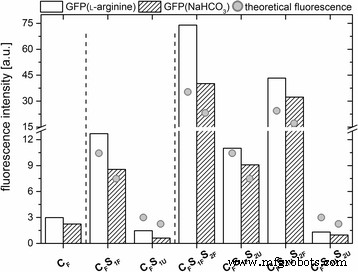
Normalisierte Fluoreszenzintensität des Emissionsmaximums bei 508 nm für jedes der verschiedenen Partikelsysteme. Außerdem ist die theoretische Fluoreszenzintensität (Graupunkte ) der Partikel im Verhältnis zur Zunahme des Partikelvolumens wird gezeigt
Nanopartikel mit nur einem markierten Kern, aber mit undotierten Hüllen zeigten die geringste Fluoreszenz. Nanopartikel mit einer zusätzlichen markierten Hülle zeigten eine mittlere Fluoreszenz, und Nanopartikel mit zwei markierten Hüllen zeigten die stärkste Fluoreszenz (Abb. 3). Bemerkenswerterweise schien das Hinzufügen einer äußeren undotierten Hülle die Fluoreszenz von Nanopartikeln im Vergleich zu Nanopartikeln mit einer dotierten äußeren Schicht leicht zu reduzieren. Dieser Effekt könnte durch Abschirmungseffekte der unmarkierten Kieselsäurehülle hervorgerufen werden. Zusammenfassend führte die Zugabe von GFP-dotierten Hüllen zu Kernpartikeln zu einer Zunahme der Fluoreszenzintensität der resultierenden Nanopartikel, die mit der Volumenänderung korreliert zu sein schien, die das Nanopartikelwachstum begleitet.
Die Einbettung von zunächst in ʟ-Arginin gelöstem GFP nach der Reinigung führte zu einer 1,3-fach höheren Fluoreszenzintensität der resultierenden Nanopartikel im Vergleich zu den Nanopartikeln, die über den analogen Einbettungsprozess ausgehend von in NaHCO gelöstem GFP erhalten wurden3 . In ähnlicher Weise zeigte GFP, verdünnt in ʟ-Arginin, eine höhere Fluoreszenzintensität im Vergleich zu GFP, verdünnt in NaHCO3 (Zusätzliche Datei 5:Abbildung S5). Der Effekt könnte durch die unterschiedlichen pH-Werte der Puffer erklärt werden (pHʟ-Arginin = 10,3, pH\( _{{\mathrm{NaHCO}}_3} \) = 9.2).
Aus diesem Grund wurde die Fluoreszenz des reinen GFP systematisch in Abhängigkeit vom pH-Wert gemessen (SI, Zusatzdatei 6:Abbildung S6). Die Daten zeigten einen hyperbolisch geformten Anstieg der Fluoreszenz mit steigendem pH-Wert im Bereich von pH 5,5 - 10,5. Die Ergebnisse stimmen mit anderen Berichten über die pH-abhängige Fluoreszenz von GFP überein. Für Wildtyp-GFP wurde berichtet, dass die Fluoreszenz im Bereich von pH 6 - 10 unverändert ist, aber bei niedrigeren pH-Werten abnimmt und bei pH-Werten> 10 zunimmt [29]. Darüber hinaus konnte die pH-Sensitivität von GFP durch Einführung von Punktmutationen modifiziert werden [30]. Das in dieser Studie verwendete GFP besitzt im Vergleich zum Aequorea . Dreipunktmutationen Wildtyp-Protein, nämlich S2A, F64L, S65T. Von diesen hat sich gezeigt, dass die Substitution von Serin an Position 65 durch Threonin die Fluoreszenzintensität des Proteins erhöht, wenn es bei 480 nm angeregt wird, da diese Aminosäure an der Bildung des Chromophors beteiligt ist. Darüber hinaus weist die Variante S65T/F64L eine pH-abhängige Fluoreszenz auf [30]. Die GFP-dotierten Nanopartikel (CF ) zeigte eine vergleichbare pH-abhängige Fluoreszenz (Abb. 3), was darauf hindeutet, dass der Mechanismus der pH-Abhängigkeit durch den Einbettungsprozess nicht beeinflusst wurde.
Fluoreszenz-Quantenausbeute
Um die Eigenschaften der fluoreszierenden Nanopartikel weiter zu charakterisieren, wurden deren Quantenausbeuten bestimmt. Dies wurde erreicht, indem die integrierte Fluoreszenzintensität gegen die Absorption bei 488 nm aufgetragen wurde (Abb. 4). Anschließend wurden die Quantenausbeuten mit Gl. 2. Mit Rhodamin 6G als Referenz die Quantenausbeuten der GFP-dotierten Nanopartikel CF S1F und die reine GFP wurden bestimmt zu φ\( _{{\mathrm{C}}_{\mathrm{F}}{\mathrm{S}}_{1\mathrm{F}}} \) = 0.62 und φreines GFP = 0,38. Die Ergebnisse wurden mit Atto488 als zweiter Referenz bestätigt (SI, Additional file 7:Figure S7). Die höhere Quantenausbeute von GFP-dotierten Nanopartikeln im Vergleich zu reinem GFP scheint auf die Einkapselung von GFP in die Silicamatrix zurückzuführen zu sein und könnte entweder mit der räumlichen Immobilisierung des Proteins oder der veränderten chemischen Umgebung der Silicamatrix zusammenhängen .
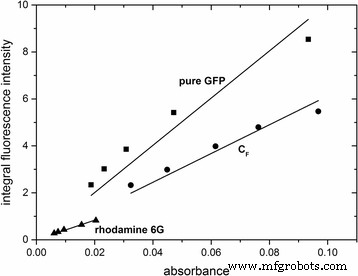
Diagramme der integrierten Fluoreszenzintensität von GFP-dotierten Partikeln und reinem GFP gegen die Absorption bei 488 nm. Als Referenz diente Rhodamin 6G. Die lineare Korrelation wurde durch die Geraden . angepasst . Die entsprechenden linearen Gleichungen lauten wie folgt:y reines GFP = 1.00554 × 10 10 × x , R 2 =0,97712; y\( _{C_F{S}_{1F}} \) = 6.12332 × 10 9 × x , R 2 =0,99331; y rhodamin6G = 4.1772 × 10 9 × x , R 2 = 0,99678
Partikelstabilität
Proteinleckage
Auslaugungsexperimente wurden durchgeführt, um die Bindungsstabilität der GFP-dotierten Nanopartikel nachzuweisen. Nach Ultrafiltration durch Membranen mit einem MWCO, der den Durchtritt von GFP (MW~27 kDa) ermöglicht, aber Nanopartikel zurückhält, konnte im Filtrat keine Fluoreszenz nachgewiesen werden, was auf eine permanente Kopplung von GFP an die Silica-Matrix hinweist.
Analytische Ultrazentrifugation
Um die erhaltenen Ergebnisse zu untermauern und die Art der GFP-Bindung an die Partikelmatrix zu bestimmen, wurde eine analytische Ultrazentrifugation durchgeführt. Zu diesem Zweck beschriftet mit CF S1F S2F Partikel und unbeschriftetes CU S1U S2U particles mixed with GFP were measured at the same particle and GFP concentrations. The results (Additional file 8:Figure S8 in the SI) indicate that most of the GFP molecules are embedded into the silica matrix during the synthesis.
Thermal Stability
To determine their thermal stability, the fluorescence signals of CF in comparison to pure GFP were measured after incubation at room temperature and 60 °C respectively (Fig. 5). After 24 h at room temperature, no decrease in the fluorescence of both samples was detectable, indicating no influence on the protein stability. However, after 24 h at elevated temperature of 60 °C, only 20% of the initial fluorescence intensity of CF could be observed, whereas no fluorescence signal of pure GFP reverted. This strongly indicates a higher thermal stability of GFP-embedded silica compared to pure GFP. Since an elevated temperature leads to a significant increase in the thermal motions of the protein molecule, which can disrupt its structure, it is hypothesised that the surrounding silica matrix protected the GFP against external influences by spatial constraints.
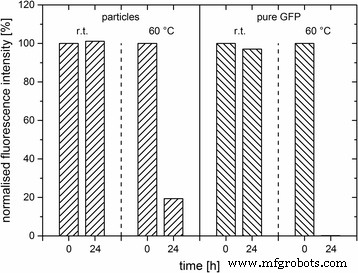
Influence of temperature (r.t., 60 °C) on the fluorescence of GFP-doped particles (CF , ʟ-arginine) and pure GFP. The normalised fluorescence intensity [%] of the emission maximum at 508 nm versus time [h] is shown
Photostability
Furthermore, the photostability of the samples was tested. For measurements, the nanoparticle stock suspension (CF , ʟ-arginine) was diluted tenfold. Pure GFP was diluted in ʟ-arginine according to the calculated concentration of GFP in the nanoparticle suspension. After exposure of the samples to light of a green LED array over a period of time up to 20 min, the fluorescence intensity was measured (Fig. 6). Within 20 min, the fluorescence intensity of the nanoparticle suspension decreased only slightly. After 20 min, 89% of the initial fluorescence (100%) of the nanoparticles was preserved. In comparison, the pure GFP seemed to be more affected by light exposure. After 20 min, only 81% of the initial fluorescence of pure GFP remained. This result indicated, that GFP, when embedded into silica nanoparticles, was better protected from photochemical alterations induced by the LED light than the pure protein.
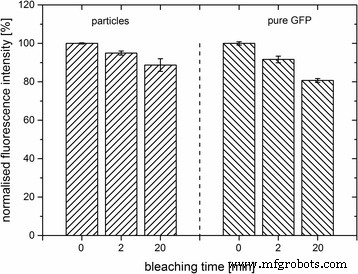
Photostability of GFP-doped nanoparticles (CF ) and pure GFP in ʟ-arginine. The normalised fluorescence intensity [%] of the emission maximum at 508 nm was measured after exposure to LED light for the given times. Data are mean values. Error bars indicate the standard deviation
Stability Against Protein Degradation
As a further characterisation step, the degradation of GFP in the presence of proteinase K was tested. Therefore, three different systems were used (pure GFP, unlabelled CU S1U S2U mixed with GFP and labelled CF S1F S2F ). For all systems, equal amounts of GFP and particles were used. After 90 min of incubation, the fluorescence intensity of pure GFP and unlabelled particles with added GFP decreased to 5 - 7% of the initial fluorescence intensity, whereas the one of the labelled particles decreased to 52% (Fig. 7). This result indicates that the GFP is protected by the silica matrix and is degraded slower than free GFP in presence of proteolytic enzymes.
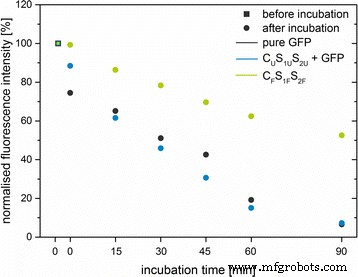
Stability against protein degradation of pure GFP (grey ), unlabelled particles mixed with GFP (CU S1U S2U , blue ), and GFP-doped silica nanoparticles (CF S1F S2F , green ). The normalised fluorescence intensity [%] of the emission maximum at 508 nm was plotted against the incubation time [min] with proteinase K
To conclude, the encapsulation of GFP into silica matrix appeared to bring about significant advantages:The stability of the protein was increased not only against elevated temperatures and light-induced photobleaching but also against the degradation through enzymes. Therefore, the silica matrix seems to protect the embedded GFP as compared to the free GFP.
Cellular Uptake Experiments
In order to determine, if the GFP-doped nanoparticles are able to deliver their cargo into cells, uptake experiments were performed (Fig. 8). A549 cells were exposed to GFP-doped nanoparticles and for comparison to the pure protein. In order to optimise the GFP load of the particles for imaging, a higher amount of GFP as compared to the nanoparticles described before was embedded into the particles. More specifically, a 20-fold amount of GFP in ʟ-arginine was used to label the second shell of the CF S1F S2F particles. These nanoparticles were diluted to a final concentration of 37 μg SiO2 per millilitre in cell culture medium and incubated for 24 h with the cells. The amount of GFP in both samples (nanoparticles and pure GFP) was 5 μg mL −1 .
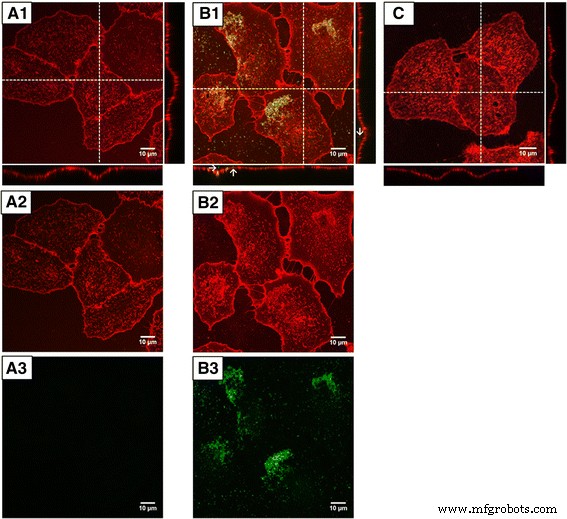
Confocal microscopy images of A549 cells after 24 h exposure to GFP dissolved in ʟ-arginine (A1 –A3 ) and GFP-doped nanoparticles CF S1F S2F (B1 –B3 ), and control cells (C ). Top (1):merge-images; middle (2):Cell membrane (WGA):red; bottom (3):GFP, green . Arrows indicate internalised nanoparticles. Contrast and brightness were enhanced by using the ImageJ software
In order to visualise the cells, the cell membrane was labelled, using tetramethylrhodamine-coupled WGA (wheat germ agglutinin). Confocal imaging was used to localise the GFP-doped nanoparticles and the pure GFP in the cells. After exposure of cells to GFP, no signal related to GFP was observed inside the cell bodies (Fig. 8a). Compared to the control cells, no difference in signal intensity of both channels could be observed (Fig. 8c).
In contrast, after exposure of the cells to the GFP-loaded nanoparticles, bright fluorescence signals were detected in the perinuclear region, indicating internalisation of the loaded nanoparticles through endocytosis. The GFP-loaded nanoparticles appeared to be excluded from the nuclear compartment. A second fraction of agglomerated nanoparticles was detected on top of the cell membrane (Fig. 8b).
In conclusion, the GFP-doped nanoparticles are internalised by the cells and are able to transport their cargo into the cells. After exposure of the cells to GFP, fluorescence signals were not detected inside the cell body. This finding is in line with the results of Pesce et al. [31], who did not observe cell-associated fluorescence after incubation of A549 cells with GFP for 24 h. The lack of cell-associated GFP signals might be due to the fact that GFP is not internalised by the cells. Alternatively, GFP fluorescence might be quenched by the low pH value present in endocytic vesicles or lysosomes or degraded by proteolytic enzymes. Therefore, the fluorescence signals of the nanoparticles might indicate a protective effect of the silica nanoparticle matrix against lysosomal degradation.
Conclusions
In this study, a novel approach is presented for synthesis of monodisperse GFP-doped silica nanoparticles with a mean particle-core size of 15 nm. By subsequent growth steps, the particle size and the amount of embedded GFP can be varied. At the end of this procedure, the fluorescence properties of GFP are kept. Incorporation of GFP into additional outer shells results in an increase in the nanoparticle fluorescence. Coverage of the nanoparticles by non-doped shells seems to slightly decrease their fluorescence. These properties indicate the potential to incorporate cargo molecules into specific particle shells. The GFP-doped nanoparticles exhibit a higher quantum yield as compared to the pure GFP. The incorporation into the silica matrix appeared to be durable, as no leaching of protein was detected by ultrafiltration. The silica matrix also seems to improve the thermal properties and photostability of the protein. Furthermore, it is possible to encapsulate different proteins in the different shells, in order to prepare multifunctional nano-carriers. Finally, the nanoparticles are applicable for intracellular delivery of their cargo. The incorporation of proteins into the particle matrix seems to increase delivery and reduce lysosomal degradation of the cargo. Therefore, the protein-doped silica nanoparticles constitute a promising novel tool for biomedical applications of nanoparticles, especially in the field of intracellular delivery of macromolecules.
Abkürzungen
- AUC:
-
Analytical ultracentrifuge
- DLS:
-
Dynamische Lichtstreuung
- FCS:
-
Foetal calf serum
- GFP:
-
Green fluorescent protein
- MW:
-
Molekulargewicht
- MWCO:
-
Molecular weight cut off membrane
- PBS:
-
Phosphate buffered saline
- r.t.:
-
Raumtemperatur
- TEM:
-
Transmission electron microscopy
- TEOS:
-
Tetraethoxysilane
Nanomaterialien
- Fortschritte und Herausforderungen fluoreszierender Nanomaterialien für die Synthese und biomedizinische Anwendungen
- 131I-gespurte PLGA-Lipid-Nanopartikel als Wirkstoffträger für die gezielte chemotherapeutische Behandlung von Melanomen
- Einfache Synthese von auf MWNT verankerten SiO2@C-Nanopartikeln als Hochleistungsanodenmaterialien für Li-Ionen-Batterien
- Neuartige biokompatible Au-Nanostars@PEG-Nanopartikel für die In-vivo-CT-Bildgebung und renale Clearance-Eigenschaften
- Nachbehandlungsmethode zur Synthese monodisperser binärer FePt-Fe3O4-Nanopartikel
- Ein einfacher Ansatz zur Synthese von fluoreszierenden Kohlenstoffquantenpunkten aus Tofu-Abwasser
- Synthese und In-vitro-Leistung von polypyrrolbeschichteten Eisen-Platin-Nanopartikeln für die Photothermie und die photoakustische Bildgebung
- Grüne Kompetenz bei der Synthese und Stabilisierung von Kupfernanopartikeln:katalytische, antibakterielle, zytotoxische und antioxidative Aktivitäten
- Neuer dualer mitochondrialer und CD44-Rezeptor, der auf Nanopartikel für die Redox-Stimuli-getriggerte Freisetzung abzielt
- Platycodon-Saponine aus Platycodi Radix (Platycodon grandiflorum) für die grüne Synthese von Gold- und Silber-Nanopartikeln



