Verwendung positiv geladener magnetischer Nanopartikel zum Einfangen von Bakterien bei extrem niedriger Konzentration
Zusammenfassung
Der Nachweis von Bakterien in geringen Konzentrationen ohne zeitaufwendige Kulturprozesse würde eine schnelle Diagnose ermöglichen. Da zwischen negativ geladenen Bakterienzellen und positiv geladenen magnetischen Nanopartikeln (NP+) elektrostatische Anziehung besteht, ist der Einfang von Bakterien vielversprechend, um dieses Ziel zu erreichen. Hier präsentieren wir einen schnellen und hocheffizienten Ansatz zur Erfassung von Escherichia coli , das als Modell für gramnegative Bakterien verwendet wurde. Erfassung von E. coli bei sehr niedrigen Konzentrationen von 10 und 100 KBE/ml mit NP+ wird schnell und effizient innerhalb von 1 h erreicht. Darüber hinaus betrug die Einfangeffizienz von NP+ über 90%, wenn die Anzahl der Bakterienkolonien auf der Platte analysiert wurde. Optische und Transmissionselektronenmikroskopie bestätigten die Fähigkeit zum Einfangen von Bakterien durch elektrisch geladene Nanopartikel (NPs). Im Gegensatz dazu zeigten negativ geladene magnetische Nanopartikel (NP−) keine Affinitäten zu E. coli . Diese Ergebnisse zeigten, dass Bakterienzellen wie E. coli , eine negative Ladung tragen. Im Gegensatz zu einem ligandenabhängigen Einfangsystem verfügt unser entwickeltes NP+ über das Potenzial, eine breite Palette von Bakterien durch elektrostatische Anziehung einzufangen.
Hintergrund
Infektionskrankheiten gehören zu den dringendsten Gesundheitsproblemen der Welt. Die mikrobielle Kontamination von Wasserressourcen ist eine große Bedrohung für die öffentliche Gesundheit. Escherichia coli (E. coli ), ein gramnegatives Bakterium, kommt sehr häufig in kontaminiertem Wasser und Lebensmitteln vor. Einige Stämme von E. coli kann sogar schwere bakterielle Infektionen verursachen. Bakterien in geringen Konzentrationen sind schwer nachzuweisen und erfordern in der Regel eine Voranreicherung vor der weiteren Analyse. Kulturbasierte mikrobiologische Methoden sind aufwendig und können mehrere Tage dauern. Darüber hinaus können einige Bakterienstämme einen lebensfähigen, aber nicht kultivierbaren Zustand erreichen, in dem sie auf Routineagar lebensfähig, aber nicht kultivierbar sind, was ihren Nachweis durch kulturbasierte Methoden erschwert [1]. Umgekehrt könnte eine schnelle Erfassung und Dekontamination von bakteriellen Krankheitserregern Echtzeitergebnisse liefern, um den Ausbruch von Infektionskrankheiten einzudämmen.
Für das schnelle Einfangen und Entfernen von Bakterien aus der kontaminierten Quelle wurden verschiedene Materialien entwickelt. Kohlenstoffnanoröhren und harzgebundene Oligoacyllysin-Beads wurden verwendet, um die Bakterien aus dem Wasser zu entfernen [2, 3]. Magnetische Nanopartikel, die durch den Einsatz magnetischer Verfahren bequem von verschiedenen Ressourcen getrennt werden können, wurden nach der Funktionalisierung mit organischen Molekülen häufig zum Nachweis von Bakterien und zur Dekontamination verwendet [4,5,6]. Die magnetbasierten Techniken haben die Vorteile der Zielerfassung durch Zeitersparnis (gemeinsame Trennzeit innerhalb von 1 h), hohe Wiederfindung, mögliche Automatisierung und Scale-up-Trennung [7]. Die Effizienz und Selektivität der magnetischen Trennung hängt stark von den Liganden ab, aber manchmal ist es schwierig, einen Liganden mit hoher Affinität und Spezifität für das Target zu erhalten. Daher ist es notwendig, ein bakterielles Einfangsystem mit ligandenunabhängigen magnetischen Nanopartikeln zu entwickeln, um die Bakterien insbesondere bei niedrigen Konzentrationen einzufangen.
Viele Wissenschaftler haben die Natur der elektrischen Ladung von Bakterien untersucht. Bechhold (1904) fand erstmals heraus, dass Bakterienzellen eine negative Ladung tragen [8]. Während bereits bekannt war, dass die großen Populationen von Bakterienzellen dazu neigen, eine negative Ladung aufrechtzuerhalten, ist über die Elektrophysiologie von Bakterien auf der Ebene einzelner Zellen wenig bekannt. Im Jahr 2011 haben Cohen et al. zeigte elektrisches Spiking in E. coli bei bis zu 1 Hz unter Verwendung eines fluoreszierenden spannungsanzeigenden Proteins [9]. Da viele Arten von Bakterienzellwänden negativ geladen sind, können positiv geladene Nanopartikel über elektrostatische Wechselwirkungen stark mit einem breiten Spektrum von Bakterien interagieren.
Um magnetische Nanopartikel und die negative Ladung einzelner Bakterien für einen schnellen Erregernachweis zu nutzen, haben wir ein System zum Einfangen von Bakterien in geringen Konzentrationen entwickelt. Positiv geladene magnetische Nanopartikel wurden aus Polyethylenimin (PEI) hergestellt, das aus zahlreichen Amingruppen besteht. Dann untersuchten wir die Affinität von PEI-funktionalisierten Nanopartikeln gegenüber E. coli . Diese innovative Methode bietet eine effiziente Bindung an die Bakterien durch elektrostatische Wechselwirkungen.
Materialien und Methoden
Nanomaterialien
Eisen(III)-chloridhydrat (FeCl3 .) ·6H2 O), Ammoniumhydroxid (NH4 .) OH, 28 Gew.-%, Salzsäure (37 Gew.-% wässrige Lösung), Ethylenglykol und Natriumacetat wurden von Shanghai (China) Reagent Company bezogen. Tetraethylorthosilicat (TEOS), (3-Aminopropyl)triethoxysilan (APTES) und Fluoresceintetramethylrhodamin (TRITC) wurden von Sigma-Aldrich (USA) bezogen. Verzweigtes Poly(ethylenimin) (PEI, 99%, Mw =10.000) wurde von Alfa Aesar gekauft. Alle Lösungen wurden unter Verwendung von Milli-Q entionisiertem Wasser hergestellt (18,2 MΩ cm bei einem spezifischen Widerstand von 25°C).
NP-Synthesen
Fe3 O4 Nanopartikel wurden durch eine solvothermale Reaktion hergestellt [10]. Kurz gesagt, 0,081 g FeCl3 ·6H2 O wurde in 30 ml Ethylenglykol unter magnetischem Rühren gelöst. Dann wurden dieser Lösung 0,3 µg Polyacrylsäure (PAA) und 1,8 µg Harnstoff zugesetzt. Nach 30 min Rühren wurde die Lösung 12 h bei 200 °C unter Verwendung eines mit Teflon ausgekleideten Edelstahlautoklaven erhitzt. Beim Abkühlen auf Raumtemperatur wurde ein schwarzes Produkt, nämlich magnetische Nanopartikelkerne, von einem Magneten gesammelt. Nach dreimaligem Waschen mit Ethanol und entionisiertem Wasser wird das Fe3 O4 Nanopartikel wurden mit 0,15 M HCl unter Ultraschall für 15 min behandelt und dann durch Hydrolyse und TEOS mit Silica beschichtet.
Um die negativ geladenen fluoreszierenden magnetischen Nanopartikel (NP−) herzustellen, wurde der APTES-TRITC (C33H44N3O6Si)-Komplex zunächst unter dunklen Bedingungen über Nacht in Ethanol umgesetzt. Der Komplex wurde dann auf das Fe3 . gepfropft O4 Nanopartikel durch Reaktion zwischen APTES und Hydroxylgruppen am Fe3 O4 @SiO2 Nanopartikel. Anschließend wurden 30 µl TEOS zugegeben und 24 Stunden im Dunkeln umgesetzt. Nach dreimaligem Waschen mit Ethanol und entionisiertem Wasser wurden fluoreszierende NP– hergestellt. Durch die Modifikation von NP− mit dem Polykation-Polymer PEI wurden die positiv geladenen magnetischen Nanopartikel (NP+) fertiggestellt.
NP-Charakterisierung
Untersuchungen mit Transmissionselektronenmikroskopie (TEM) wurden von einem TECNAI F − 30 . durchgeführt hochauflösendes Transmissionselektronenmikroskop bei 300 kV. Die Partikelgröße und das Zetapotential von NPs wurden mit der Malvern Zeta Sizer Nano-Serie (Westborough, MA) bestimmt. Die Fluoreszenz wurde mit einem konfokalen Laser-Scanning-Mikroskop Carl Zeiss LSM5 EXITER (Zeiss, Jena, Deutschland) untersucht.
Bakterienvorbereitung
Der gramnegative Stamm E. coli Als Modellbakterien wurden BL21 verwendet. E. coli wurde in 100 µl Luria Broth Wachstumsmedium kultiviert [11]. Die Bakterien wurden in einem thermostatischen Inkubator bei 200 U/min, 37 °C für 16 Stunden kultiviert. Anschließend wurden 100 μL der Bakterienkultur entnommen, mit dem Medium 1 × 10 5 . verdünnt auf den Agar beschichtet und 24 h bei 37 °C kultiviert. Die Anzahl der koloniebildenden Einheiten wurde gezählt. Die verbleibenden Bakterienzellen wurden durch Zentrifugation bei 5000 U/min für 5 Minuten geerntet, dreimal mit 1 × PBS (10 μM, pH 7,4) gewaschen und auf Konzentrationen von ungefähr 1 × 10 3 . verdünnt Koloniebildende Einheit (KBE)/ml. Aus Sicherheitsgründen wurden alle Bakterienproben vor der Entsorgung in einen Autoklaven bei 121 °C für 20 °C gegeben, um Bakterien abzutöten, und alle Glaswaren, die mit den Bakterien in Kontakt kamen, wurden vor und nach der Verwendung sterilisiert.
Experiment zum Einfangen von Bakterien
Alle Batch-Capture-Studien wurden in sterilisiertem 1 × PBS-Puffer durchgeführt. Vierzig Mikroliter NPs (1 µg/µl) wurden in der sterilisierten Kochsalzlösung unter Ultraschall für 10 min dispergiert und dann 1 µl der Bakterienlösung (ungefähr 10 3 CFU/ml) wurde der Suspension zugesetzt. Nach 10&supmin; Inkubation wurden die NP-gebundenen Bakterien über einen Permanentmagneten an der Gefäßwand eingefangen und freie Bakterien mit der Waschlösung entfernt. Die eingefangenen Bakterien wurden durch Entfernen des Magneten freigesetzt und in PBS resuspendiert. Für die mikroskopische Analyse wurde ein Aliquot von Bakterien auf Objektträger verteilt und mit Hema-3 (Fisher Diagnostics) gefärbt. Für die Immunfluoreszenzanalyse wurde ein Aliquot der Bakterien auf Objektträger ausgestrichen und mit 4′-6-Diamidino-2-phenylindol (DAPI) gefärbt.
Bakterielle Einfangeffizienz von NPs bei verschiedenen Konzentrationen
Ein Milliliter Bakteriensuspension (ca. 2 × 10 2 CFU/ml) wurde mit unterschiedlichen Mengen (5, 10, 20, 30, 40, 50, 75 und 100 µg/ml) NP+ oder NP− für 10 min inkubiert. Nach der magnetischen Abtrennung durch die Nanopartikel wurde der Gesamtlösung dann eine Probe entnommen und mittels einer Plattenzählmethode auf Bakterienkonzentration analysiert. Die Bakterieneinfangeffizienz der NPs wurde durch Zählen der KBE auf den LB-Agarplatten getestet.
Fähigkeit von NPs, Bakterien in geringen Konzentrationen einzufangen
40 Mikrogramm NP+ oder NP− wurden mit 1 µl Bakteriensuspension in sehr geringen Konzentrationen (10 und 10 2 KBE/ml). Nach der magnetischen Abtrennung durch die Nanopartikel wurde der Gesamtlösung dann eine Probe entnommen und mittels einer Plattenzählmethode auf Bakterienkonzentration analysiert. Die Bakterieneinfangeffizienz der NPs wurde durch Zählen der KBE auf den LB-Agarplatten getestet.
Statistische Analyse
Die Ergebnisse wurden als Mittelwert ± Standardabweichung (SD) ausgedrückt, wie in den Legenden der Abbildung angegeben. Eine Zwei-Wege-Varianzanalyse (ANOVA) mit richtiger Hoc-Analyse wurde mit der GraphPad Prism-Software mit P . berechnet Werte < 0,05 gelten als statistisch signifikant.
Ergebnisse
Charakterisierung magnetischer NPs
Das schematische Diagramm zur Herstellung der oberflächengeladenen magnetischen Komposit-Nanopartikel ist in Abb. 1 dargestellt. Das Fe3 O4 Nanopartikel werden mit APTES konjugiert, um eine dünne Schicht aus SiO2 . zu bilden Schale auf der Oberfläche von Nanopartikeln bei Reaktion mit TEOS und NH4 OH. Um eingefangene Zellen direkt sichtbar zu machen und zu quantifizieren, wird zunächst der APTES-TRITC-Komplex umgesetzt, gefolgt von der Pfropfung auf die Oberfläche des Fe3 O4 @Silica-Komposite durch eine klassische Sol-Gel-Reaktion. Die gesamte Oberfläche dieses Produkts wird von zahlreichen SiOH-Gruppen bestimmt, die eine starke negative Oberflächenladung aufweisen, nämlich negativ geladene magnetische Nanopartikel (NP−). Bei positiv geladenen magnetischen Nanopartikeln (NP+) werden PEI-Moleküle verwendet, um die Oberfläche von NP− zu bedecken und zu modifizieren. Das modifizierte Produkt weist aufgrund der reichlich vorhandenen Amingruppen eine stark positive Oberflächenladung auf.
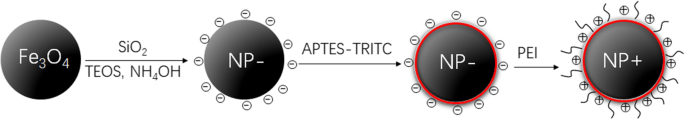
Design der Nanopartikel. Schematische Darstellung des Designs oberflächengeladener, fluoreszierender, superparamagnetischer Nanopartikel (NPs)
Wie in Abb. 2a gezeigt, zeigte TEM, dass magnetische Verbundnanopartikel einen Durchmesser von 450 nm aufwiesen, die aus gleichförmigem SiO2 . bestanden Beschichtung (Größe von 60 nm). Die dynamische Lichtstreuung (DLS) der Partikel in Abb. 2b zeigte eine enge Größenverteilung mit einem erhöhten durchschnittlichen Durchmesser nach der Oberflächenfunktionalisierung. Die maximalen Größen der zusammengesetzten Nanopartikel mit positiver und negativer Ladung betragen 620 bzw. 700 nm. Mit DLS gemessene Nanopartikel sind normalerweise größer als die mit TEM gemessenen. Dies liegt daran, dass die DLS-bewertete Größe durch die Brownsche Bewegung beeinflusst wird und von der Umgebungstemperatur, dem dynamischen Radius des Nanopartikels und dem Ausmaß der Nanopartikel-Agglomeration abhängt, die durch eine statische Umgebung über das Auftreten von Konflikten ausgelöst wird [12]. Abbildung 2c zeigt die Zetapotenzialverteilungen der negativen und positiven Nanopartikel. In entionisiertem Wasser (pH 7.0) betragen die Zetapotentiale von NP− und NP+ − 26.6 mV bzw. + 28.1 mV. Die pH-Abhängigkeiten der Zetapotentiale für NP+ sind in Zusatzdatei 1:Abbildung S1 dargestellt. Diese Ergebnisse zeigten, dass die oberflächengeladenen Nanopartikel unter neutralen Bedingungen in wässriger Lösung gut dispergiert sind, was zum Einfangen von Zellen verwendet werden könnte.
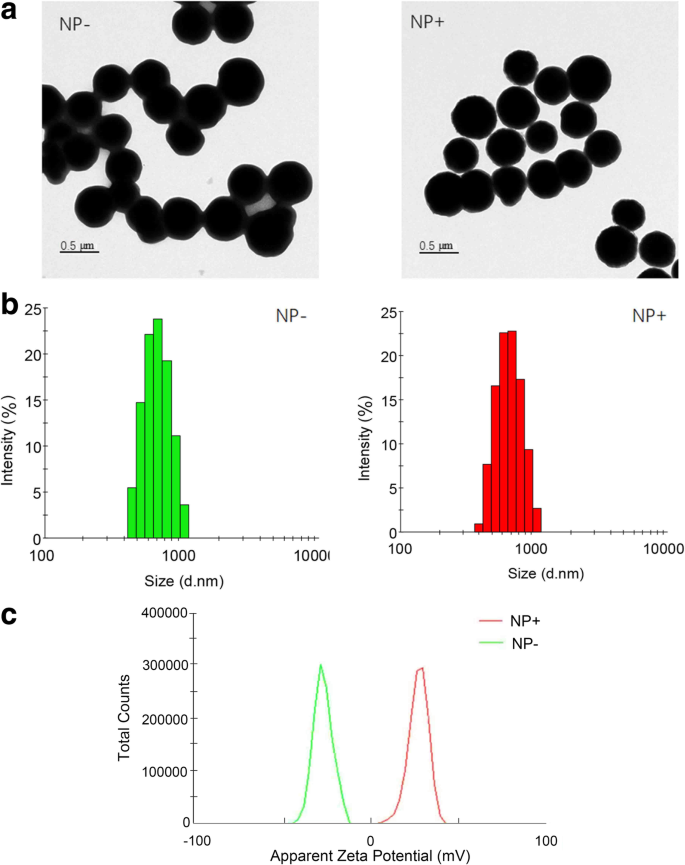
Charakterisierung der Nanopartikel. a Transmissionselektronenmikroskopische (TEM) Aufnahme von positiv geladenen Nanopartikeln (NP+) und negativ geladenen Nanopartikeln (NP−). b Dynamische Lichtstreuungsgröße und Verteilung von NPs. c Zeta-Potentialverteilungen von NPs
Fähigkeit magnetischer NPs, E. coli
Abbildung 3 zeigt das allgemeine experimentelle Verfahren zum Einfangen von Bakterien. Die NPs wurden mit einer Bakterienlösung vermischt und 10 min bei Raumtemperatur inkubiert. Anschließend haben wir die „magnetisierten“ Bakterien (an die Zelloberfläche gebundene magnetische Nanopartikel) mit einem Permanentmagneten an der Wand des Röhrchens festgehalten. Nach dem Entfernen der restlichen Lösung und dem Waschen der Aggregate mit PBS (mit einem Magneten außen) übertrugen wir die Aggregate zur mikroskopischen Analyse auf einen Objektträger. Wie im Schema gezeigt, bietet NP+ eine ausreichende elektrostatische Reaktionsfähigkeit, um E schnell anzureichern. coli . Im Gegensatz dazu werden die Bakterien im NP−-Experiment mit Spülen von PBS entfernt.
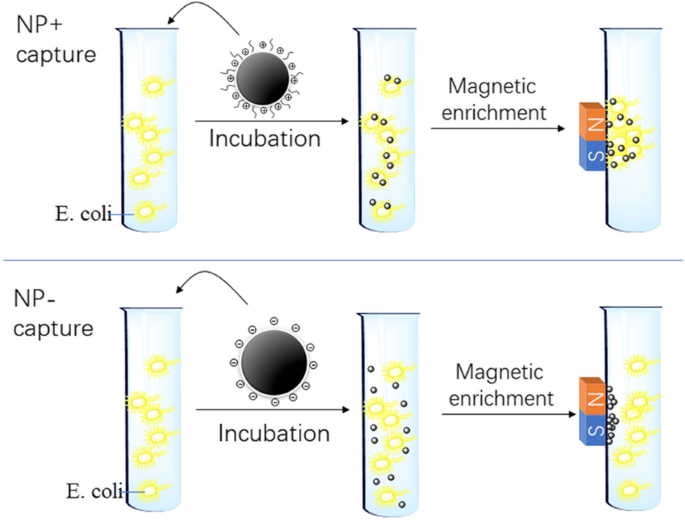
Illustration der Verfahren zum Einfangen von Bakterien. E. coli in Suspension werden jeweils mit NP+ und NP– gemischt, gefolgt von 10 min Inkubation. Die „magnetisierten“ Bakterien wurden von einem Magneten an die Wand des Fläschchens angezogen. Nach dem Entfernen der restlichen Lösung wurden die Bakterien von NPs eingefangen, mit PBS gewaschen und anhand der Anzahl der Bakterienkolonien gezählt
Optische Bilder für die Nanopartikel sind in Abb. 4 gezeigt. Optische Bilder von NP− zeigten die monodispersen und gleichmäßig verteilten Partikel. NP+ neigte jedoch zur Agglomeration. Die Größenverteilung von NP+ ist offensichtlich breiter als die von NP−. Diese Ergebnisse zeigten, dass NP+ und NP− ein völlig unterschiedliches Interaktionsmuster mit E aufweisen. coli . Um die bakterielle Affinität von NP+ weiter zu untersuchen, wurde die Lokalisation von NP+ in E. coli wurde mittels Fluoreszenzanalyse untersucht. Abbildung 5a zeigt, dass das erfasste E. coli sind sowohl für DAPI (blaue Farbe) als auch für TRITC (rote Farbe) positiv. Um die Affinität von NP+ für Bakterien eindeutig zu bestätigen, wurde die TEM-Technologie verwendet. In Abb. 5b wurde beobachtet, dass eine Reihe von NP+ an der Bakterienzellwand aggregiert. Diese Ergebnisse legen nahe, dass NP+ eine starke Affinität zu Bakterien hat.
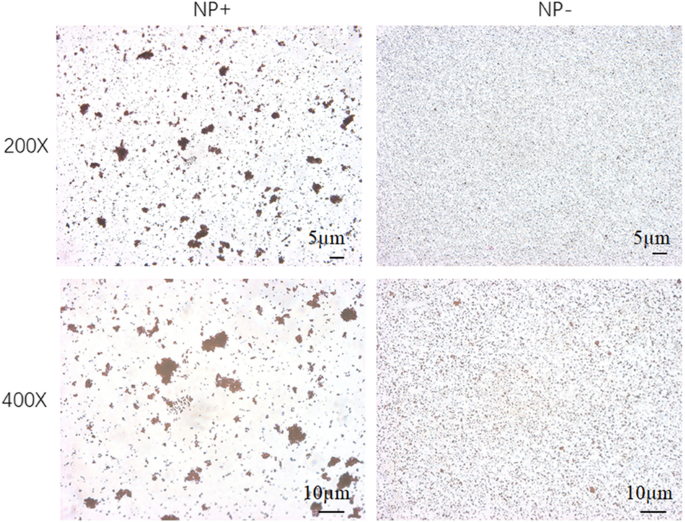
Vergleich von E. coli Bindungskapazität von NP+ und NP−. Die linken Bilder sind die Phasenkontrastfelder von Bakterien, die mit NP+ binden. Die rechten Bilder enthalten nur Nanopartikel, was darauf hindeutet, dass NP− ohne Bindungskapazität an E. coli Zellen
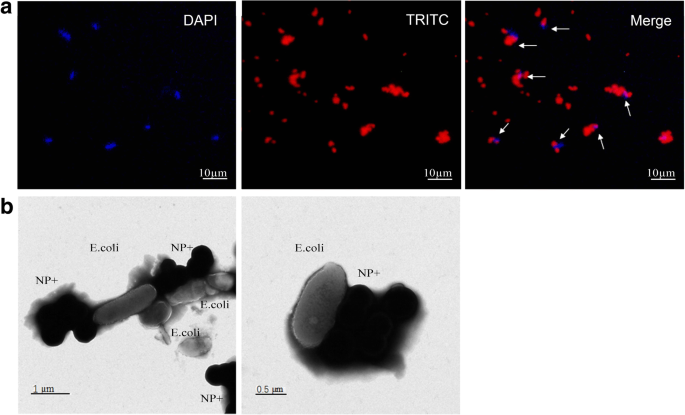
Fluoreszenz- und TEM-Bilder von E. coli Bindung mit NP+. a Die oberen Felder zeigen die Fluoreszenzbilder von E. coli Zellen, die mit NP+ binden. DAPI wird verwendet, um den Zellkern zu färben. TRITC ist in NPs beschriftet. b Untere Felder sind repräsentative TEM-Bilder, die NP+ und E zeigen. coli Komplexe
Nachweis niedriger Konzentrationen von E. coli
Um die Dynamik der Bakterien- und NP+-Wechselwirkungen weiter zu charakterisieren, inkubierten wir E. coli bei einer konstanten Zahl (2 × 10 2 KBE/ml) mit verschiedenen Konzentrationen von NPs im Bereich von 5 bis 100 µg/ml. Die NP-gebundenen Bakterien wurden dann magnetisch eingefangen und getrennt. Die magnetische Einfangeffizienz von Bakterien durch NP+ und NP– ist wie in Abb. 6 dargestellt aufgetragen. Die Zahl der E. coli eingefangen von NP− beträgt selbst bei einer hohen Konzentration von 100µg/ml nur 12%. Im Gegensatz zu NP− zeigte NP+ signifikante Bakterienfangkapazitäten und erreichte mit 40 μg/ml (P < 0,001). Wie an den LB-Agarplatten zu erkennen ist, konnte eine dosisabhängige Zunahme von Bakterienkolonien mit NP+ nachgewiesen werden.
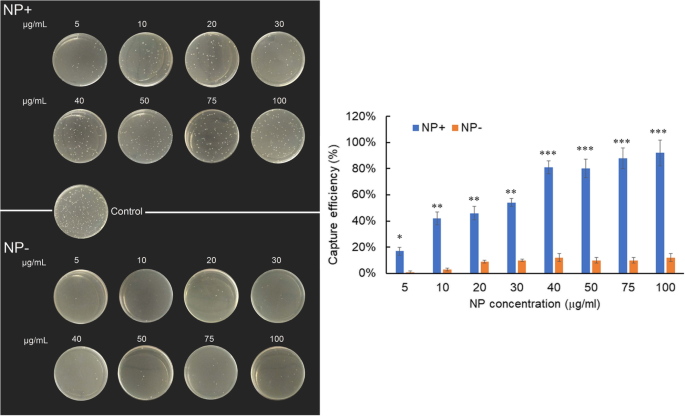
Erfassungseffizienz von E. coli durch NP+ oder NP− bei verschiedenen angegebenen Konzentrationen. Das linke Bild ist das Foto von mit E beschichteten LB-Agarplatten. coli eingefangen von NP+ und NP−. Das rechte Bild zeigt die Bakterienfangeffizienz von NP bei verschiedenen angegebenen Konzentrationen. E. coli (2 × 10 2 CFU in 1 ml PBS-Lösung) ohne NP-Inkubation wurde als 100 % gezählt und diente als Kontrolle. *P < 0,05, **P < 0.01, ***P <0,001
Um die NP+-Affinität für E. coli Bei ultraniedriger Konzentration haben wir 40 µg NP mit 1 ml PBS-Lösung gemischt, die nur 10 und 100 °CFU E enthält. coli . Abbildung 7 zeigt Fotos der resultierenden Kolonien in Agarplatten für alle Proben. Wie erwartet hat das NP+ tatsächlich E eingefangen. coli bei einer ultraniedrigen Konzentration, während die Bakterienkolonien in Platten mit NP– nicht sichtbar waren. Die wenigen Kolonien in der Platte könnten auf die unspezifische Affinität der Nanopartikel zurückgeführt werden. Weitere Analysen zeigten, dass bei einer ultraniedrigen Konzentration (10 und 100 KBE/ml) unter Verwendung von NP+ über 90 % Bakterieneinfangeffizienz erzielt wurden. Im Gegensatz dazu beträgt die Einfangeffizienz bei NP− bei gleichen Bedingungen weniger als 4% und weist einen signifikanten Unterschied auf (P < 0,001). Diese Ergebnisse legen nahe, dass NP+ eine starke Affinität zu Bakterien hat, was durch die elektrostatische Anziehung erklärt werden könnte. Um die Bakterienfangeigenschaften mit breitem Spektrum zu untersuchen, haben wir drei grampositive Bakterien (Bacillus subtilis , Staphylococcus aureus , und Lactococcus lactis ) als Vorbilder. Wie in Zusätzliche Datei 1:Abbildung S2 veranschaulicht, haben NP+ eine höhere Adsorptionskapazität für Bazillen (E. coli und B. subtilis ) als Staphylokokken (S. aureus ) und Streptokokken (L. lactis ). Darüber hinaus fanden wir auch, dass negativ geladene Moleküle wie 3-Brompyruvat (3-BP) und DNA den bakteriellen Einfangeffekt stören könnten. Der tote E.coli ist für ein solches System ungültig (zusätzliche Datei 1:Abbildung S3).
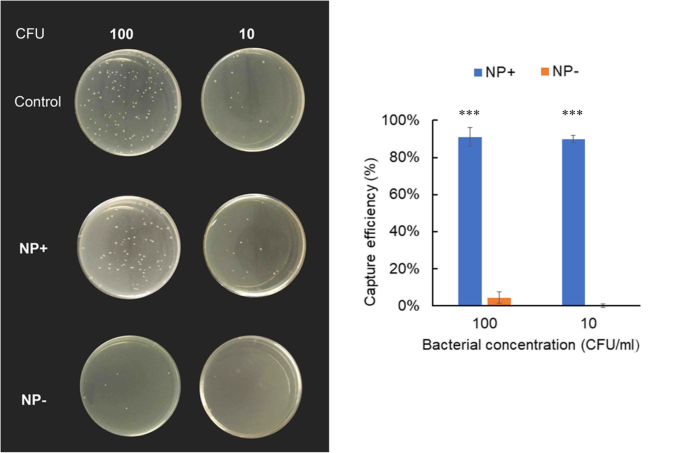
Erfassungseffizienz von E. coli durch NP+ oder NP− bei ultraniedrigen Konzentrationen. Das linke Bild ist das Foto von mit E beschichteten LB-Agarplatten. coli eingefangen von NP+ und NP−. Das rechte Bild zeigte die E. coli Erfassungseffizienz von NPs (40 µg/mL) mit ultraniedrigen Bakterienkonzentrationen. E. coli (10 und 100 CFU in 1 ml PBS-Lösung) ohne NP-Inkubation wurde als 100 % gezählt und diente als Kontrolle. ***P <0,001
Diskussion
Es ist bekannt, dass sowohl gramnegative Bakterien (wie E.coli ) und grampositive Bakterien (wie B. subtilis ) über elektrostatische Anziehung viel leichter mit positiv geladenen Teilchen interagieren als mit negativ geladenen Teilchen [13, 14]. Dies wurde auch in unserer Studie festgestellt. Ein Vorteil unseres Fangsystems besteht darin, dass die PEI-funktionalisierten Nanopartikel mehr Amingruppen aufweisen, die Bakterien in ultraniedrigen Konzentrationen einfangen konnten. Bis heute gibt es einige allgemeine und zufriedenstellende Assays, die Bakterien in Konzentrationen von weniger als 10 2 . nachweisen können KBE/ml ohne Voranreicherung von Bakterien über einen Kulturprozess. Diese Studie zeigte einen einfachen Assay, der elektrisch magnetische Nanopartikel verwendet, um gramnegative Bakterien (die Organismen haben eine zytoplasmatische Membran, eine Zellwand und eine intakte äußere Membran) innerhalb von 1 h bei einer Konzentration von 10 KBE/ml einzufangen und nachzuweisen.
In unserem Experiment haben wir festgestellt, dass die Oberflächenladung von NPs die Einfangeffizienz von Bakterien beeinflussen kann. Hier wurden die Auswirkungen der Ladung an der Oberfläche von NPs auf die Bakterieneinfangeffizienz mit E.coli . untersucht als Modellbakterien. Wenn NP+ im Einfangassay verwendet wurden, zeigten sie eine effiziente Adsorptionsfähigkeit der Bakterien. Die Bakterienfangeffizienz stieg mit der Dosierung von NP+. Die TEM-Mikroskopie zeigt makroskopische Aggregate aus Nanopartikeln und Bakterienzellen. Im Gegensatz dazu zeigte NP– selbst bei hohen Konzentrationen eine geringe Fähigkeit zum Einfangen von Bakterien. Insgesamt zeigten diese Beobachtungen, dass NP+ eine deutlich höhere Einfangfähigkeit als NP− haben.
Gram-positive und gram-negative Bakterien weisen zwar Unterschiede in ihrer Membranstruktur auf, jedoch sind die meisten von ihnen bei Kultivierung bei physiologischen pH-Werten negativ geladen [15, 16]. Die Zelloberflächenladung von Bakterienzellen wurde durch elektrostatische Interaktionschromatographie (ESIC) charakterisiert [17]. Die Zellwand grampositiver Bakterien besteht hauptsächlich aus einer dicken Peptidoglycanschicht, in die Teichonsäure eingebettet ist. Andererseits haben die gramnegativen Bakterien eine Lipopolysaccharidschicht an der äußeren Oberfläche, gefolgt von einer dünnen Peptidoglycanschicht. Die Teichonsäure und Lipopolysaccharide verleihen der Oberfläche von Bakterienzellen eine negative Ladung [18]. Zuvor zeigten die positiv geladenen Silbernanopartikel (AgNPs) eine bemerkenswerte Wirksamkeit gegen Mikroorganismen, darunter E. coli [19]. Sie fanden heraus, dass die kleineren Partikel eine größere antibakterielle Aktivität aufweisen. Wir dachten, dass größere Partikel einen anderen Vorteil der bakteriellen Absorptionsfähigkeit haben können. Erstens erreichen größere Partikel nur schwer den Kerninhalt von Zellen und verursachen die Toxizität für die Bakterien. Zweitens können sie eine größere Oberfläche und damit eine stärkere bakterielle Interaktion bereitstellen. Aus diesem Grund haben wir größere, positiv geladene Nanopartikel als „Schwamm“ zum Einfangen von Bakterien entwickelt und angewendet.
Schlussfolgerungen
Zusammenfassend haben wir mit PEI-magnetischen Nanopartikeln einen einfachen und schnellen Assay demonstriert, der E. coli erfasst und analysiert werden. Die vorhandenen Archive mit optischen und TEM-Profilen von Bakterien ermöglichen eine einfache Identifizierung eingefangener Bakterien. Die hohe Wiederfindung durch positiv geladene magnetische Nanopartikel ermöglicht den Nachweis anderer Bakterienstämme in extrem niedrigen Konzentrationen.
Abkürzungen
- APTES:
-
3-Aminopropyltriethoxysilan
- KBE:
-
Koloniebildende Einheit
- DLS:
-
Dynamische Lichtstreuung
- E. coli :
-
Escherichia coli
- NP:
-
Negativ geladene magnetische Nanopartikel
- NP+:
-
Positiv geladene magnetische Nanopartikel
- NPs:
-
Nanopartikel
- PAA:
-
Polyacrylsäure
- PEI:
-
Polyethylenimin
- SD:
-
Standardabweichung
- TEM:
-
Transmissionselektronenmikroskopie
- TEOS:
-
Tetraethylorthosilikat
- TRITC:
-
Tetramethylrhodamin
Nanomaterialien
- Herstellung und magnetische Eigenschaften von kobaltdotierten FeMn2O4-Spinell-Nanopartikeln
- Perkolationsmagnetismus in ferroelektrischen Nanopartikeln
- Untersuchungen zur Sorption von Schwermetallionen unter Verwendung von Nanokompositen aus eisenmodifizierter Pflanzenkohle
- Wechselwirkungseffekte beim Zusammenbau magnetischer Nanopartikel
- Silica-Nanopartikel für die intrazelluläre Proteinzufuhr:ein neuartiger Syntheseansatz unter Verwendung von grün fluoreszierendem Protein
- Bestimmung der katalytischen Aktivität von Übergangsmetall-dotierten TiO2-Nanopartikeln mittels spektroskopischer Oberflächenanalyse
- Auswirkung der Synthesemethode von La1 − xSr x MnO3 Manganit-Nanopartikeln auf ihre Eigenschaften
- Schwerwiegende Grenzflächeneffekte bei CoFe2O4/Fe3O4 und Fe3O4/CoFe2O4 Kern/Schale-Nanopartikeln
- Graphenoxid-basierte Nanokomposite, dekoriert mit Silbernanopartikeln als antibakterielles Mittel
- Runde Goldnanopartikel:Einfluss der Partikelgröße und -konzentration auf das Wurzelwachstum von Arabidopsis thaliana



