Nachweis von microRNA-335-5p auf einer interdigitalen Elektrodenoberfläche zur Bestimmung des Schweregrades von Bauchaortenaneurysmen
Zusammenfassung
Unter einem Bauchaortenaneurysma (AAA) versteht man die Vergrößerung der unteren Arterie der Bauchschlagader, und die Identifizierung eines Früherkennungsinstruments ist für die Diagnose dringend erforderlich. In der aktuellen Studie wurde eine Sensoroberfläche mit interdigitalen Elektroden (IDE) verwendet, um miRNA-335-5p zu identifizieren, die die Bildung von AAAs widerspiegelt. Die Gleichmäßigkeit des Silicamaterials wurde durch 3D-Profilometrie beobachtet, und die chemisch modifizierte hochleitfähige Oberfläche verbesserte die Detektion im I-V-Modus. Die gezielte miRNA-335-5p wurde dosisabhängig nachgewiesen und basierend auf linearer Regression und 3σ-Analysen wurde die Sensitivität mit einer biotinylierten Sonde zu 1 fM bestimmt. Die hohe Spezifität wurde durch die Unterscheidung der Zielsequenz von nicht komplementären und einfach und dreifach fehlgepaarten Sequenzen gezeigt. Diese Ergebnisse demonstrierten die leistungsstarke Detektion von miRNA-335-5p mit guter Reproduzierbarkeit zur Bestimmung des Schweregrads von AAA.
Einführung
Abdominale Aortenaneurysmen (AAA) sind lebensbedrohliche Erkrankungen, die als abdominale Aorta mit einem Durchmesser> 3 cm oder größer als normal definiert sind [1, 2]. Dieser Zustand tritt bei Arteriosklerose oder Plaquebildung auf, die die Wände der Bauchschlagader schwächt und zu einer Ausbuchtung nach außen führt, ähnlich einem Ballon. Im Laufe der Zeit weitet sich die Arterienwand, und diese Situation ist vergleichbar mit der Alterung von Gartenschläuchen. Der Druck des Blutes, das durch die Aorta pumpt, bewirkt, dass sich dieser geschwächte Bereich nach außen wölbt, was als Aneurysma bezeichnet wird. AAA wird gebildet, wenn der geschwächte Teil der Aorta zu Komplikationen führt [3,4,5,6]. AAA kann zum Tod durch Ruptur kleiner Aneurysmen führen. Derzeit werden körperliche Untersuchungen, computergestützte axiale Tomographie-Angiogramme, Magnetresonanztomographie und Ultraschallsonographie verwendet, um diesen Zustand zu diagnostizieren [7,8,9,10]. Es gibt jedoch keine Nachweismethoden für AAA, das häufig bei der Analyse anderer Gesundheitsprobleme identifiziert wird. Diese Situation führt zu einer verzögerten Identifizierung von AAA, was letztendlich zu unnötigen Gesundheitsproblemen führt. Um dieses Problem zu überwinden, müssen Forscher Früherkennungsmethoden entwickeln, und eine potenzielle Strategie ist die Entwicklung eines Sensorsystems.
Eine frühzeitige, schnelle und empfindliche quantitative Erkennung von Krankheiten ist ein wichtiges Ziel für die klinische Diagnose. Die gegenwärtigen Biosensor-Plattformen haben mehrere Anforderungen erfüllt und erfordern geeignete Laboreinstellungen und Schulung. Somit sind die meisten Methoden nicht portabel, was für eine optimale Point-of-Care-Erkennung erforderlich ist [11, 12]. Um Ärzte bei der Entscheidungsfindung auf genaue und schnelle Weise zu unterstützen, ist eine Analyse der Veränderungen der Biomarker-Spiegel sehr wünschenswert. Zirkulierende Biomarker, die in bestimmten Bereichen exprimiert werden, sollten weiter untersucht werden, um ein AAA zu diagnostizieren und den Behandlungsverlauf zu verfolgen. Die Identifizierung dieser Arten von zirkulierenden Biomarkern wird helfen, die Krankheit zu diagnostizieren und die Patienten nach der Behandlung weiterzuverfolgen. Um diesen Bedarf zu decken, schlägt diese Studie vor, Sensoren für geeignete Biomarker für AAA zu generieren. Der in dieser Studie vorgeschlagene Sensor (interdigitalisierte Elektrode) hat das Potenzial für eine Hochleistungsanalyse mit einer Vielzahl von Biomarkern. Es ist ein Dielektrodensystem mit abwechselnden Spalten und Fingern, das unter dielektrischen Messungen arbeitet [13,14,15].
Die Biomarker können beliebige Biomoleküle sein, zu denen DNA, RNA, Proteine, Kohlenhydrate, Lipide und deren modifizierte Formen gehören [16, 17]. Darüber hinaus haben Forscher vorgeschlagen, dass verschiedene Biomarker, wie nicht kodierende RNAs, im Zellsystem exprimiert, aber nicht in Proteine übersetzt werden [18]. Nichtkodierende RNAs werden normalerweise nicht in Proteine übersetzt und haben im Allgemeinen kurze Sequenzen [18,19,20,21]. Es wurde über verschiedene Klassen nichtkodierender RNAs wie microRNAs (miRNAs), ribosomale RNAs, Transfer-RNAs, kleine nukleoläre RNAs, kleine nukleäre RNAs, Telomerase-RNAs, snRNAs, Xist-RNAs, Vault-RNAs und 7SL-RNAs berichtet. miRNAs fungieren hauptsächlich bei der transkriptionellen und posttranskriptionellen Regulation der Genexpression und führen oft zum Gen-Silencing [22]. Vor kurzem beschrieben Forscher die Bedeutung von miRNAs für die Vorhersage von AAA und berichteten über eine Verringerung der Expression von miRNA-335-5p bei AAA-Patienten [23]. Es ist nachgewiesen, dass die Kombination klinischer Faktoren und der Expression von microRNAs die Vorhersage von Krankheiten drastisch verbessert und eine erhöhte Genauigkeit aufweist [24]. Die Forscher haben sich speziell auf miRNA-335-5p konzentriert, das bei Personen mit schnell wachsendem AAA eine signifikant minimale Reichweite aufwies [2, 23]. Darüber hinaus erhöhte eine Abnahme der miRNA-335-5p-Spiegel das Vertrauen in den Nachweis von wachsendem AAA. Mit anderen Worten, die negative Ausgabe (höhere Werte) von miR-335-5p zeigt den Schweregrad von AAA an und minimiert mühsames Screening. Dieser Befund wurde von Wanhainen et al. [23] und zeigten, dass miRNAs nützliche Biomarker für das Screening von AAA und die Eliminierung des Risikos eines schnell wachsenden AAA sind. Die aktuelle Studie demonstriert die Anwendung der miRNA-335-5p-Detektion durch einen Sensor mit interdigitalen Elektroden (IDE), um den Schweregrad von AAA bei betroffenen Personen zu bestimmen.
Materialien und Methoden
Reagenzien und Biomoleküle
Streptavidin, 1,1'-Carbonyldiimidazol (CDI) und Phosphatpufferlösung (PBS) wurden von Sigma-Aldrich, USA, bezogen. Ethanolamin wurde von Fisher Scientific, UK, bezogen. Alle Oligos wurden kommerziell von Apical Scientific Sdn. synthetisiert. Bhd., Malaysia.
Musterdesign auf einer Chrome-Maske
Ursprünglich wurde das Muster des dielektrischen Sensors mit der AutoCAD-Software entworfen. Die gewünschten Abmessungen waren eine Länge von 7500 µm, eine Breite von 4100 µm, 20 Fingerspaltpaare, eine Spaltgröße von 85 µm, Elektrodengröße von 100 µm, Elektrodendicke von 40 µm und eine Fingerlänge von 4000 µm. Das Muster wurde auf eine leere Fotomaske gedruckt und auf die Oberfläche des Chromglases geklebt. Dieses Chromglas wurde unter einem UV-Belichtungssystem für das Musterübertragungsverfahren fixiert. Ein mit einem dünnen Aluminiumfilm abgeschiedenes Siliziumdioxidsubstrat wurde in entgegengesetzter Richtung gegen das Chromglas gelegt und 10 s lang UV-Licht ausgesetzt. Das Muster wurde übertragen, gefolgt vom Entwicklungsprozess unter Verwendung des Resistentwicklers.
Herstellung der Interdigitalelektrode
Die Oberflächenelektrode wurde über einen herkömmlichen mikroelektronischen Herstellungsprozess zur Herstellung der Oberfläche wie folgt hergestellt:(i) Ein 4-Zoll-Wafer mit vorhandenem nativem Oxid wurde hergestellt. (ii) Der Wafer wurde unter Verwendung von gepufferter Oxidätzung (BOE) und Piranha-Lösung gereinigt, um das native Oxid und anorganische Verunreinigungen zu entfernen. (iii) Eine 2800 dicke Oxidschicht wurde über eine Stunde nasser thermischer Oxidation bei einer Temperatur von 1000 C gezüchtet. (iv) Der Wafer mit der Oxidschicht wurde zur besseren Handhabung viergeteilt. (v) Die Metallschicht wurde mit 1 als Haftschicht für die leitfähige Schicht abgeschieden. Die Aluminiumschicht (40 nm) wurde mit einer Länge von 3 cm und einem Durchmesser von 0,5 mm über ein physikalisches Abscheidungsverfahren auf Basis eines thermischen Verdampfers hergestellt. (vi) Eine Schicht aus positivem Photoresist war eine Schleuderbeschichtung auf dem Substrat, (vii) Der Wafer wurde 60 s lang bei 90°C weich gebacken. (viii) Der Wafer wurde mit einer Chrommaske ausgerichtet und 10 s lang UV-Licht ausgesetzt. (ix) Der Wafer wurde innerhalb von 15 s in RD6-Entwicklerlösung gespült, um den unbelichteten Bereich zu entfernen, was zu dem Interdigitalelektroden-(IDE)-Muster mit Oxid führte. (x) Der Wafer wurde 120 s lang bei 90°C hartgebacken. (xi) Der Wafer wurde unter Verwendung von Königswasser geätzt, um den freigelegten Bereich beider Schichten zu entfernen. (xii) Das Oberflächenmuster wurde erzeugt und der verbleibende Photoresist wurde unter Verwendung von Aceton weggewaschen. Dann wurde eine 3D-Nanoprofilometrie-Analyse (Hawk 3D-Profilometrie, USA) durchgeführt, um das scharfe Bild der Lücken zwischen den Elektroden zu beobachten. Strommessungen (A) wurden unter Verwendung von Keithley 6487 mit einem linearen Sweep von 0 bis 2 V in einem Schritt von 0,1 V (bei 20 s) durchgeführt.
Chemische Oberflächenfunktionalisierung:Tetravalente Streptavidin-Biotin-Strategie
Zur chemischen Oberflächenfunktionalisierung wurde die Kieselsäureoberfläche zunächst gründlich mit 1 µM Kaliumhydroxid (pH 9,0) gewaschen, um die Oberfläche zu aktivieren. Diese Oberfläche wurde durch CDI (0,5 µM) mit einer Inkubationszeit von 1 Stunde weiter modifiziert, um mit 100 µm Streptavidin (für 1 Stunde) zu reagieren, verdünnt aus dem Originalstamm in 10 µM phosphatgepufferter Kochsalzlösung (PBS; pH ~7,4). Nach diesem Schritt wurden die nicht umgesetzten Räume mit 1 µM Ethanolamin (für 1 Stunde) blockiert. Dann wurde das tetravalente Streptavidin mit 1 µM biotinylierter Sonde miRNA-335-5p (für 10 min) bei Raumtemperatur umgesetzt. Nach Abschluss jeder Modifikation wurde die Oberfläche gründlich mit PBS gewaschen, sofern nicht anders angegeben.
Entwerfen der Sonden- und Zielsequenzen von miRNA-335-5p
Die miRNA-335-5p in voller Länge (5′-UGUUUUGAGCGGGGGUCAAGAGCAAUAACGAAAAAUGUUUGUCAUAAACCGUUUUUCAUUAUUGCUCCUGACCUCCUCAUUUGCUAUAUUCA-3′) mit dem Zugangscode MIMAT0000765 wurde aus der Datenbank gesammelt. Die gewünschte Region für diese Studie als Ziel ist 5′-UCAAGAGCAAUAACGAAAAAUGU-3′. Um die Zielregion miRNA-335-5p einzufangen, wurde die Sonde als 5'-ACAUUUUUCGUUAUUGCUCUUG-3' entworfen. Am 3'-Ende wurde Biotin so markiert, dass es mit auf der Sensoroberfläche immobilisiertem Streptavidin interagiert. Die Sekundärstruktur der miRNA-335-5p voller Länge wurde von der Online-Software mfold vorhergesagt (http://unafold.rna.albany.edu/?q=mfold).
Sonden-Ziel-Interaktionen:miRNA-335-5p-Sequenzen
Nach Bindung der biotinylierten Sonde an die Streptavidin-Oberfläche, wie oben beschrieben, interagierte die Ziel-miRNA-335-5p-Sequenz (für 10&supmin;&sup7;) bei Raumtemperatur von niedrigen femtomolaren zu niedrigen nanomolaren Konzentrationen. Diese Konzentrationen reichten von 1 µm bis 1 µm mit 10-facher serieller Verdünnung in PBS. Die Anhaftungen wurden unabhängig von niedrigeren zu höheren Konzentrationen getestet, und nach der Duplexbildung wurde die Oberfläche mit dem 5-fachen Volumen an PBS gewaschen und es wurden Messungen vorgenommen.
I-V-Messungen
Die elektrischen Charakterisierungen wurden durch I-V-Messungen unter Verwendung eines Keithley 6487 Picoammeters mit einer Spannungs-/Stromversorgung durchgeführt. Die Messungen wurden bei den oben genannten Volt auf der blanken Oberfläche aufgezeichnet, gefolgt von einer chemischen Modifikation auf jeder Oberfläche. Ebenso wurde für die interaktive Analyse zwischen der Sonde und der Ziel-miRNA-335-5p bei jeder Konzentration die Messung nach dem Waschschritt mit 10 Reaktionsvolumina durchgeführt. Alle Modifikationen, Interaktionen und Messungen wurden unter nassen Bedingungen durchgeführt, sofern nicht anders angegeben.
Ergebnisse und Diskussion
Herstellung des IDE-Sensors und Oberflächenanalyse
In der aktuellen Studie wurde miRNA-335-5p als Werkzeug verwendet, um den Schweregrad des AAA durch interaktive Analyse auf einem Sensor mit interdigitalen Elektroden (IDE) aufzuklären (Abb. 1). Für die interaktive Analyse mit der Sonde und dem Target, die aus der gesamten Länge von miRNA-335-5p entworfen wurden, wurde eine IDE-Oberfläche entworfen und unter Verwendung konventioneller photolithographischer Techniken hergestellt. Es wurden schrittweise Oberflächenmodifikationen durchgeführt und mit einem Amperemeter gemessen (Abb. 2b, c). Die für die Herstellung von IDE entworfene Maskenausrichtung und die Gleichmäßigkeit wurden durch hochauflösende 3D-Nanoprofilometrie-Analyse bestätigt. Diese Beobachtung bestätigte, dass die Herstellung und der Abstand zwischen den einzelnen Fingern gut aufeinander abgestimmt waren (Abb. 2d). IDEs haben eine Elektrodengröße von ~100µm mit einem Abstand von ~85µm, und es entsteht ein großes interkaliertes Netzwerk mit Millionen von Biomarkern in Serie/Parallel. Um den Erfolg der hergestellten IDEs sicherzustellen, haben wir verschiedene nackte Geräte verwendet, um die Reproduzierbarkeit unter dem dielektrischen System zu bestimmen. Dieses System misst die durch das Dipolmoment verursachten molekularen Schwingungen zwischen den Dielektroden (Abb. 2c). Diese dielektrische IDE ist ein gut akzeptiertes System, das für verschiedene biomolekulare und chemische Analysen weit verbreitet ist [15, 25, 26, 27, 28]. In dieser Studie wurden alle chemischen Oberflächenmodifikationen und interaktiven Analysen im I-V-Strommodus gemessen.
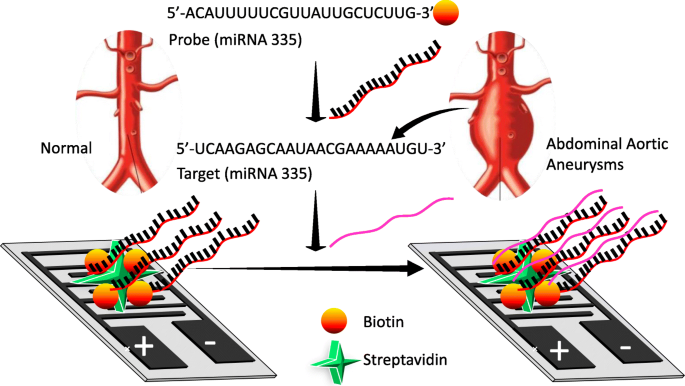
Darstellung des Nachweises von miRNA-335-5p an einer IDE. Die entworfene Sonde und das Ziel werden zusammen mit der Duplexbildung auf der IDE-Oberfläche gezeigt
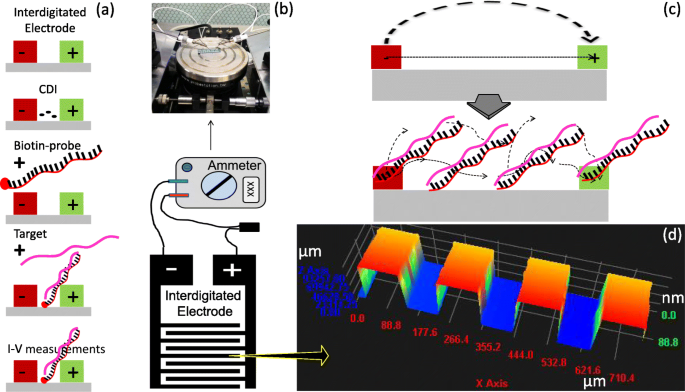
Oberflächenmessungen auf einer IDE. a Messschritte. b Messmodus. Das IDE-Muster und das Verbindungssystem werden angezeigt. Eine dielektrische Messung wird ebenfalls angezeigt. c Oberflächenmechanismus während der Messung. Es wird ein Dipolmoment gezeigt. d Das 3D-Profilometrieprofil der hergestellten IDE
Molekulare Baugruppe mit hoher Dichte:Streptavidin-Biotin-Strategie
Es hat sich gezeigt, dass molekulare Anlagerungen mit hoher Dichte auf der Sensoroberfläche oder Biochips entscheidend sind und stark von der Oberflächenfunktionalisierung abhängig sind [29, 30]. Diese Studie zielte darauf ab, die geeignete Oberfläche zu bestimmen, die für eine große Anzahl von molekularen Akkommodationen erforderlich ist, die für die Genexpression, Entdeckung und molekulare Analyse erforderlich sind. Für diese potentiellen Anwendungen erfordert die Erzeugung von analytischen Systemoberflächen die richtige Kopplung von Molekülen, um sie in geeigneten Formaten zu immobilisieren. Diese Materialoberflächen wurden untersucht, um den Mechanismus der Oberflächeninteraktion zu untersuchen, da sie billig sind und unterschiedliche Packungsdichten, Morphologie und Dicke bieten. Dieser Befund wird die richtige Anlagerung von Molekülen erleichtern und den Verlust der biomolekularen Aktivität verhindern. Darüber hinaus wird die richtige Ausrichtung der biomolekularen Immobilisierung mit der richtigen sekundären strukturellen Bestätigung beibehalten.
Um hochdichte Moleküle auf der Oberfläche zu erreichen, haben wir die Kieselsäureoberfläche zunächst gründlich mit Alkalilösung gewaschen und per CDI modifiziert. Um hohe Mengen an Biotinylierungssonden auf der CDI-Oberfläche einzufangen, haben wir die Sonde mit Streptavidin immobilisiert. Streptavidin ist ein tetravalentes Protein mit vier Bindungsstellen für Biotin und gilt in den Biowissenschaften als hochaffines Molekül [31, 32]. Die Oberfläche von IDE hatte ein aktuelles Niveau von 2.08E− 10 A, und nach der CDI-Anlagerung wurde es auf 9.2E− 06 A erhöht.Dieser Befund zeigt deutlich, dass die durch CDI modifizierte Oberfläche der IDE für unsere Analysen geeignet ist. Wenn Streptavidin hinzugefügt wurde, wurde das aktuelle Niveau weiter auf 2,36E− 05 A erhöht.Mit dieser Strategie immobilisierten wir eine größere Anzahl biotinylierterSonden, die aus miRNA-335-5p voller Länge entworfen wurden, auf der IDE-Oberfläche (Abb. 3a). Biotin-Wechselwirkungen wurden nach dem Blockierungsschritt auf der IDE-Oberfläche durchgeführt, um die nicht reagierten Bereiche zu maskieren. Bei jeder Oberflächenmodifikation wurden die Stromänderungen bestimmt (Abb. 3b). Wie in Abb. 3c gezeigt, betrugen die Stromänderungen nach Zugabe von Ethanolamin 2,53E− 05 A, und wenn 1 nM der biotinylierten Einfangsequenz zugegeben wurde, wurde die Stromstärke auf 1,29E− 08 A verringert der Strom bestätigte die Immobilisierung der Einfangsonde auf der IDE-Sensoroberfläche. Im Allgemeinen trägt ein einzelsträngiges Oligonukleotid eine negativere Ladung mit dem exponierten 5-Carbonat-Phosphat-Rückgrat (Sonde), während die Nettoladung von Ethanolamin neutral ist, wie vom Lieferanten angegeben. Dieser große Ladungsunterschied führte zu einer scheinbaren Änderung des gemessenen Stroms, wie in Fig. 3c gezeigt. Außerdem wurde dieser Parameter bei der Duplexbildung mit dem Zielstrang wieder rückgängig gemacht. Dieser Befund ist auf die reduzierte Exposition des Phosphatrückgrats zurückzuführen, und die zusätzlichen positiven Ladungen stammen von den Nukleobasen im miRNA-Strang (Abb. 4a).
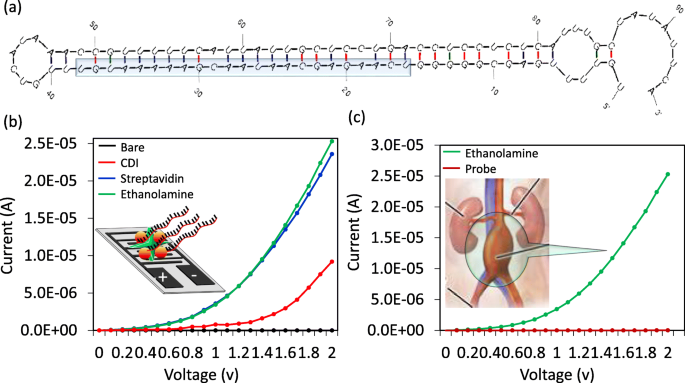
a Vorhergesagte Sekundärstruktur von miRNA-335-5p. Die miRNA-335-5p in voller Länge wird mit den Stamm- und Schleifenregionen angezeigt. Die ausgewählte Zielregion für die interaktive Analyse wird angezeigt. b I-V-Messungen am IDE für die oberflächenchemischen und biologischen Modifikationen (schwarzer Kreis, blank; roter Kreis, CDI; dunkelblauer Kreis, Streptavidin; grüner Kreis, Ethanolamin). Der Figureneinschub ist schematisch dargestellt. c I-V-Messungen an der IDE für den Sondenanschluss. (grüner Kreis, Ethanolamin; dunkelroter Kreis, Sonde). Das Bild für die AAA-Komplikation wird als Zahleneinschub gezeigt
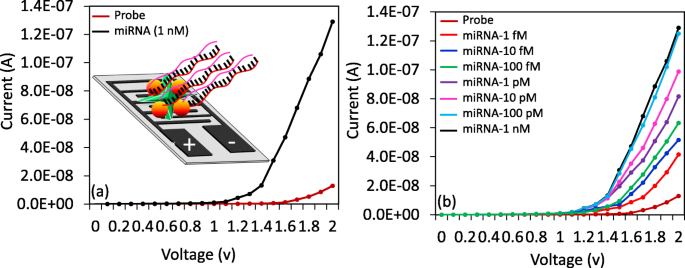
Interaktive Analyse von Sonde und Ziel. a Hochdosierte Interaktion der Sonde und miRNA-335-5p (Ziel). (dunkelroter Kreis, Sonde; schwarzer Kreis, Ziel). b Dosisabhängige Analyse von miRNA-335-5p. Prüfung vom unteren Femtomolaren bis zum unteren Nanomolaren Bereich. (dunkelroter Kreis, Sonde; oranger Kreis, 1 fM; blauer Kreis, 10 fM; grüner Kreis, 100 fM; lila Kreis, 1 pM; rosa Kreis, 10 pM; hellblauer Kreis, 100 pM; schwarzer Kreis, 1 nM)
Dosisabhängige interaktive Analyse auf der IDE
Nach Anbringen der biotinylierten Sonde an der Sensoroberfläche wurde die zielinteraktive Analyse mit unterschiedlichen Dosen durchgeführt. Diese Validierung wurde vom niedrigen Femtomolaren bis zum niedrigen Nanomolaren Bereich durchgeführt. Um diesen Bereich sicherzustellen, führten wir die vorläufige Analyse mit 1 µm der Ziel-MiRNA-335-5p-Sequenzen durch. Nach Zugabe von 1 µm der Ziel-miRNA-335-5p-Sequenz auf der mit der Einfangsonde modifizierten Oberfläche wurde das aktuelle Niveau von 1,29 E− 08 A auf 1,29E− 07 A geändert (Abb. 4a); dieses Ergebnis zeigte, dass zwischen der Einfangsonde und der Zielsequenz eine Hybridisierung stattfand. Nach dieser Bestätigung, um die Nachweisgrenze zu bestimmen, titrierten wir die Zielsequenz von 1 fM auf 1 nM, die unabhängig auf die mit der Einfangsonde modifizierten Oberflächen tropfte. Die Stromveränderungen wurden vor und nach der Immobilisierung festgestellt. Die Stromunterschiede für die 1 fM-, 10 fM-, 100 fM-, 1 pM-, 10 pM-, 100 pM- und 1 nM-Hybridisierungen betrugen 2,87, 3,87, 5,04, 6,88, 8,59, 11,21 bzw. 11,61E – 08 A. Basierend auf den erhaltenen Ergebnissen wurden in allen getesteten Konzentrationen der Zielsequenz deutliche dosisabhängige Stromänderungen festgestellt (Abb. 4b).
Hohe analytische Leistung:Sensitivität, Spezifität und Reproduzierbarkeit
Basierend auf den obigen Ergebnissen mit konzentrationsabhängigen Inkrementen wurde eine lineare Regressionsanalyse durchgeführt. Die Sensitivität des Nachweises wurde mit der 3σ-Rechnung abgeschätzt und fiel auf 1 fM (Abb. 5a, b). Die Konzentration betrug weniger als 1 fM, was das Hintergrundsignal und niedrigere Werte anzeigt. Weiterhin wurde eine Spezifitätsanalyse durchgeführt, um die Zielsequenzen von den nicht komplementären und einfach und dreifach fehlgepaarten Sequenzen zu unterscheiden. Es war offensichtlich, dass die entworfene Sonde eine stärkere Reaktion mit den perfekten miRNA-335-5p-komplementären Sequenzen zeigte als die anderen Sequenzen ( 6a ). Eine einzelne fehlgepaarte Sequenz führte aufgrund der teilweisen Duplexbildung durch die einfach fehlgepaarte Sequenz auf der Sondensequenz zu einer höheren Stromantwort als die nicht komplementären und dreifach fehlgepaarten Sequenzen. Es wurden Reproduzierbarkeitsanalysen mit allen chemischen und biologischen Modifikationen auf der IDE-Oberfläche in dreifacher Ausfertigung durchgeführt und die Daten gemittelt. Die gemittelten Daten zeigen, dass es keine signifikanten Veränderungen gab, wenn die verschiedenen Experimente durchgeführt wurden (Abb. 6b). Um die Leistung der IDE zu unterstützen, wurden außerdem die Eigenschaften der derzeit verfügbaren Methoden verglichen (Tabelle 1).
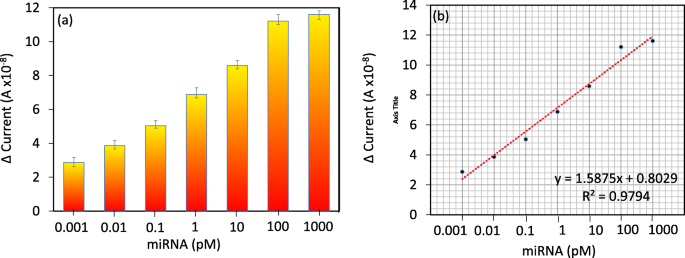
Dosisabhängigkeit. a Dosisabhängige Interaktion von miRNA-335-5p mit der Sonde. b Lineare Regressionsanalyse mit verschiedenen Konzentrationen von miRNA-335-5p. LOD wurde als die niedrigste Konzentration eines Analyten gegenüber dem Hintergrundsignal angesehen (S/N =3:1)
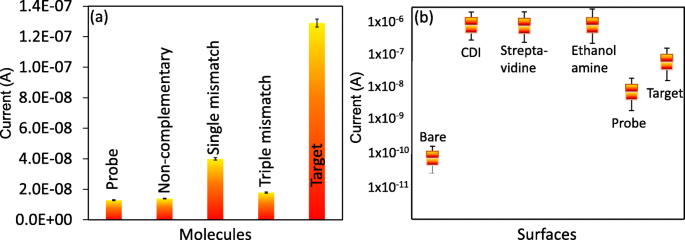
Hochleistungsanalyse. a Spezifitätsanalyse. Zielsequenzen wurden von den nicht komplementären und einfach und dreifach fehlgepaarten Sequenzen unterschieden. b Reproduzierbarkeitstest. Gemittelte Werte werden bei dreifachen Experimenten angezeigt
Schlussfolgerungen
Die Entwicklung einer analytischen Methode mit biologischen Biomarkern hilft bei der Diagnose von AAA bei körperlichen Untersuchungen. In der aktuellen Forschung wurde ein miRNA-335-5p-vermittelter Nachweis zur Vorhersage des Schweregrads von AAA entwickelt, da festgestellt wurde, dass miRNA-335-5p mit AAA exprimiert wird. Die Ausgabe der generierten IDE-Erkennung zeigte niedrigere bis höhere Erkennungsstufen an, um die Vorhersage des Schweregrads von AAA zu erleichtern. Die untere Nachweisgrenze lag bei 1 fM mit hoher Spezifität und Reproduzierbarkeit für die Hochleistungsanalyse. Die in dieser Forschung verwendete tetravalente Streptavidin-Biotin-Methode lieferte gute Ergebnisse und kann für andere Biomarker-Analysen verwendet werden.
Verfügbarkeit von Daten und Materialien
Alle Daten sind uneingeschränkt verfügbar.
Abkürzungen
- AAA:
-
Bauchaortenaneurysma
- CDI :
-
1,1′-Carbonyldiimidazol
- PBS:
-
Phosphatpufferlösung
- IDE:
-
Ineinandergreifende Elektrode
- UV:
-
Ultraviolett
Nanomaterialien
- N,N-Dimethylformamid, das die Fluoreszenz von MXen-Quantenpunkten für die empfindliche Bestimmung von Fe3+ reguliert
- Das Design der Emissionsschicht für Elektronenvervielfacher
- Der verbesserte photoelektrochemische Nachweis von Harnsäure auf Au-Nanopartikel-modifizierten Glaskohlenstoffelektroden
- Ein hochempfindlicher elektrochemischer DNA-Biosensor aus Acryl-Gold-Nanokomposit zur Bestimmung des Geschlechts von Arowana-Fischen
- Ultrasensitiver Biosensor zum Nachweis von Vibrio cholerae-DNA mit Polystyrol-Co-Acrylsäure-Komposit-Nanosphären
- Defekte auf der Oberfläche von Ti-dotiertem MgAl2O4-Nanophosphor
- Die Herstellung einer Au@TiO2-Dotter-Schale-Nanostruktur und ihre Anwendungen für den Abbau und den Nachweis von Methylenblau
- Was ist die beste SPI-Oberflächengüte für Ihr Spritzgussteil?
- Das richtige Elektrodenmaterial für Ihre Widerstandsschweißanwendung
- Die Lösung für einige häufige Probleme mit Elektrodenverbindungen



